| Мнемоника организма | AC в EMBL | Позиции в последовательности начало..конец |
| LACLM | AM406671 | 511423..512971 |
| LACAC | CP000033 | 59255..60826 |
| ENTFA | AE016830 | 248466..249987 |
| LISMO | CP002001 | 241024..242561 |
| BACSU | AL009126 | 635433..636987 |
| STAA1 | AP009324 | 531922..533476 |
| CLOTE | AE015927 | complement(8715..10223) |
| FINM2 | AP008971 | 197837..199361 |
Все последовательности рРНК, кроме последовательности LISMO, были найдены по описанию в поле FT в описании полного генома, который был найден по ссылке с белков IF2 соответствующих организмов. Полный геном LISMO, на который ссылался белок IF2_LISMO не содержал описаний рРНК. Я запустила blastn с последовательностью рРНК BACSU, указав нужный вид организма (Lysteria monocytogenes). Аналогичная последовательность была найдена в полном геноме LISMO в записи с AC CP002001. Были высокие показатели coverage, score, e-value. Поэтому я взяла этот фрагмент.
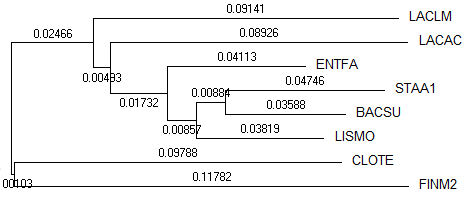
Для построения дерева я воспользовалась программой fneighbor, потому что это популярная и быстрая программа, которая строит достаточно правдоподобные деревья. Результат:

У этого дерева с правильным есть общие ветви: {BACSU, LISMO, STAA1} vs {...}, {CLOTE, FINM2} vs{...}. Это неплохой результат. Но качество реконструкции хуже, чем по белкам.
Я воспользовалась программой BLAST на сайте NCBI. Чтобы найти ортологи CLPX_BACSU я искала по белкам других бактерий и выбирала белки с мнемоникой CLPX. Чтобы найти паралоги, я искала похожие последовательности в одном организме, используя в качестве запроса последовательность CLPX соответствующего организма. И построила дерево, используя fneighbor.
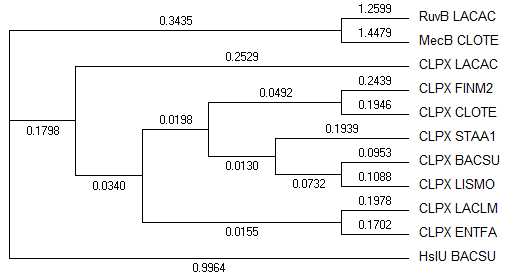
Ортологами, например, являются: CLPX_BACSU и CLPX_LISMO, CLPX_FINM2 и CLPX_LACLM, CLPX_CLOTE и CLPX_ENTFA. Паралогами являются: RUVB_LACAC и CLPX_LACAC, MECB_CLOTE и CLPX_CLOTE, HSIU_BACSU и CLPX_BACSU.