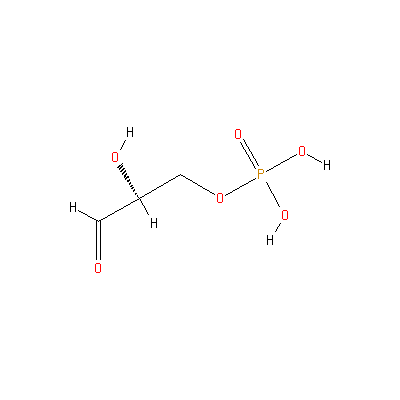
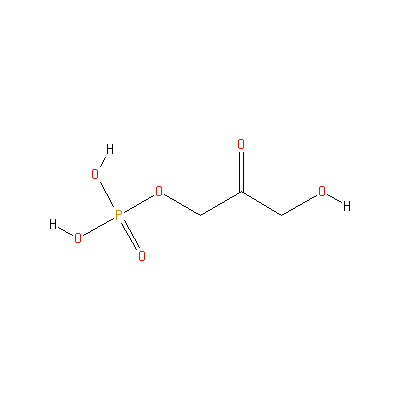
EC 5 — изомеразы (isomerases)
EC 5.3 — интрамолекулярные оксидоредуктазы (Intramolecular Oxidoreductases)
EC 5.3 — взаимное превращение Альдоз и Кетоз (Interconverting Aldoses and Ketoses)
EC 5.3.1.1 — триоз-фосфат изомераза (triose-phosphate isomerase)
IUPAC был организован в 1919
Международная комиссия по номенклатуре ферментов была организована в 1955
Последние изменения/уточнения в номенклатуре ферментов были утверждены в 2006
 |
= |  |
Общее число ферментов с таким кодом — 413
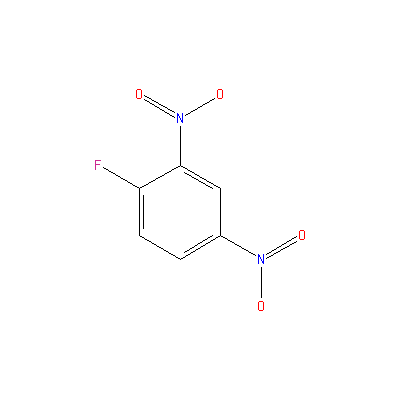
Примеры ингибиторов фермента:
| 2,4-Dinitrofluorobenzene | 5,5'-Dithiobis(2-nitrobenzoate) |
 |
.png) |
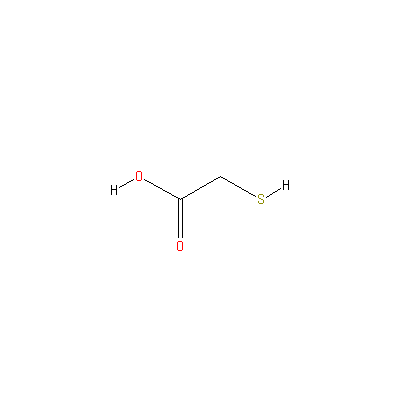
Примеры активаторов фермента:
| Sulfhydryl reagents | thioglycolate |
 |
 |
pH оптимум Min : 6,5-8,4 средн 7,5
pH оптимум Max : 7,5-11 средн 8.9
Болезни человека, так или иначе связанные с данным ферментом:
Анемия, Гемофилия, нехватка тирозин-фосфат изомеразы
Для Фермента более характерна структура димера с молекулярной массой около 2x27000 (найдено 12 ферментов).
Встречаются также мономеры (найдено 4 фермента) и тетрамеры (найдено 5 ферментов).
Найдена одна модификация
TPIS_ECOLI — PF00121 (4-246)
TPIS_HUMAN — PF00121 (6-244)
TPIS_METJA — PF00121 (1-212)
Как видно положение доменов в последовательностях практически совпадает.
Последовательности были получены с помощью команды seqret:
seqret sw:P0A858 -sask
seqret sw:P60174 -sask
seqret sw:Q58923 -sask
Попарная идентичность была определена с помощью программы needle
needle tpis_ecoli_domain.fasta tpis_human_domain.fasta -auto
needle tpis_ecoli_domain.fasta tpis_metja_domain.fasta -auto
needle tpis_human_domain.fasta tpis_metja_domain.fasta -auto
Выравнивания:
#=======================================
#
# Aligned_sequences: 2
# 1: TPIS_ECOLI
# 2: TPIS_HUMAN
# Matrix: EBLOSUM62
# Gap_penalty: 10.0
# Extend_penalty: 0.5
#
# Length: 245
# Identity: 111/245 (45.3%)
# Similarity: 145/245 (59.2%)
# Gaps: 8/245 ( 3.3%)
# Score: 481.0
#
#
#=======================================
TPIS_ECOLI 1 PLVMGNWKLNGSRHMVHELVSNLRKELAGV-AGCAVAIAPPEMYIDMAKR 49
..|.||||:||.:..:.||:..| ..|.| |...|..|||..|||.|::
TPIS_HUMAN 1 FFVGGNWKMNGRKQSLGELIGTL--NAAKVPADTEVVCAPPTAYIDFARQ 48
TPIS_ECOLI 50 EAEGSHIMLGAQNVDLNLSGAFTGETSAAMLKDIGAQYIIIGHSERRTYH 99
:.: ..|.:.|||.....:||||||.|..|:||.||.::::||||||...
TPIS_HUMAN 49 KLD-PKIAVAAQNCYKVTNGAFTGEISPGMIKDCGATWVVLGHSERRHVF 97
TPIS_ECOLI 100 KESDELIAKKFAVLKEQGLTPVLCIGETEAENEAGKTEEVCARQIDAVLK 149
.||||||.:|.|....:||..:.||||...|.|||.||:|...|...:
TPIS_HUMAN 98 GESDELIGQKVAHALAEGLGVIACIGEKLDEREAGITEKVVFEQTKVI-- 145
TPIS_ECOLI 150 TQGAAAFEGAVIAYEPVWAIGTGKSATPAQAQAVHKFIRDHI-AKVDANI 198
......:...|:||||||||||||:|||.|||.||:.:|..: :.|...:
TPIS_HUMAN 146 ADNVKDWSKVVLAYEPVWAIGTGKTATPQQAQEVHEKLRGWLKSNVSDAV 195
TPIS_ECOLI 199 AEQVIIQYGGSVNASNAAELFAQPDIDGALVGGASLKADAFAVIV 243
|:...|.|||||..:...||.:|||:||.||||||||.:...:|
TPIS_HUMAN 196 AQSTRIIYGGSVTGATCKELASQPDVDGFLVGGASLKPEFVDII- 239
#---------------------------------------
#---------------------------------------
#=======================================
#
# Aligned_sequences: 2
# 1: TPIS_ECOLI
# 2: TPIS_METJA
# Matrix: EBLOSUM62
# Gap_penalty: 10.0
# Extend_penalty: 0.5
#
# Length: 271
# Identity: 61/271 (22.5%)
# Similarity: 96/271 (35.4%)
# Gaps: 87/271 (32.1%)
# Score: 143.5
#
#
#=======================================
TPIS_ECOLI 1 PLVMGNWKLNGSRHMVHELVSNLRKELAGVA-------GCAVAIAPPEMY 43
.|::.|:| ..:|.:.|...|:|.:| |..:.:|| .:
TPIS_METJA 1 MLIVINYK------TYNESIGNRGLEIAKIAEKVSEESGITIGVAP--QF 42
TPIS_ECOLI 44 IDMAKREAEGSHIMLGAQNVDLNLSGAFTGETSAAMLKDIGAQYIIIGHS 93
:|: :...|..:|.:.||::|....|:.||...|..:||.|.:..:|.||
TPIS_METJA 43 VDL-RMIVENVNIPVYAQHIDNINPGSHTGHILAEAIKDCGCKGTLINHS 91
TPIS_ECOLI 94 ERRTYHKESDELIAKKFAVLKEQGLTPVLCIGETEAENEAGKTEEVCARQ 143
|:|....:.:.:|.| .|..||..: ||...
TPIS_METJA 92 EKRMLLADIEAVINK----CKNLGLETI-----------------VCTNN 120
TPIS_ECOLI 144 IDAVLKTQGAAAFEGAVIAYEPVWAIGTGKSATPAQAQAVHKFIR----- 188
|:. ::..||.....||.||...||||...:.|..:.|...:|
TPIS_METJA 121 INT---SKAVAALSPDYIAVEPPELIGTGIPVSKANPEVVEGTVRAVKEI 167
TPIS_ECOLI 189 ----------------DHIAKVDANIAEQVIIQYGGSVNASNAAELFAQP 222
|..|.:|.. ||.|::. .|.|.|.|..|..
TPIS_METJA 168 NKDVKVLCGAGISKGEDVKAALDLG-AEGVLLA-SGVVKAKNVEEAI--- 212
TPIS_ECOLI 223 DIDGALVGGASLKADAFAVIV 243
TPIS_METJA 213 --------------------- 212
#---------------------------------------
#---------------------------------------
#=======================================
#
# Aligned_sequences: 2
# 1: TPIS_HUMAN
# 2: TPIS_METJA
# Matrix: EBLOSUM62
# Gap_penalty: 10.0
# Extend_penalty: 0.5
#
# Length: 246
# Identity: 50/246 (20.3%)
# Similarity: 90/246 (36.6%)
# Gaps: 41/246 (16.7%)
# Score: 118.0
#
#
#=======================================
TPIS_HUMAN 1 FFVGGNWKMNGRKQSLGELIGTLN------AAKVPADTEVVCAPPTAYID 44
..:..|:| :..|.||... |.||..::.:.......::|
TPIS_METJA 1 MLIVINYK------TYNESIGNRGLEIAKIAEKVSEESGITIGVAPQFVD 44
TPIS_HUMAN 45 FARQKLDPKIAVAAQNCYKVTNGAFTGEISPGMIKDCGATWVVLGHSERR 94
......:..|.|.||:...:..|:.||.|....|||||....::.|||:|
TPIS_METJA 45 LRMIVENVNIPVYAQHIDNINPGSHTGHILAEAIKDCGCKGTLINHSEKR 94
TPIS_HUMAN 95 HVFGESDELIGQKVAHALAEGLGVIACIGEKLDEREAGITEKVVFEQTKV 144
.:..:.:.:|.: ....||..|.|... ...:|.
TPIS_METJA 95 MLLADIEAVINK----CKNLGLETIVCTNN--------------INTSKA 126
TPIS_HUMAN 145 IADNVKDWSKVVLAYEPVWAIGTGKTATPQQAQEVHEKLRGWLKSNVSDA 194
:|....|: :|.||...||||...:....:.|...:|...:.|
TPIS_METJA 127 VAALSPDY----IAVEPPELIGTGIPVSKANPEVVEGTVRAVKEIN---- 168
TPIS_HUMAN 195 VAQSTRIIYGGSVTGATCKELASQPDVDGFLVGGASLKPEFVDII- 239
:..:::.|..::.....:.|.....:|.|:....:|.:.|:..
TPIS_METJA 169 --KDVKVLCGAGISKGEDVKAALDLGAEGVLLASGVVKAKNVEEAI 212
#---------------------------------------
#---------------------------------------
Идентичность последовательностей не большая, однако домены находятся на тех же местах в последовательности, что позволяет с делать вывод об их сходной функции, и также большей консервативности трехмерной структуры.
В ENZYME можно найти название фермента, реакцию, которую он катализирует, ссылки на базы данных.
На главную страницу четвертого семестра