Выравнивание геномов: DotPlot
Поиск геномов
На странице NCBI Genomes я решила найти геном понравившейся мне бактерии (которая была упомянута в нескольких предыдущих практикумах) - Cupriavidus cauae. Запрос построила по видовому названию и в фильтрах выбрала сборку "хромосомы и полный геном".
Для сравнения выбрала геном другого организма из того же рода - Cupriavidus necator. Искала тем же методом, что и бактерию выше.
Получение картинок
Я загрузила эти геномы в BLAST и запустила сначала megablast (для быстрого поиска глобальных очень похожих участков последовательностей), а потом blastn (для более точной оценки схожести). Все с дефолтными параметрами.
В случае blastn покрытие генома составило 62%, а схожесть - 85.91%. В случае megablast: покрытие 50%, идентичность 92.17%.
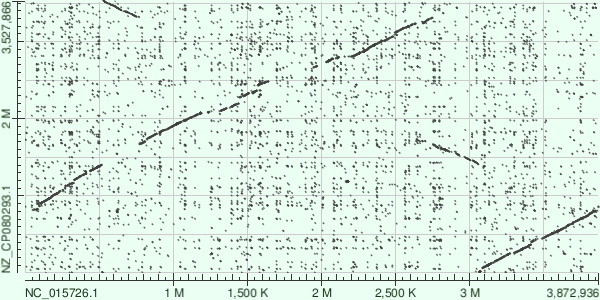
Рис1. DotPlot по результатам blastn.
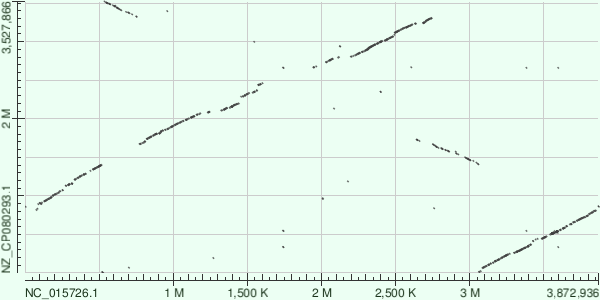
Рис2. DotPlot по результатам megablast.
Анализ DotPlot
На картинке выдачи blastn можно увидеть оченть много "шума" (точек), которые показывают практически единичные совпадения в двух последовательностях. Выдача megablast в этом плане чище, но при этом длинные участки сходства представлены менее четко (с большим количеством разрывов).
Проанализирую более подробно DotPlot, построенный megablast-ом.
По оси ОУ расположен геном бактерии Cupriavidus cauae (NZ_CP080293.1), а по оси ОХ - Cupriavidus necator (NC_015726.1).
Можно увидеть, что на графике есть длинные участки сходства, расположенные на главной диагонали, что говорит об одинаково выбранных цепях в банке геномов.
Рис3 - A: На участке 500К-700К вероятнее всего произошла инверсия с транспозицией.
Рис3 - B: Участок 1200К-1600К не очень хорошо вырoвнялся, но можно предположить что произошли вствки, или несинонимичные замены, или делеции, или просто есть не особо консервативные участки.
Рис3 - C: На участке 1600К-1950К либо произошла делеция (что объяснило бы несовпадение в размерах выравнивания на 350К нуклеотидов), либо это неконсервативный участок.
Рис3 - D: На участке 2600К-2650К вероятнее всего произошла инверсия с транспозицией.
Рис3 - E: На участке 2750К-3050К вероятнее всего произошла инверсия с транспозицией (либо это произошло между участками D и Е, что привело к возвращению на главную диагональ).
Рис3 - F: Участок 3050К-"конец графика" лежит на главной диагонали и показывает очень хорошее выравнивание. На самом деле он является примером того, что геномы бактерий порезаны по разному (выбрана разная точка старта), то есть в идеале с этого участка должна начинаться диагональ слева.
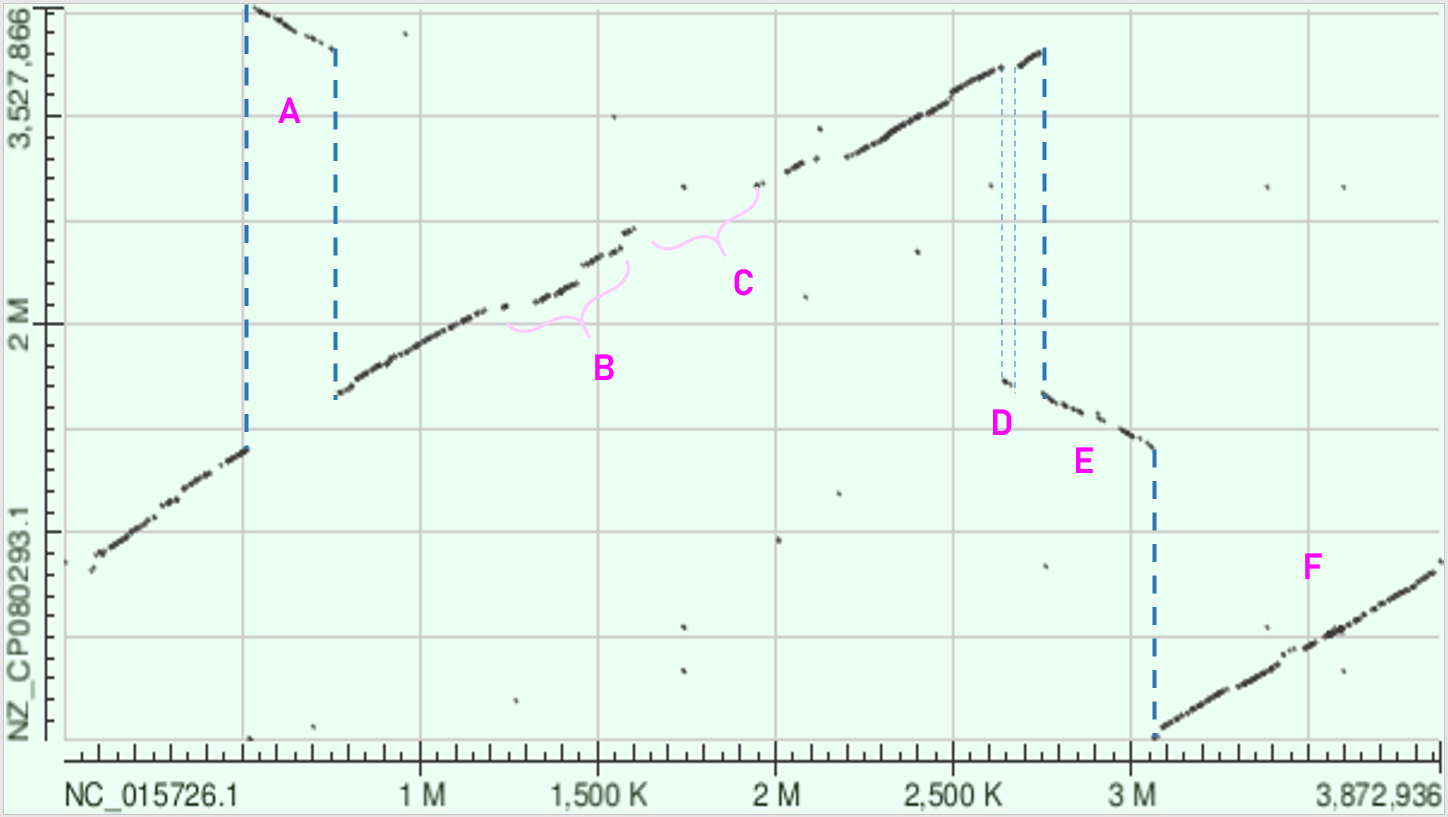
Рис3. DotPlot, анализ.