Мотивы и их белковые друзья
Поиск консервативных мотивов в выравнивании
Из предложенной в подсказках таблички доменов Pfam я выбрала PF00225: кинезиновый моторный домен (этот белок занимается переносом различных крупных частиц в клетке (к примеру, хромосом, лизосом с веществами), двигаясь по микротрубочкам с затратой АТФ (он очень смешно-мило ходит))
В выравнивании seed содержалось 69 последовательностей. Никакие строчки я из него не удаляла, так как они все достаточно различаются между собой В Jalview поставила окраску Clustal, Above identity threshold 100% и постепенно снижала порог, чтобы проявились мотивы. Нашлось достаточно много одиночных консервативных колонок и проявлялись некоторые мотивы.
Я выбрала самый консервативный мотив на позициях 441-445 в своем вырвнивании (почти нет вариации букв - толькое есть какие-то две выбивыющиеся последовательности, не выравнивающиеся с другими). Сам мотив: SSRSH - консервативный сайт связывания микротрубочек.

Рис1. Мотив SSRSH на позициях 441-445
Загрузила свой получившийся мотив в Scan Prosite. Нашлось 574 совпадений в 571 последовательностях. Выравнивание выглядит ужасно, потому что есть каки-то странные куски-последовательности, которые состоят только из мотива... (очень странно, не понимаю, почему так). Так же нашлись последовательности, которые не выровнялись на мотив (или выровнялись частично). Почему-то внезапно мотив вообще оказался разорван во многих последоваательностях (SSRS вместе, а Н через большой гэп).
Мотив, специфичный для одной клады филогенетического дерева
По изначальному выравниванию построила дерево в Jalview методом NJ. Выделила кладу с 6 представителями. Нашла у них довольно консервативный мотив на позиции 575-585: HIPYRDSKLRT. Паттерн: H[IV]PR[DN]S[KN]LT[RQ]. Такой длинный мотив не попадался больше нигде в выравнивании (хотя некоторые позиции консервативны для всех последовательностей).
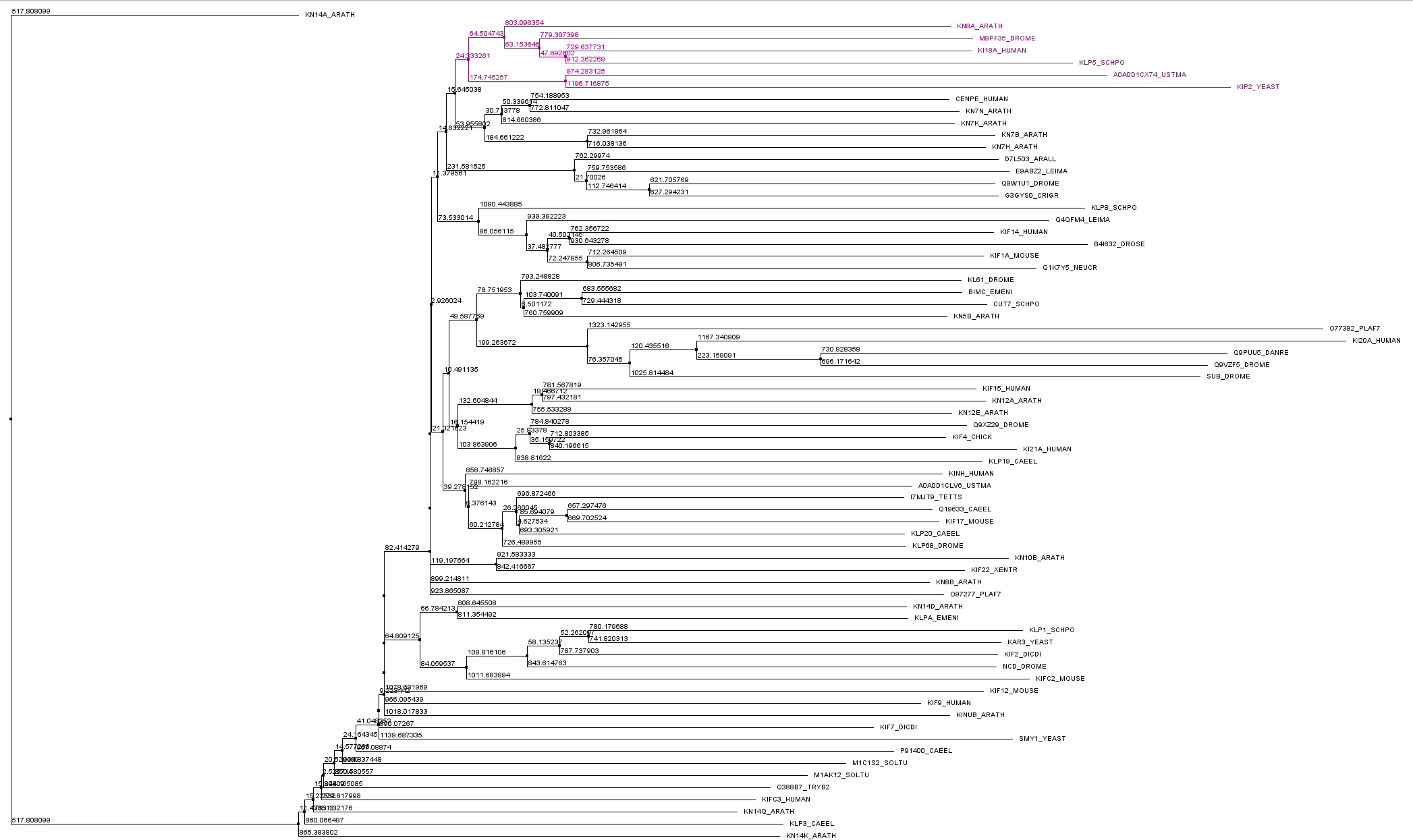
Рис2. Дерево по изначальному выравниванию seed

Рис3. Мотив HIPYRDSKLRT на позициях 575-585 в кладе
PSI-BLAST
Методом рандома я выбрала АС: P74518. Это фактор, способствующий переходу рибосом в спящий режим, выделенный из цианобактерии Synechocystis sp..
Я забила свой АС в PSI BLAST и получила такую выдачу:
| Номер итерации | Число находок выше порога (0,005) | Идентификатор худшей находки выше порога | E-value этой находки | Идентификатор лучшей находки ниже порога | E-value этой находки |
| 1 | 24 | P33987.1 | 0,00003 | - | - |
| 2 | 28 | P9WMA8.1 | 0,000004 | - | - |
| 3 | 28 | P24694.1 | 2E-20 | - | - |
| 4 | 28 | P24694.1 | 2E-20 | - | - |
С первой итерацией уже нет находок ниже порога, а с третьей количество перестает меняться, получается, мы действительно нащли обособленное семейство.
Поиск с помощью MEME
Я скачала выборку полных последовательностей (из нее ничего не удаляла), содержащих выбранный мной домен, отличную от выборки seed, через выбор таксона. Мне понравилось название Noctiluca scintillans (Sea sparkle) - это одноклеточная микроводоросль, которая живет в прибрежных зонах и теплых тропических водоемах, которая красиво люменисцирует.
Для анализа запустила следующую команду на kodomo:
meme match.fasta -o results -mod anr -minw 4 -maxw 8 -nmotifs 4
Нашлось 4 мотива, один из которых я изучала в первых заданиях (SSRSH) и еще 3 тоже консервативных.
Так же запустила FIMO для анализа:
fimo results/meme.txt match.fasta
Выдача программы: html
Эти 4 мотива нашлись 322 раза в 82 последовательностях, что звучит очень хорошо.
Представленность сайта GATC в геноме
Снова вернулась к своей любимой Cupriavidus cauae. Воспользовавшись кодом старшекурсников, я смогла построить график для obs/exp по методу Карлина для всех сайтов длины 4, полученных перестановками (без повторений) букв A, T, G, C.
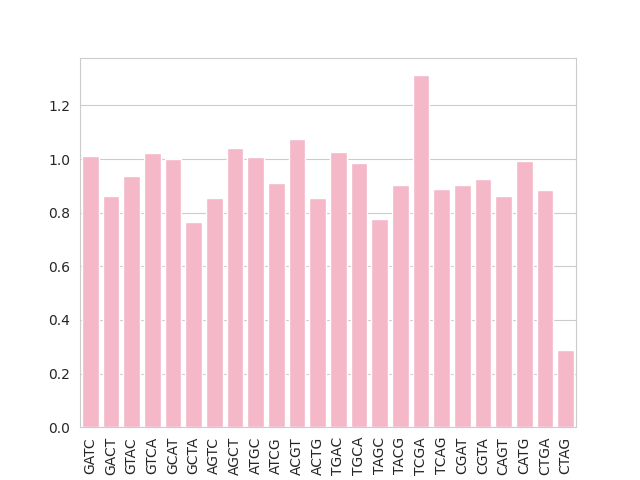
Рис4. График GATC
(Самый популярный сайт - TCGA, самый редкий - CTAG)