Поиск de novo сигналов в ДНК
(К сожалению, подсказок к практикуму не было, сделала как поняла задание)
Название бактерии: Cupriavidus cauae
NCBI RefSeq assembly: GCF_026210475.1
Файлы: FASTA с последовательностью генома (в файле 2 последовательности с двух "хромосом"), GFF с анотацией
Для того, чтобы найти найти основные сигналы, связанные с инициацией транскрипции и/или трансляции, я решила найти опероны своей бактерии программой Operon-mapper. Получился список оперонов. Это действие необходимо, чтобы взять оперон и пойти на 100 нуклеотидов до него (промотор), где лежат (закодированы) разные сигналы.
Очень удобно, что программа выдает описание оперонов, поэтому можно отобрать те, которые являются "генами домашнего хозяйства" (являются консервативными и необходимыми). Я воспользовалась кодом старшекурсников (Муравьева Георгия) (он показался мне наиболее понятным). Этот код генирирует 3 выборки: обучающая (с генами домашнего хозяйства), тестовая и отрицательный контроль (случайные последовательности длины 100).
Результатом кода являются 3 файла: housekeeping.fasta - обучающая выборка (промоторы генов домашнего хозяйства, отобрали по ключевым словам в Function), promotors.fasta - тестовая выборка (промоторы оперонов), negative.fasta - отрицательный контроль (случаные последовательности длиной 100 нуклеотидов из генома)
Поиск с помощью MEME
Для анализа запустила следующую команду на kodomo:
meme housekeeping.fasta -dna -nmotifs 3 -minw 6 -maxw 50
Параметры: -dna = показываю, что на вход дается последовательность ДНК, -nmotifs 3 = прошу найти 3 мотива, -minw 6 -maxw 50 = длина мотива должна быть от 6 до 5
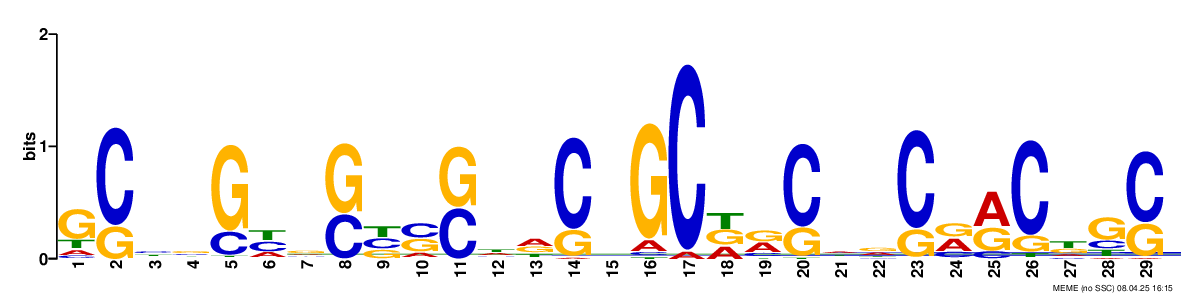
Рис1. Logo Мотив 1: GCHBGHRSBSSWDCNGCDRCHRCRRCKGC, width=29, E-value = 2.0e-030

Рис2. Logo Мотив 2: ARCNAWMWWWAHYBNAAWBDDHMWNKMBTHNWWDWHNTHHD, width=41, E-value = 4.3e-030
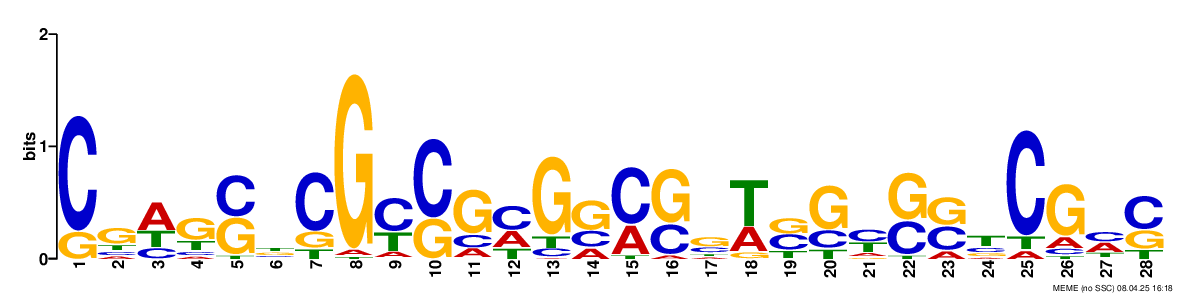
Рис3. Logo Мотив 3: CGWGSBCGYSGMGSMSSWBGYSSTCGHS, width=28, E-value = 9.1e-002
Для дальнейшего анализа я выбрала 1 мотив, у него наименьший E-value, хотя он выглядит больше как GC богатый участок (Upd: при анализе с помощью FIMO на негативном контроле будет хорошо видно, что выбор был не самый удачный)
Поиск с помощью FIMO
Я проверила первый мотив, полученный МЕМЕ, на тестовой сборке (promotors.fasta) и на отрицательном контроле (negative.fasta):
fimo --norc -motif GCHBGHRSBSSWDCNGCDRCHRCRRCKGC -thresh 0.001 meme.txt promotors.fasta
fimo --norc -motif GCHBGHRSBSSWDCNGCDRCHRCRRCKGC -thresh 0.001 meme.txt negative.fasta
Параметры: --norc = поиск по одной цепи, -motif = мотив, -thresh 0.001 = порог на p-value
Мотив нашелся в 20195 последовательностях теста и в 2329 последовательностях отрицательного контроля. Я бы сказала, что для отрицательного контроля это многовато (думаю так получилось из-за обилия GC в мотиве). Так же я провела анализ для разных порогов (-thresh 0.001 / 0.0001 / 0.005 / 0.05), но приводить результаты не буду, только скажу, что число находок сильно менялось, но для 0.001 оптимальнный результат, при котором не слишком много и не слишком мало находок (минимизировала совсем плохие вхождения)