Построение простейших филогенетических деревьев
Отобранные бактерии
| Название | Мнемоника |
| Bacillus anthracis | BACAN |
| Enterococcus faecalis | ENTFA |
| Finegoldia magna | FINM2 |
| Geobacillus kaustophilus | GEOKA |
| Lactobacillus delbrueckii | LACDA |
| Staphylococcus aureus | STAA1 |
| Streptococcus pyogenes | STRP1 |
Скобочная формула дерева
(FINM2,(((ENTFA,STRP1),LACDA),((BACAN,GEOKA),STAA1)));
Изображение дерева
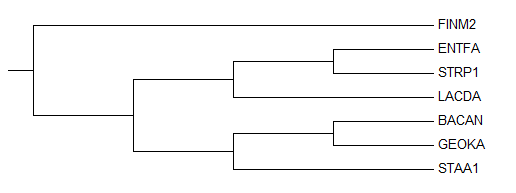
Ветви дерева
Дерево содержит 4 нетривиальные ветви:
{ENTFA,STRP1}против{FINM2,LACDA,BACAN,GEOKA,STAA1}
{ENTFA,STRP1,LACDA}против{FINM2,BACAN,GEOKA,STAA1}
{BACAN,GEOKA}против{FINM2,ENTFA,STRP1,LACDA,STAA1}
{BACAN,GEOKA,STAA1}против{FINM2,ENTFA,STRP1,LACDA}
Занятие1
Таксономия
BACAN;Bacillus anthracis: root; cellular organisms; Bacteria; Firmicutes; Bacilli; Bacillales; Bacillaceae
ENTFA;Enterococcus faecalis:root; cellular organisms; Bacteria; Firmicutes; Bacilli; Lactobacillales
FINM2;Finegoldia magna:root; cellular organisms; Bacteria; Firmicutes; Clostridia; Clostridiales
GEOKA;Geobacillus kaustophilus: root; cellular organisms; Bacteria; Firmicutes; Bacilli; Bacillales;Bacillaceae
LACDA;Lactobacillus delbrueckii: root; cellular organisms; Bacteria; Firmicutes; Bacilli; Lactobacillales
STAA1;Staphylococcus aureus:root; cellular organisms; Bacteria; Firmicutes; Bacilli; Bacillales; Staphylococcaceae
STRP1;Streptococcus pyogenes:root; cellular organisms; Bacteria; Firmicutes; Bacilli; Lactobacillales; Streptococcaceae
{ENTFA,STRP1,LACDA}-Lactobacillales
{BACAN,GEOKA,STAA1}-Bacillales
Анализ белков по выбранной функции
Из списка функций был выбран EFG, по нему реконструировалось филогенетическое дерево.
Были выбраны те же белки, по которым было построено исходное дерево:FINM2,ENTFA,STRP1,LACDA,BACAN,GEOKA,STAA1
По выравниванию программой muscle было построено одно дерево программой fprotraps
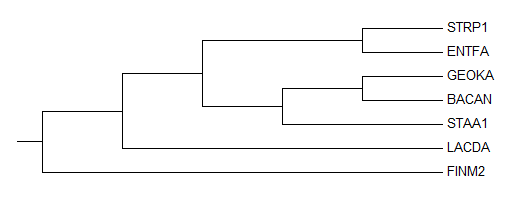
((((STRP1,ENTFA),((GEOKA,BACAN),STAA1)),LACDA),FINM2);
Сходные ветви
{BACAN,GEOKA}против{FINM2,ENTFA,STRP1,LACDA,STAA1}
{BACAN,GEOKA,STAA1}против{FINM2,ENTFA,STRP1,LACDA}
{ENTFA,STRP1}против{FINM2,LACDA,BACAN,GEOKA,STAA1}
Отсутствующие ветви ветви
{ENTFA,STRP1,LACDA}против{FINM2,LACDA,BACAN,GEOKA,STAA1}
Матрица расстояний
Была построена матрица рсстояний и по ней были построены деревья программой fneighbor
7
GEOKA 0.000000 0.439417 0.357603 0.260597 0.273624 0.262766 0.155204
FINM2 0.439417 0.000000 0.587763 0.509628 0.469923 0.501674 0.402205
LACDA 0.357603 0.587763 0.000000 0.384294 0.355202 0.375166 0.389869
STAA1 0.260597 0.509628 0.384294 0.000000 0.300771 0.291651 0.240469
ENTFA 0.273624 0.469923 0.355202 0.300771 0.000000 0.141123 0.241099
STRP1 0.262766 0.501674 0.375166 0.291651 0.141123 0.000000 0.229509
BACAN 0.155204 0.402205 0.389869 0.240469 0.241099 0.229509 0.000000
Ультраметричность
A-FINM2 B-LACDA C-ENTFA
d(A,B)=0.587763; d(A,C)=0.469923; d(B,C)= 0.355202
d(A,B)>d(A,C),но слишком сильно d(B,C) отстаёт от d(A,B).Ультраметричность не выполняется(отклонение~0.23)
A-GEOKA B-FINM2 C-ENTFA
d(A,B)=0,439417; d(A,C)=0,260597; d(B,C)=0,469923
Отклонение~0,03
Аддитивность
A-GEOKA B-ENTFA C-STPR1 D-BACAN
d(A,B)+d(C,D)~0.5
d(A,C)+d(B,D)~0.5
d(A,D)+d(B,C)~0.29
1 и 2 суммы равны и больше 3-аддитивность выполняется
Neighbor-joining и UPGMA
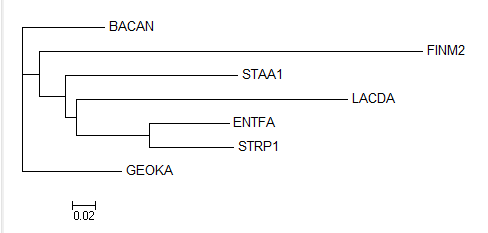
Neighbor-joining
(BACAN:0.07036,(FINM2:0.32827,(STAA1:0.14779,(LACDA:0.23261,
(ENTFA:0.06855,STRP1:0.07258):0.06202):0.00987):0.02189):0.01494,GEOKA:0.08485);
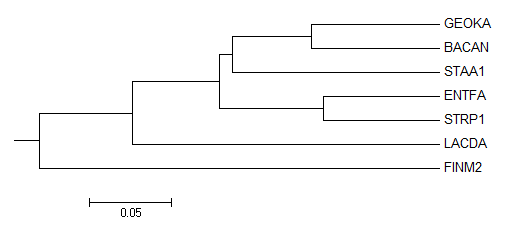
UPGMA
(((((GEOKA:0.07760,BACAN:0.07760):0.04766,STAA1:0.12527):0.00802,
(ENTFA:0.07056,STRP1:0.07056):0.06272):0.05293,LACDA:0.18621):0.05634,
FINM2:0.24255);
Отличия:
Отсутствие ветви {BACAN,GEOKA,STAA1}против{FINM2,ENTFA,STRP1,LACDA}
у первого дерева и {ENTFA,STRP1,LACDA}против{FINM2,BACAN,GEOKA,STAA1} у второго.