Занятие 10
Задание 1
Расшифровка обозначений в дереве:
-сначала цифра: 1- или 3- доменная архитектура.
-буква: E-эукаритоы; B-бактерии.
Были испробованы 2 алгоритма реконструкции дерева: fprotraps и fneighbor (UPGMA и Neighbor-joining).
В итоге был выбран fneighbor(neighbor-joining), т.к была получена наиболле правдоподобная струеура дерева.
Выравнивание отобранных последовательностей
Выравнивание отобранных последовательностей
Скобочная формула дерева
Дерево оказалось довольно удачным. Эукариоты и бактерии хоть и не оказались на отдельных кладах,
зато произошло разделение на клады по доменным архитектурам
Есть несколько исключений - один эукариотический 1-доменный белок (1_E_B3SAV7), забравшийся к 3-x доменным эукариотам,
а также один эукариотический 3-х доменный белок (3_E_A5BU55) появившийся в кладе с однодоменными.
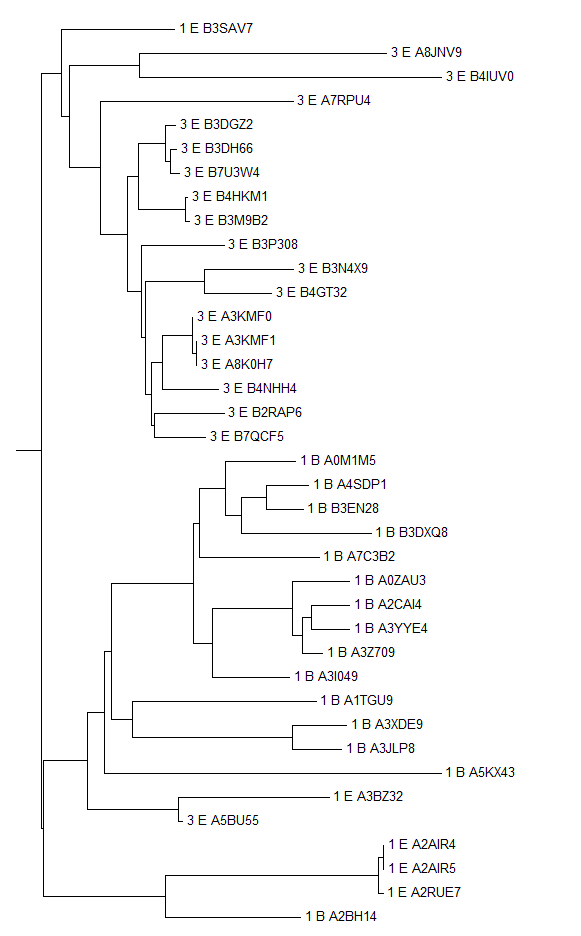
Можно предположить, что у предков была однодоменная архитектура, сохранившаяся у бактерий и части
однодоменных белков эукариот. Затем у эукариот из однодоменной образовалась и закрепилась полидоменная
архитектура, и какое-то время эволюционировала.
В данном случае логичнее разделить последовательности на основе таксономии
Построенные профили:
Для эукариотических
Для прокариотических белков
Графики зависимости ошибок 1 и 2 рода от порогового значения(1 рода-false negatives,2 рода-false positives)
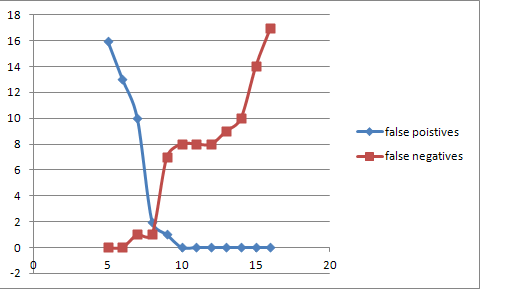
Зависимости для прокариот
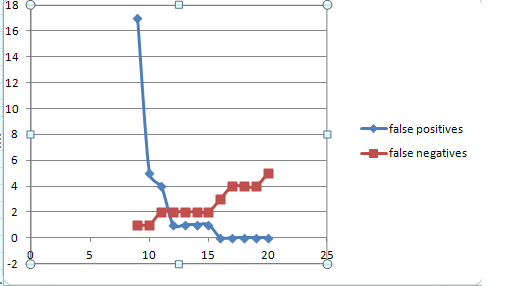
Зависимости для эукариот
При поиске по набору последовательностей для выделения бактерий наиболее оптимальным оказался порог 8.545
True positives = 19; true negatives = 16; false positives = 2; false negatives = 1,
Профиль для эукариот: при пороговом значении 10.6 результат был следующим
true positives = 17 ; true negatives = 15; false positives = 5; false negatives = 1.
Из такой разделимости можно сделать вывод о том, что белки из группы бактерий и из группы эукариот давно разделились,
а затем изменялись медленно, стали консервативны (оказались достаточно выгодными в эволюционном плане).
При поиске по эукариотическому профилю в банке swissprot было найдено 106 последовательностей
В случайно отобранных 25 последовательностей все содержали домен, покоторому вёлся поиск, но не все белки имели заданную
3-хдоменную архитектуру. Не было найдено ни одного из искомых белков.
Были найдены только эукариотические белки. Можно сделать вывод, что профиль можно с оговорками использовать для поиска заданных архитектур
Многие эукариотические белки содержали домены с теми же функциями , что и изначальные.
Профиль по однодоменным белкам бактерий работал лучше.
Было найдено 43 белка. Все из них есть в выдаче для предыдущего профиля. Есть смешение
однодоменных и многодоменных архитектур. Также не было найдено искомых белков, но есть совпадение архитектур
В результате можно сказать, что используя только один домен нельзя построить хороший профиль.