Понятие о выравнивании
Я решил,что 0 в данном случае-это отрицательное значение(N к K -0)
% идентичности=% сходства=66.66%(для данных seq1 и seq2)

Выравнивание seq1 и seq2
Процент идентичности(Identities),сходства(Positives) и число гэпов(Gaps) указаны в сводке данных над выравниванием
выравнивание куска последовательности seq1 из SPSA_BACSU с полной последовательностью SPSA_BACSU
lcl|41817 seq1
Length=20
Subject:SPSA_BACSU
Query:SPSA_BACSU(seq1)
Score = 43.9 bits (102), Expect = 3e-10, Method: Compositional matrix adjust.
Identities = 20/20 (100%), Positives = 20/20 (100%), Gaps = 0/20 (0%)
координаты искомого участка:55-74
Выравнивагие последовательностей белка SPSA_BACSU и WCAA_ECOLI
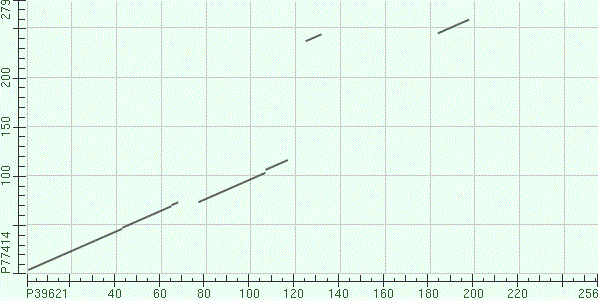
Карта локального сходства
Query:SPSA_BACSU(home organism:Bacillus subtilis)
Subject:WCAA_ECOLI(home organism:Escherichia coli (strain K12).
Score = 69.3 bits (168), Expect = 9e-17, Method: Compositional matrix adjust.
Identities = 44/121 (37%), Positives = 68/121 (57%), Gaps = 14/121 (11%)
координаты выравненого участков:2-117 и 5-116
Score = 15.8 bits (29), Expect = 1.0, Method: Compositional matrix adjust.
Identities = 5/14 (36%), Positives = 7/14 (50%), Gaps = 0/14 (0%)
координаты выравненого участков:185-198 и 246-259
Score = 13.5 bits (23), Expect = 4.9, Method: Compositional matrix adjust.
Identities = 5/7 (72%), Positives = 5/7 (72%), Gaps = 0/7 (0%)
координаты выравненого участков:126-132 и 238-244
Дополнительное задание1
 Выравнивание SPSA_BACSU и WCAA_ECOLI
Выравнивание SPSA_BACSU и WCAA_ECOLI


 Выравнивание SPSA_BACSU и WCAA_ECOLI
Выравнивание SPSA_BACSU и WCAA_ECOLI