A,B,Z-формы ДНК.
Параметры структур.| A-форма | B-форма | *Z-форма | |
| Тип спирали (правая или левая) | правая | правая | левая |
| Шаг спирали (A) | 26.04 | 34 | 37.13 |
| Число оснований на виток | 11 | 10 | 12 |
| Ширина большой бороздки | 7,99(от фосфата ДНК) | 20.31(от фосфата ДНК) | 9.85(от фосфата ДНК) |
| Ширина малой бороздки | 18.47(от фосфата ДНК | 11.21(от фосфата ДНК) | 18(от фосфата ДНК) |
Торсионные углы для A18
B:
?=-29.89
?=136.32
?=31.14
?=143.35
?=-140.76
?=-160.46
?=-97
A:
?=-51.68
?=174.5
?=41.6
?=79.09
?=173.24
?=-75.10
?=-156.7
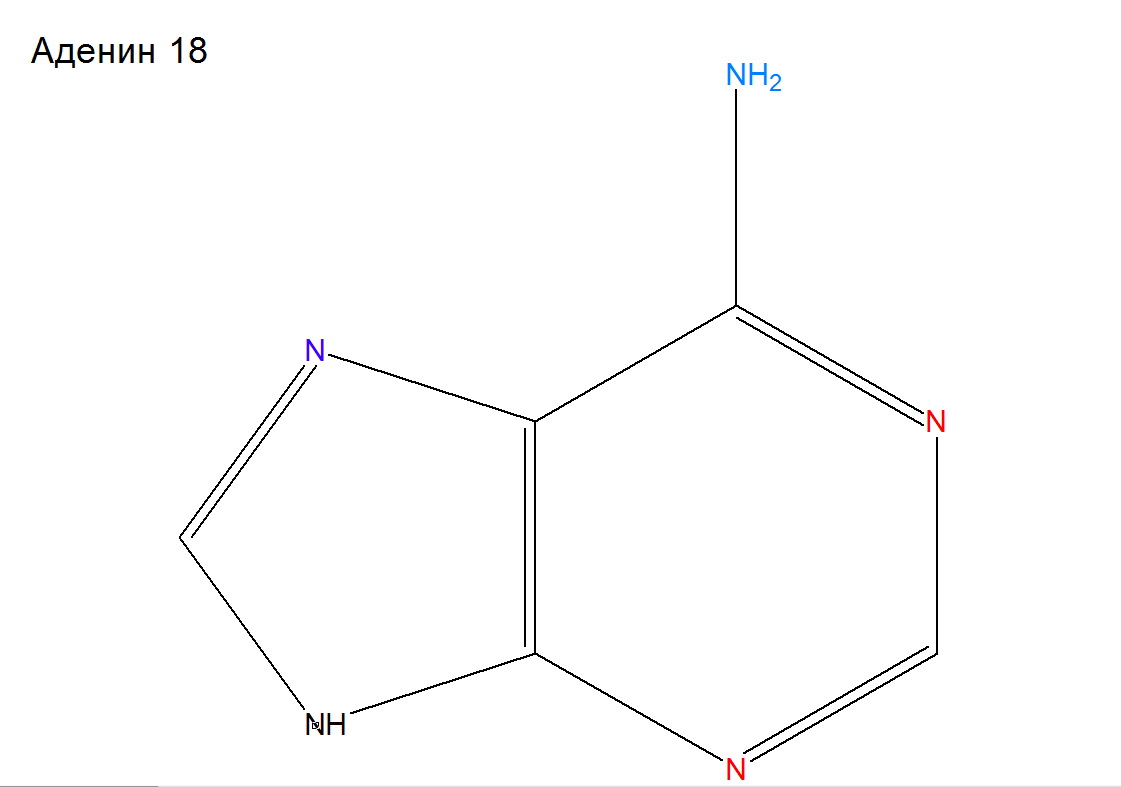
Аденин 18
В сторону большой бороздки-N1,C2,N3,C4.
В сторону малой бороздки-C8,N7,C5,N6,C6
Остальные атомы основания в плоскости спирали
Разные атомы A-формы ДНК
•сахарофосфатный остов ДНК
•все нуклеотиды
•все аденины
•атом N7 во всех гуанинах
•атом N7 в аденине 1 по последовательности(выделен жёлтым)
"Исследование структуры тРНК"
- Краткое описание структуры в файле XXXX.pdb В файле приведены координаты атомов следующих молекул
( Escherichia coli ,В структуре 2 молекулы:тРНК(GLUTAMINE TRNA APTAMER) и белок(GLUTAMINYL-TRNA SYNTHETASE).Каждая молекула представлена в единичном экземпляре..
Для исследования была выбрана цепь B, представляющая собой тРНК со следующей последовательностью
902_5':[..G]G-----C[..C]:3_971(посдедовательность в fasta формате:>1exd_B mol:nucleic length:73 Glutamine tRNA Aptamer 5'-GGGGUAUCGCCAAGCGGUAAGGCACCGGAUUCUGAUUCCGGAGGUCGAGGUUCGAAUCCUCGUACCCCAGCCA-3'
где 902 и 971 - номера первого и последнего нуклеотида.
В последовательности на 3'-конце ecnm триплет CCA.Приведены координаты его атомов. - Исследование вторичной структуры
С помощью программы find_pair пакета 3DNA были определены возможные водородные связи
между азотистыми основаниями (ссылка на выходной файл). В соответствии с полученными данными
акцепторный стебель состоит из участка 902-907 и комплементарного ему участка 966-971.
Т-стебель-из 949-953 и 961-965
D-стебель-из участков 910-912 и 923-925
антикодоновый стебель из 937-944 и 926-933
Структуру стеблевых дуплексов поддерживают 19 канонических и 9 неканонических пар оснований.Рис.1. Вторичная структура тРНК и 1EXD.pdb Скрипт для получения изображения restrict nucleic
pause
backbone 50
color white
pause
wireframe off
pause
select 902-907 or 966-971
color red
pause
select 949-953 or 961-965
color green
pause
select 938-944 or 926-932
color orange
pause
select 910-912 or 923-925
color blue
Пара Аденин-Аденин
Желательно отметить также
а)вариабельная петля отсутствует
а)Тимидин в т-петле отсутствует
а)дигидроуридинов в D-петле-U948
*Дополнительное задание
- Исследование третичной структуры
1. Программа analyze показала возможные стекинг взаимодействия между 2 парами оснований соединённых водородными связями.
Взаимодействия 27 пар оснований.Были выбраны взаимодействия между концом акцепторного стебля и начала T-стубля:
и между антикодоновым и D-стеблем.
Суммарная площадь перекрывания в первом случае составляет 9.51 для 1 случая,что довольно много и 1.56 для второго.
2. Если рассматривать водородные связи между основаниями D и T-петель,товыясниться,что существует 3
2 взаимодействия:955U-918G,915G-956C
- Предсказание вторичной структуры тРНК
Приведите результаты выполнения задания 9: таблицу, рисунок, полученный с помощью mfold,
не забудьте привести параметры использованных программ, а также краткие выводы. Желательно
описать процедуру подбора параметров.
Участок структуры
Позиции в структуре (по результатам find_pair)
Результаты предсказания
с помощью einvertedРезультаты предсказания по алгоритму Зукера
Акцепторный стебель
5' 902-907 3'
5' 966-971 3'
Всего 6 пар
предсказано 7 пар, из которых 6 реальныхпредсказано 6 пар
D-стебель
5' 910-912 3'
5' 923-925 3'
Всего 3 пары
Нет предсказаний
Предсказано 3 пары.
T-стебель
5' 949-953 3'
5' 961-965 3'
Всего 5 парНет предсказаний
Предсказаны все 5 пар
Антикодоновый стебель
5' 937 - 944 3'
5' 926-933 3'
Всего 7 парПредсказано 5 из 7 реальных
Предсказано 5 пар, включающих в себя 3 верных
Общее число канонических пар нуклеотидов
22
11
20
Выводы
Предсказание с помощбю EINVERTED
Для начала можно сказать,что программа почему-то подменяет урацилы на тимины.После многочисленных итераций программы
наиболее оптимальными оказались:gap penalty-1,minimum score threshold-20,match and mismatchscore-default.Отыскать T- и D-стебли не удалось.
В целом,программа неточная и очень требовательная к параметрам,что делает её нудобной в использовании.Предсказание с помощью MFOLD
Эта программа выдавала куда более правильные рузультатаы.Изменение сстандартных значений(P,в частности) приводило к увиличению количества структур,
тогда как число стеблей в каждой структуре не менялось.Я остановился на параметре P=5.Важно ещё отметить,что нумерация в последовательности и в pdb файле отличается:
в pdb файле отсутствуют некоторые остатки.
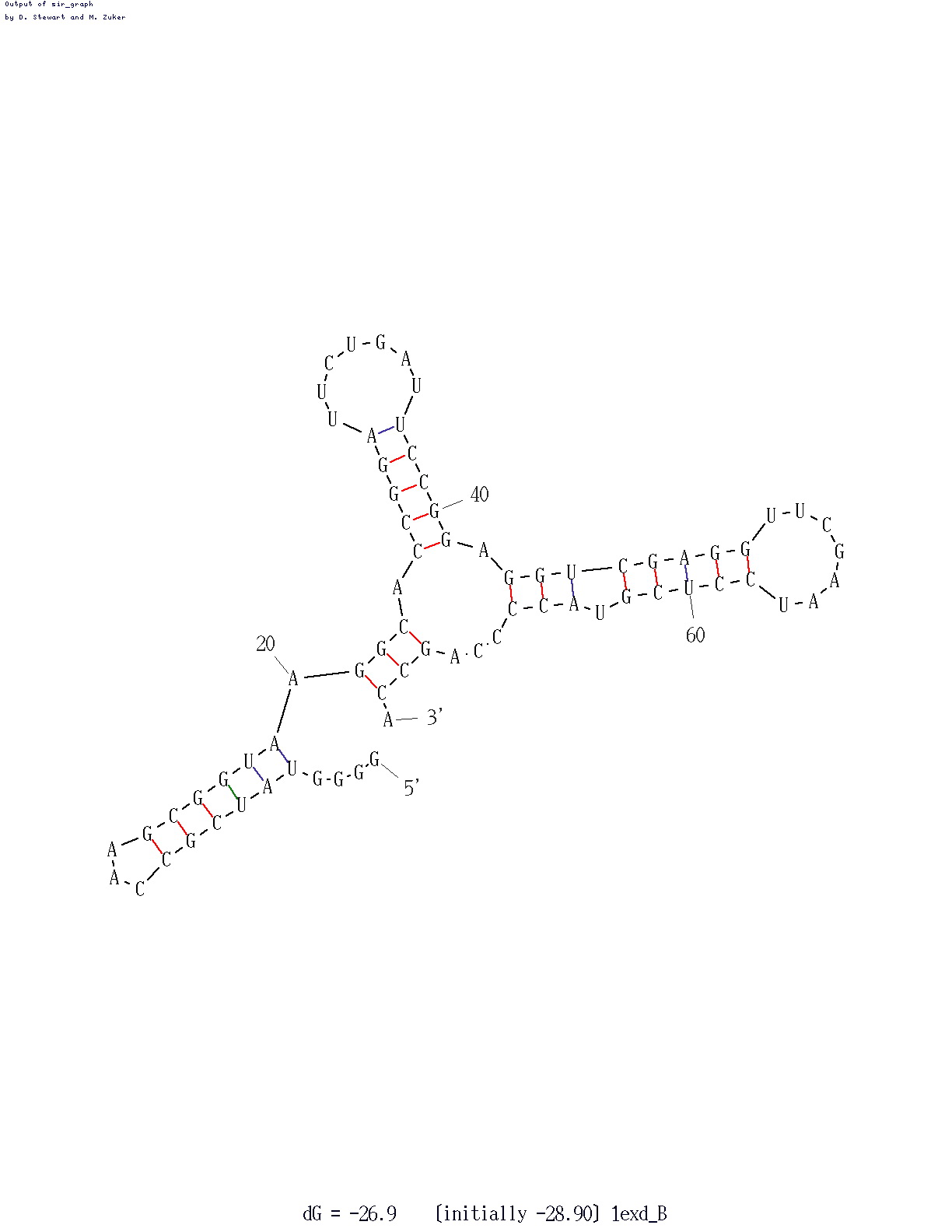
strucnure 2
Folding bases 1 to 73 of 1exd_B
Initial dG = -28.30
10 ----- .-UC| .-AA GG
GGGGUA GCC GC U
CCCCAU UGG CG A
ACCGA \ --^ \ -- GA
70 20
30
A UUC
CCGGA \
GGCCU U
A UAG
40
50
UUC
CGAGG \
GCUCC G
UAA
60