Я выбрала семейство каталитических субдоменов порфираназы 1. ID семейства - Porphyrn_cat_1, AC - PF18206. Этот домен обнаружен в белке порфираназе, присутствующем в Bacteroides plebeius. Порфираназа расщепляет порфиран во время переваривания гликанов красных водорослей. Стоит отметить, что гликаны красных водорослей содержат сульфатные эфиры, отсутствующие у наземных растений. Этот домен составляет часть каталитического домена белка порфираназы.
Общее число последовательностей – 199, а число последовательностей в выравнивании – 38, что соответствует ограничениям. Есть 41 архитектура с данным доменом, из которых я выбрала две: W7QFP9_9ALTE и A0A5C5ZD99_9BACT. Первая представлена 85 белками, а вторая – 23.
3D-структура известна для трех белков: A0A2D0TCD2_BACUN, A0A2D0TCD3_BACUN и PORA_BACPM. Белки встречаются только в домене Bacteria, поэтому я рассмотрела следующие по рангу таксоны. Среди Proteobacteria встречается 22 белка, среди Bacteroidetes – 56, среди Planctomycetes – 3, среди Firmicutes – 13, а также 105 белков относятся к группе Uncategorised bacterium.
HMM профиль выравнивания был создан 13 октября 2021 года и содержит 105 позиций.
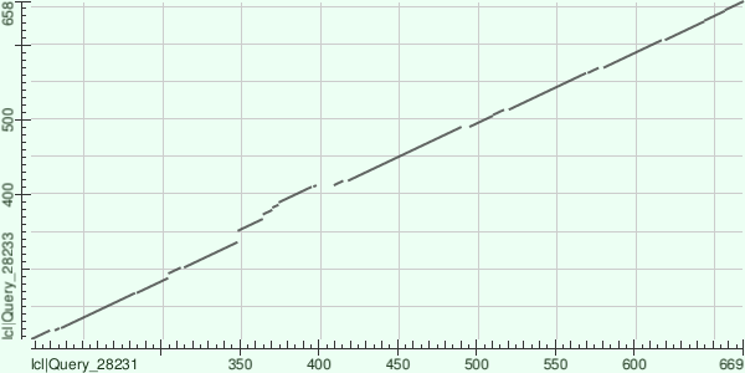
Я выбрала белки Beta-agarase и Beta-porphyranase A и построила карту локального сходства при двух значениях E-value: 0.05 (рис.1) и 50 (рис.2).

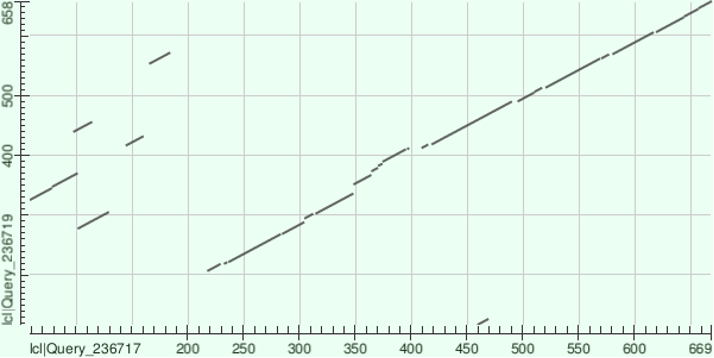
В первом случае выравнивание достаточно хорошее, есть участки с делецией или вставкой (350, 400, 415, 490). Во втором случае появилось несколько консервативных участков, но они небольшие, поэтому, скорее всего, это сходство обусловлено случайными совпадениями.
Я скачала выравнивание full, состоящее из 199 последовательностей. На основании филогенетического дерева я выделила 5 групп. Из них были выбраны для сравнения 2 самые большие группы.
Ссылка на полное выравнивание, выравнивание двух подгрупп.
Еще во второй группе есть консервативные столбцы, не характерные для первой (45, 57, 84).
Таблица доступна по ссылке.
Находок оказалось в 2 раза больше, чем последовательностей в full в Pfam. Из них белков из Swiss-Prot 0.5% , а белков с Protein evidence, не равной predicted, 3%. Также появились белки в других доменах. Все белки в Pfam принадлежали домену Bacteria, а в выдаче UniProt появилось 3 белка, относящихся к Eukaryota, и один белок, относящийся к Archaea.