Я скачала архив с чтениями с помощью команды:
wget ftp://ftp.sra.ebi.ac.uk/vol1/fastq/SRR424/001/SRR4240361/SRR4240361.fastq.gz
Далее я удалила остатки адаптеров с помощью программы trimmomatic, предварительно создав файл adapters.fasta, в котором все адаптеры из файлов директории /mnt/scratch/NGS/adapters:
java -jar /usr/share/java/trimmomatic.jar SE -phred33 SRR4240361.fastq SRR4240361_trim.fastq ILLUMINACLIP:adapters.fasta:2:7:7
Остатками адаптеров оказалось 0.47% чтений (34532 чтений).
Следующим шагом я удалила с правых концов чтений нуклеотиды с качеством ниже 20 и оставила только такие чтения, длина которых не меньше 32 нуклеотидов.
java -jar /usr/share/java/trimmomatic.jar SE -phred33 SRR4240361_trim.fastq SRR4240361_final.fastq TRAILING:20 MINLEN:32
В результате было удалено 403754 (5.58%) чтений.
Итого, исходно было 7272621 чтений, а после использования программы trimmomatic осталось 6834335. Изначальный размер файла с чтениями 193M, а конечный - 178M.
Подготовка k-меров:
velveth Assem 31 -short -fastq.gz SRR4240361_final.fastq.gz
Assem – директория, в которую помещаются k-меры, 31 – длина k-мера, short – указание на короткие и непарные чтения.
Сборка генома:
velvetg Assem/
В результате N50 = 25683. В таблице 1 приведены длины самых больших контигов и их покрытие:
| ID контига | Длина | Покрытие |
|---|---|---|
| 6 | 49238 | 26.66 |
| 2 | 45555 | 26.45 |
| 34 | 43866 | 23.51 |
Контиги с аномально большим и малым покрытием имеют маленькую длину (1). Это значит, что они отсутствуют в файде contigs.fa, так как длина k-мера 31. Из тех контигов, что попали в файл, низким покрытием обладает 391 контиг. Он имеет покрытие 2.24 и длину 63.
1. Контиг с длиной 49238 (ID 6):
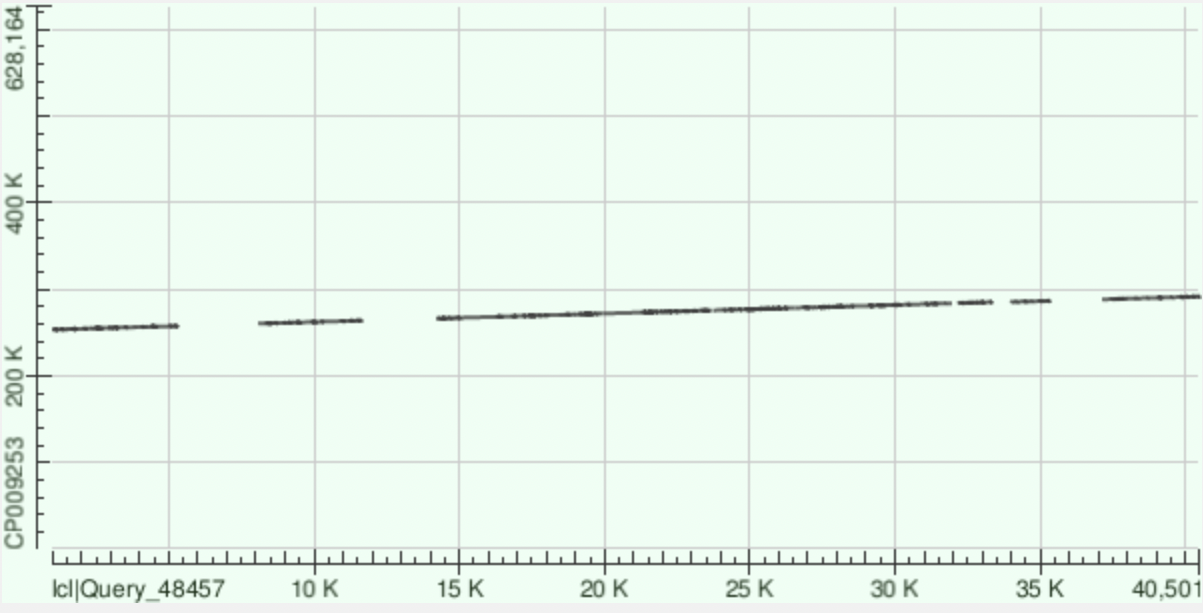
Картируется на следующие участки хромосомы 127825 – 140555, 144368 – 151796, 153752 – 161738, 161898 – 166752, 166750 – 173180. Идентичность на первом участке составляет 75%, на втором и третьем – 78%, на четвертом – 80% и на пятом – 76%. Процент гэпов меняется от 2 до 4% на разных участках. На рис. 1 представлен DotPlot для этого выравнивания.

2. Контиг с длиной 45555 (ID 2):
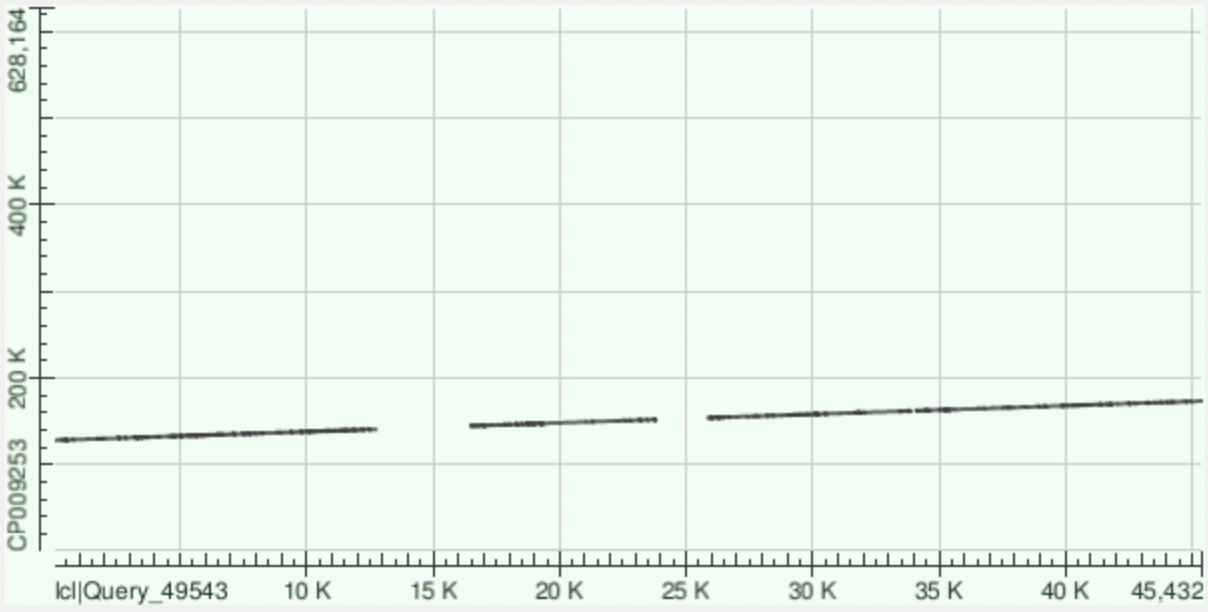
Картируется на следующие участки хромосомы: 440755 – 440944 (89%), 441135 – 442817 (79%), 442877 – 445895 (80%), 449411 – 454069 (75%), 462496 – 467421 (77%), 467412 – 474667 (77%), 474844 – 480660 (74%), 480874 - 481545 (82%) и 481997 – 485679 (77%). Идентичность для каждого из девяти участков указана в скобках. Процент гэпов меняется от 1 до 4%. На рис. 2 представлен DotPlot. Можно заметить, что прямая перевернута. Это говорит о том, что контиг картируется на геном в противоположном направлении.
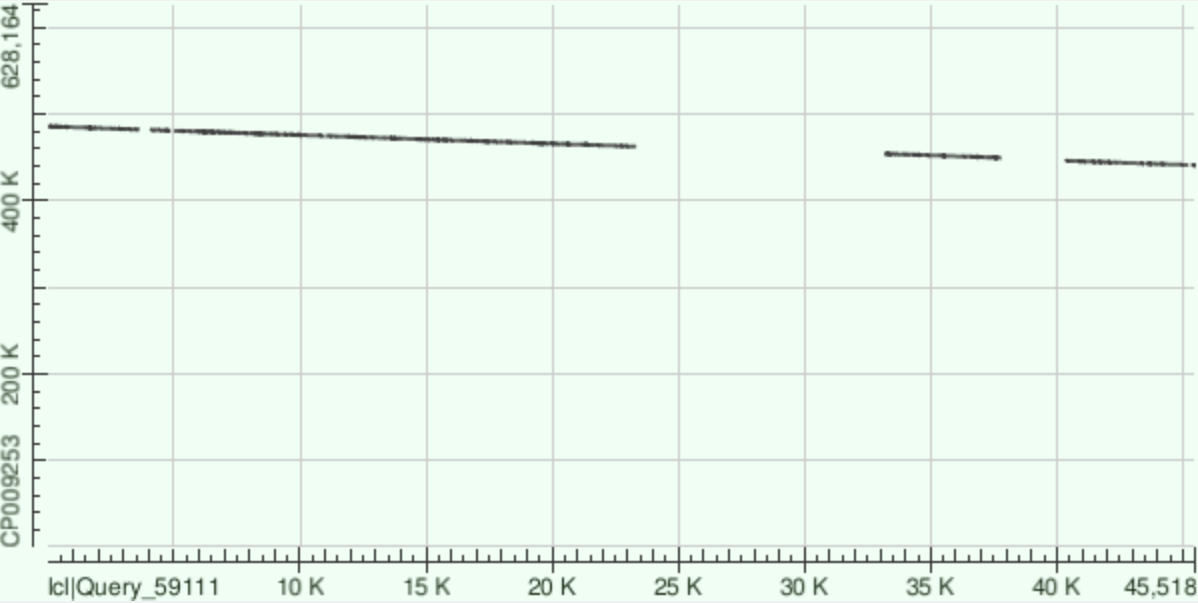
3. Контиг с длиной 43866 (ID 34):
Картируется на следующие участки хромосомы: 253223 – 257546 (73%), 260224 – 263784 (77%), 266073 – 275551 (79%), 275566 – 283706 (76%), 283963 – 285070 (76%), 285200 – 286535 (76%), 288181 – 291560 (78%). В скобках указана идентичность для каждого из семи участков. Процент гэпов меняется от 2 до 5%. На рис. 3 представлен DotPlot для данного выравнивания.