С помощью программы fiber пакета 3DNA я построила A-, B- и Z-форму ДНК.
Я использовала следующие команды:
fiber -a -seq=GATC -rep=5 gatc-a.pdb
fiber -b -seq=GATC -rep=5 gatc-b.pdb
fiber -z -seq=GC -rep=15 gc-z.pdb
Полученные файлы:
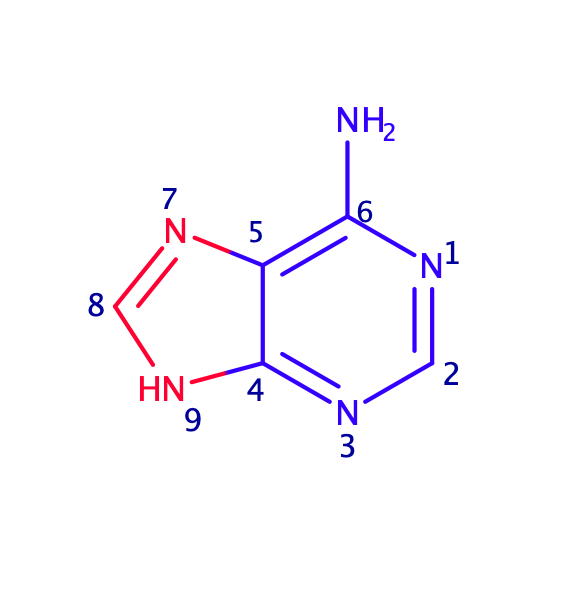
Я рассмотрела экспериментальную структуру B-формы ДНК (1bna) в Jmol. На рис.1 изображено заданное мне азотистое основание - аденин - в положении 17 B цепи ДНК.
Изображение было получено с помощью MarvinSketch. На нем красным цветом выделены атомы, смотрящие в сторону большой бороздки, синим - в сторону малой.
В сторону большой бороздки обращены атомы: A17.N7, A17.C8, A17.N9.
В сторону малой бороздки обращены атомы: A17.N1, A17.C2, A17.N3, A17.C4, A17.C5, A17.C6, A17.N6.

Далее я сравнила различные формы ДНК по структурам, полученным в задании 1. Данные приведены в Таблице 1.
| A-форма | B-форма | Z-форма | |
|---|---|---|---|
| Тип спирали (правая или левая) | правая | правая | левая |
| Шаг спирали (Å) | 28,16 | 33,75 | 43,5 |
| Число оснований на виток | 11 пар | 10 пар | 12 пар |
| Ширина большой бороздки | 16,97 [DA]10:A.P - [DA]34:B.P | 17,21 [DT]27:B.P - [DT]11:A.P | 18,3 [DC]48:B.P - [DC]10:A.P |
| Ширина малой бороздки | 7,98 [DC]24:B.P - [DT]11:A.P | 11,69 [DG]9:A.P - [DC]36:B.P | 8,68 [DG]49:B.P - [DG]17:A.P |
Я исследовала структуру заданной мне тРНК - 2cv1. Сначала файл был переведен в старый формат, чтобы можно было работать с пакетом 3DNA:
remediator --old ''2cv1.pdb'' > ''2cv1_old.pdb''
Затем с помощью программ find_pair и analyze были определены спаренные основания и положения спиралей в структуре.
find_pair -t 2cv1_old.pdb stdout | analyze
Все данные были получены из файла 2cv1_old.out
1) Координаты стеблей:
501-507 и 572-566
549-555 и 565-518
538-544 и 532-526
510-515 и 525-548
2) Неканонические пары оснований:
502G-571U
555U-518G
538A-532C
544A-526G
513U-522G
3) Дополнительная водородная связь, стабилизирующая структуру тРНК, образуется между 519G и 556C.