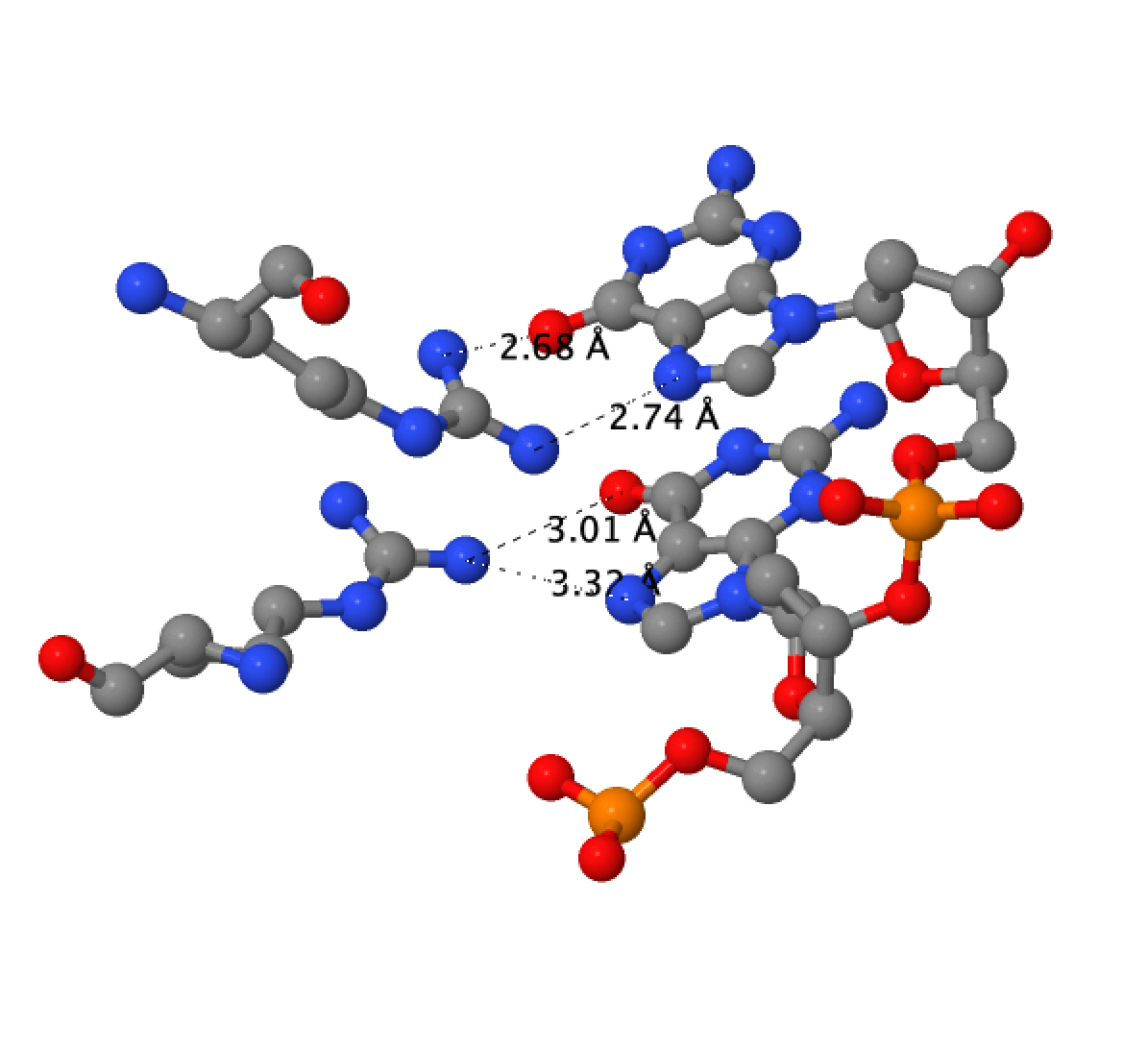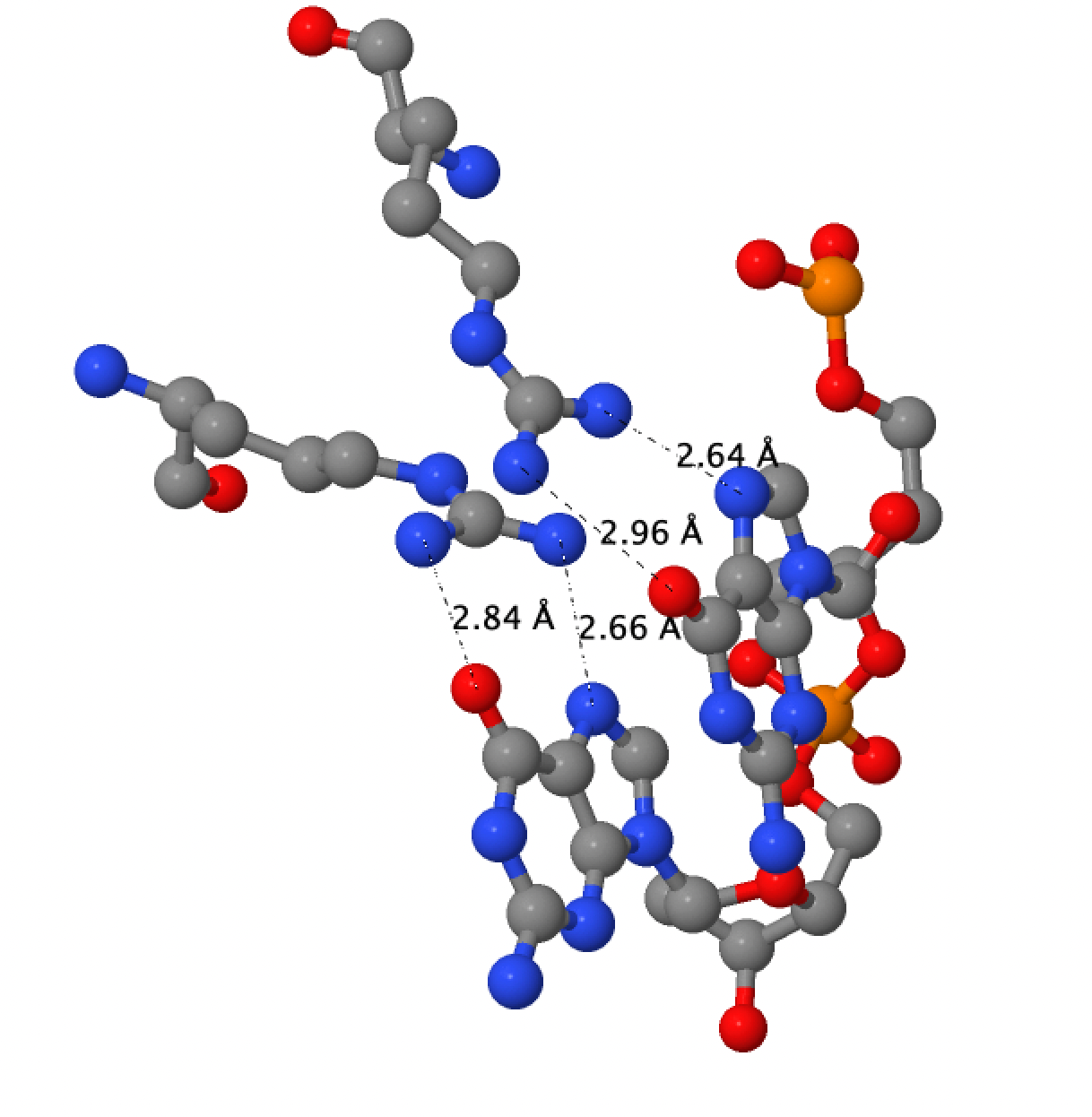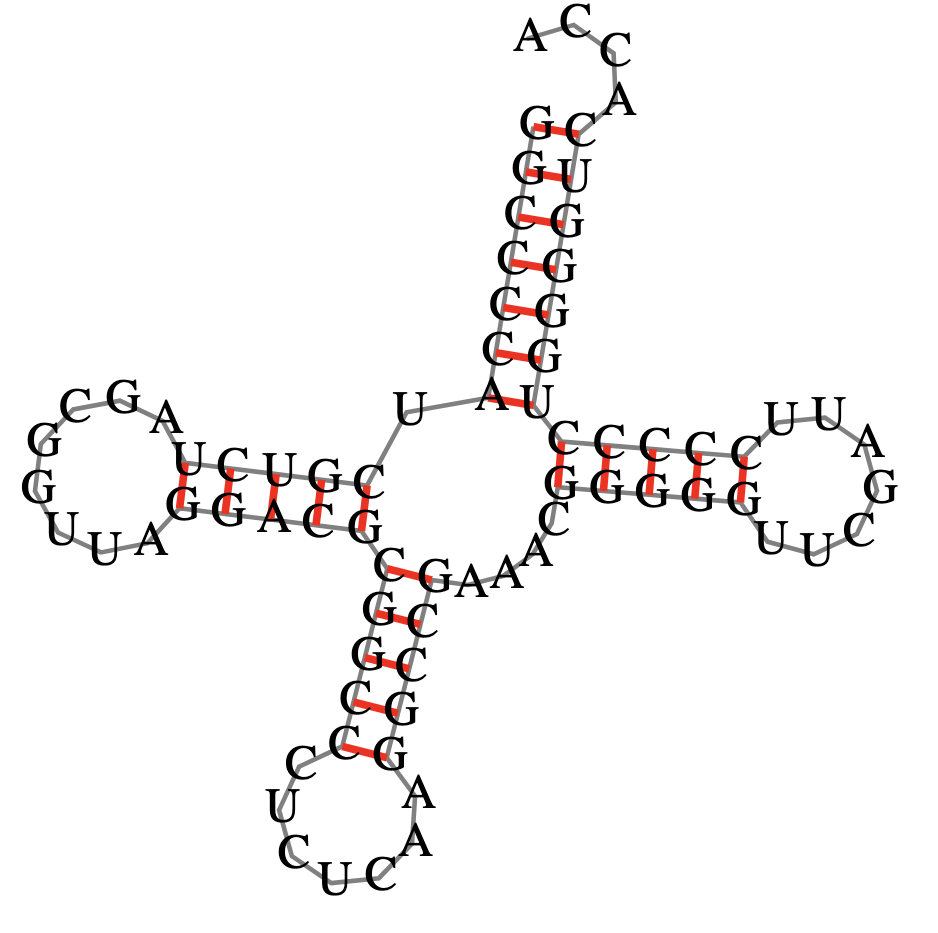
Я исследовала структуру тРНК 2cv1. Для предсказания вторичной структуры тРНК была использована программа einverted из пакета EMBOSS:
einverted -sequence 2cv1_rna -gap 12 -threshold 0 -match 3 -mismatch -4 -outfile outfile -outseq seqout
Полученные файлы
Далее для предсказания вторичной структуры был использован алгоритм Зукера с помощью ViennaRNA.
Предсказание показано на рис.1

На основе предсказания программ find_pair, einverted и ViennaRNA была построена таблица:
| Участок структуры | Позиции в структуре | Результаты предсказания с помощью einverted | Результаты предсказания по алгоритму Зукера |
|---|---|---|---|
| Акцепторный стебель | 1G - 7A и 72C- 66U | 3C - 7A и 69G - 65T | 1G - 7A и 71C - 65U |
| D-стебель | 10G - 13U и 25C - 22G | - | 10C - 13U и 25G - 22G |
| T-стебель | 49G - 53G и 65C - 61C | - | 48G - 52G и 64C - 60C |
| Антикодоновый стебель | 38A - 44A и 32C - 26C | - | 39G - 43G и 31C - 27C |
| Общее число канонических пар нуклеотидов | 23 | 5 | 21 |
Для этого задания я использовала структуру 1h9t.
С помощью команды define JMol были заданы следующие множества атомов:
- Множество атомов кислорода 2'-дезоксирибозы (set1)
- Множество атомов кислорода в остатке фосфорной кислоты (set2)
- Множество атомов азота в азотистых основаниях (set3)
Для этого был создан скрипт 1.
Далее я создала скрипт 2, вызов которого в JMol даст последовательное изображение всей структуры, только ДНК в проволочной модели, той же модели, но с выделенными шариками множеством атомов set1, затем set2 и set3.
Затем были построены ДНК-белковые контакты в заданной структуре и создана таблица 2.
| Контакты атомов белка с | Полярные | Неполярные | Всего |
|---|---|---|---|
| остатками 2'-дезоксирибозы | 4 | 32 | 36 |
| остатками фосфорной кислоты | 6 | 32 | 38 |
| остатками азотистых оснований со стороны большой бороздки | 3 | 7 | 10 |
| остатками азотистых оснований со стороны малой бороздки | 0 | 2 | 2 |
С помощью программы nucplot была получена популярная схема ДНК-белковых контактов в комплексе 1h9t.
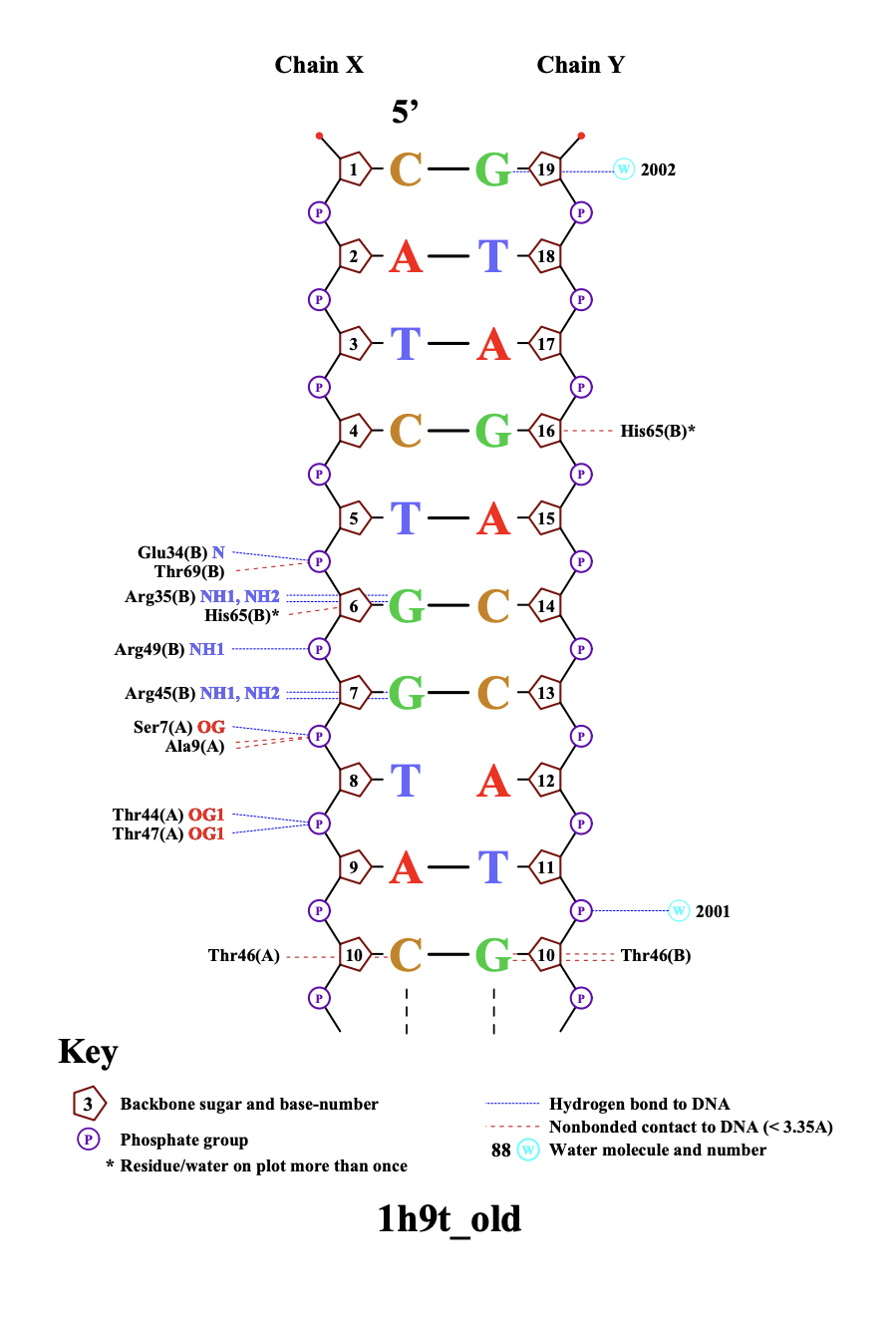
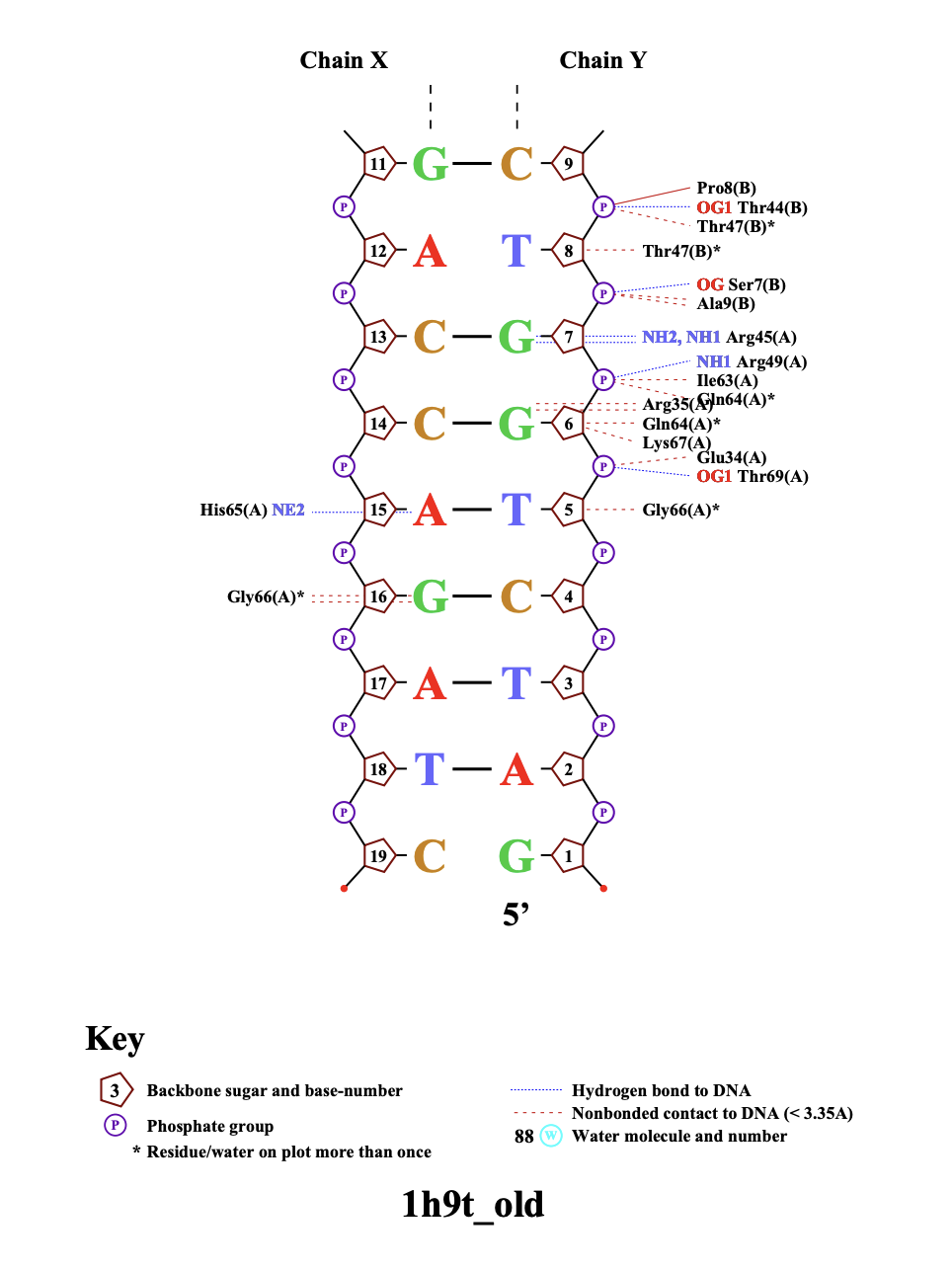
На основании предсказания nucplot можно заметить, что Arg35 и Arg45 специфично связываются с гуанином 6 и 7 соответственно, поэтому я выбрала эти аминокислотные остатки как наиболее важные для распознавания последовательности ДНК. Схема взаимодействий показана на рис.2 и 3.