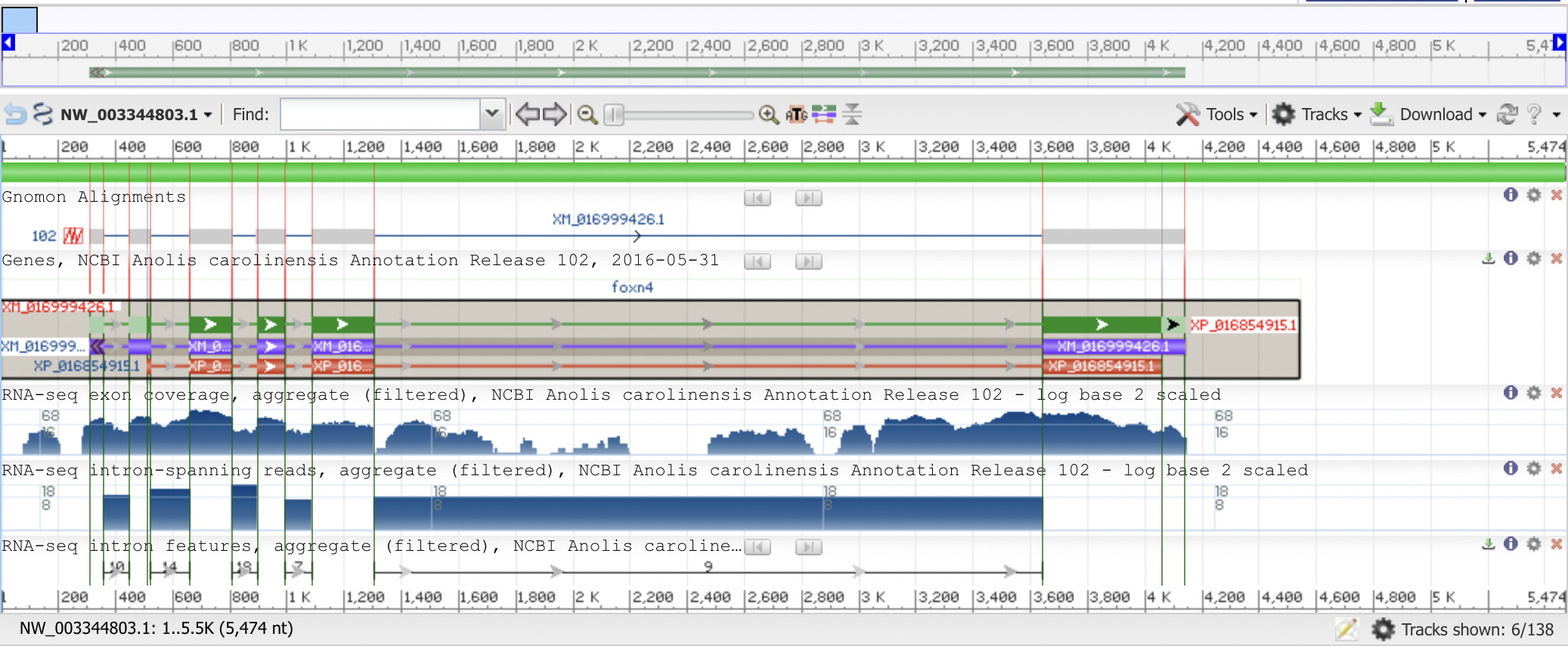
Для работы я выбрала контиг NW_003344803.1. Его длина составляет 5474 п.н. На рисунке 1 показана схема расположения генов на данном контиге.

В данном контиге есть 1 CDS, состоящий из 5 экзонов. Они показаны темно-зелеными прямоугольниками. Зеленые линии соответствуют интронам. Фиолетовым цветом показана мРНК, а красным - белок.
По ссылке доступен файл с последовательностью данного контига в FASTA-формате.
Далее был осуществлен поиск BLAST по эукариотическим последовательностям для контига. Для исключения был выбран таксон Хордовые, использовались следующие алгоритмы BLAST:
1. blastn - используется, если нужно получить нуклеотидные последовательности, гомологичные данной. При этом запрос не транслируется и поиск идет по нуклеотидной базе данных. Например, нужно найти гомологи гена тРНК.
Поиск: длина слова 11, 1498 находок, максимальное количество находок поменяла на 5000. Были найдены такие участки в хромосомах других организмов, хотя в исходной сборке этот контиг нелокализованный. Также найдено много предсказанных фрагментов.
2. megablast - используется, когда мы точно знаем, какому организму принадлежит секвенированная последовательность. Например, надо определить положение секвенированного фрагмента в хромосоме какого-то организма.
Поиск: длина слова 28, 10 находок (максимальное количество находок стояло 100). Также найдены последовательности, локализованные в хромосомах.
3. blastx - получает нуклеотидную последовательность и возвращает гомологичные белки, то есть транслирует запрос. Например, есть ген, нужно найти среди эукариот гомологи белка, который ген кодирует.
Поиск: длина слова 6, 4393 находок, максимальное количество находок поменяла на 5000. Неохарактеризованные белки, есть этот же белок (forkhead box N4) среди других таксонов.
4. tblastx - получает нуклеотидную последовательность и находит гомологичные, при этом транслируя запрос. Например, нужно найти последовательности, гомологичные гену, кодирующему мРНК.
Поиск: не было получено результатов при любой комбинации параметров.
Последовательности генома Anolis carolinensis были проиндексированы с помощью команды:
makeblastdb -in genome.fasta -dbtype nucl
Далее я скачала последовательности рРНК Escherichia coli и разделила их на 2 файла: для 16S рРНК и 23S рРНК. Обе рРНК играют структурную роль, выступая в качестве каркаса, определяющего положение рибосомальных белков, и связывают большую и малую рибосомальные субъединицы. 16S рРНК содержит последовательность анти-Шайна-Дальгарно, с помощью которой 16S рРНК связывается с мРНК, 23S рРНК входит в каталитический пептидилтрансферазный центр на рибосоме.
Затем был проведен локальный поиск BLAST отдельно для каждой рРНК по полученной базе данных с помощью алгоритма blastn с параметрами по умолчанию:
blastn -task blastn -query 16SrRNA_ecoli.fasta -db genome.fasta -out 16SrRNA_ecoli_out
blastn -task blastn -query 23SrRNA_ecoli.fasta -db genome.fasta -out 23SrRNA_ecoli_out
Данный алгоритм был выбран, так как было необходимо найти гомологи не кодирующей белок РНК, то есть запрос не нужно транслировать. Для 16S рРНК в топе находок участки хромосомы 1, 2, 3 и митохондриальный геном. Для 23S рРНК - участки 1 - 6 хромосом. К сожалению, во всех последовательностях отсутствуют аннотированные гены, поэтому сложно оценить полученные данные.