Для поиска гомологичных белков протеомы выбранных бактерий были собраны в один файл, который был проиндексирован командой makeblastdb. Затем был проведен поиск командой blastp с порогом на E-value 0,001
Cписок находок из выдачи BLAST представлен в файле.
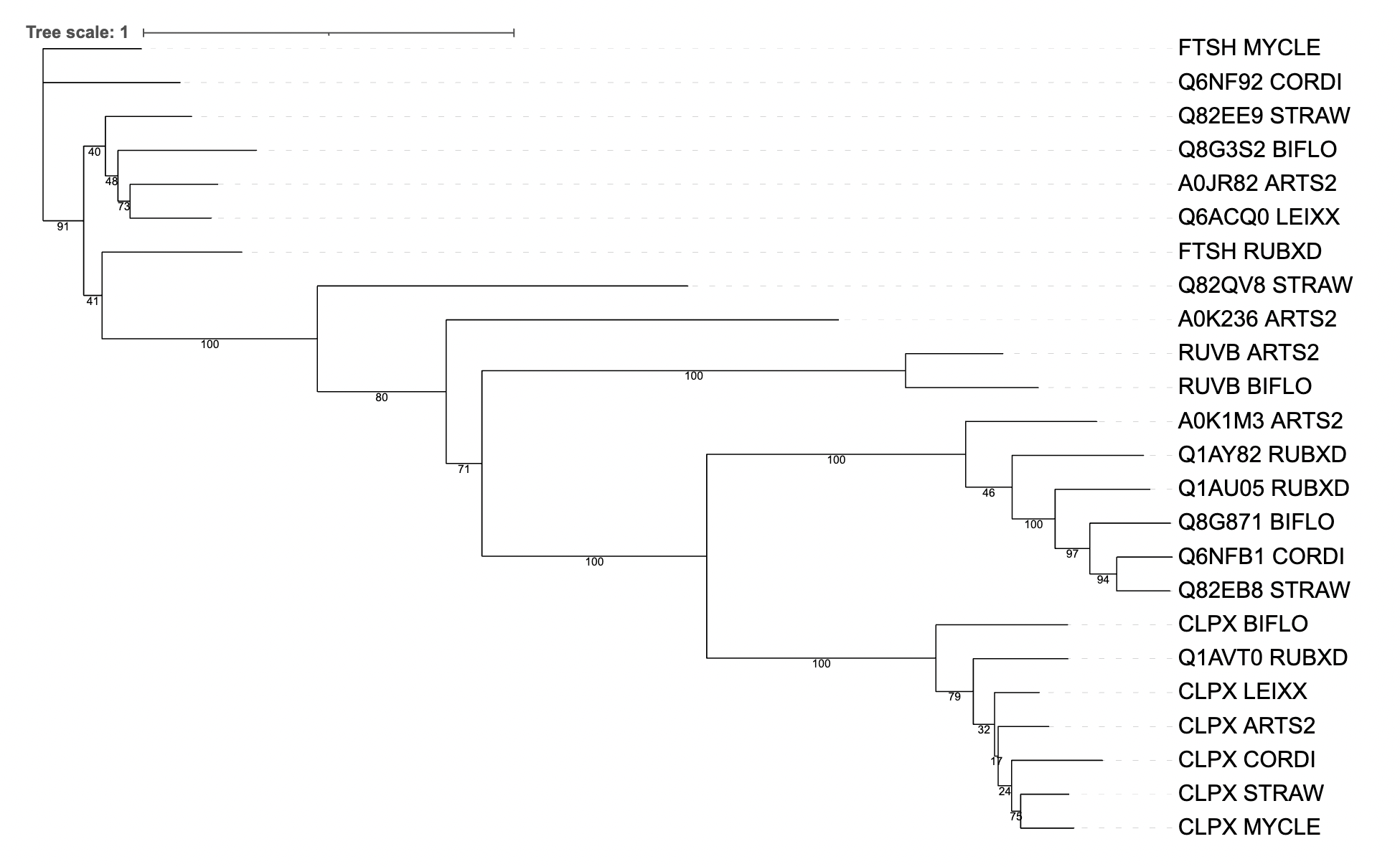
По найденным гомологам было построено дерево (рис.1) программой FastME. Его формула в Newick формате представлена в файле.

На основании полученного дерева можно выделить следующие ортологи: CLPX LEIXX и CLPX ARTS2, CLPX STRAW и CLPX MYCLE, A0JR82 ARTS2 и Q6ACQ0 LEIXX, а также паралоги: Q8G871 BIFLO и RUVB BIFLO, Q1AU05 RUBXD и Q1AY82 RUBXD, RUVB ARTS2 и A0JR82 ARTS2.
На рис.2 цветом показаны ортологичные группы, содержащие более 3 последовательностей.
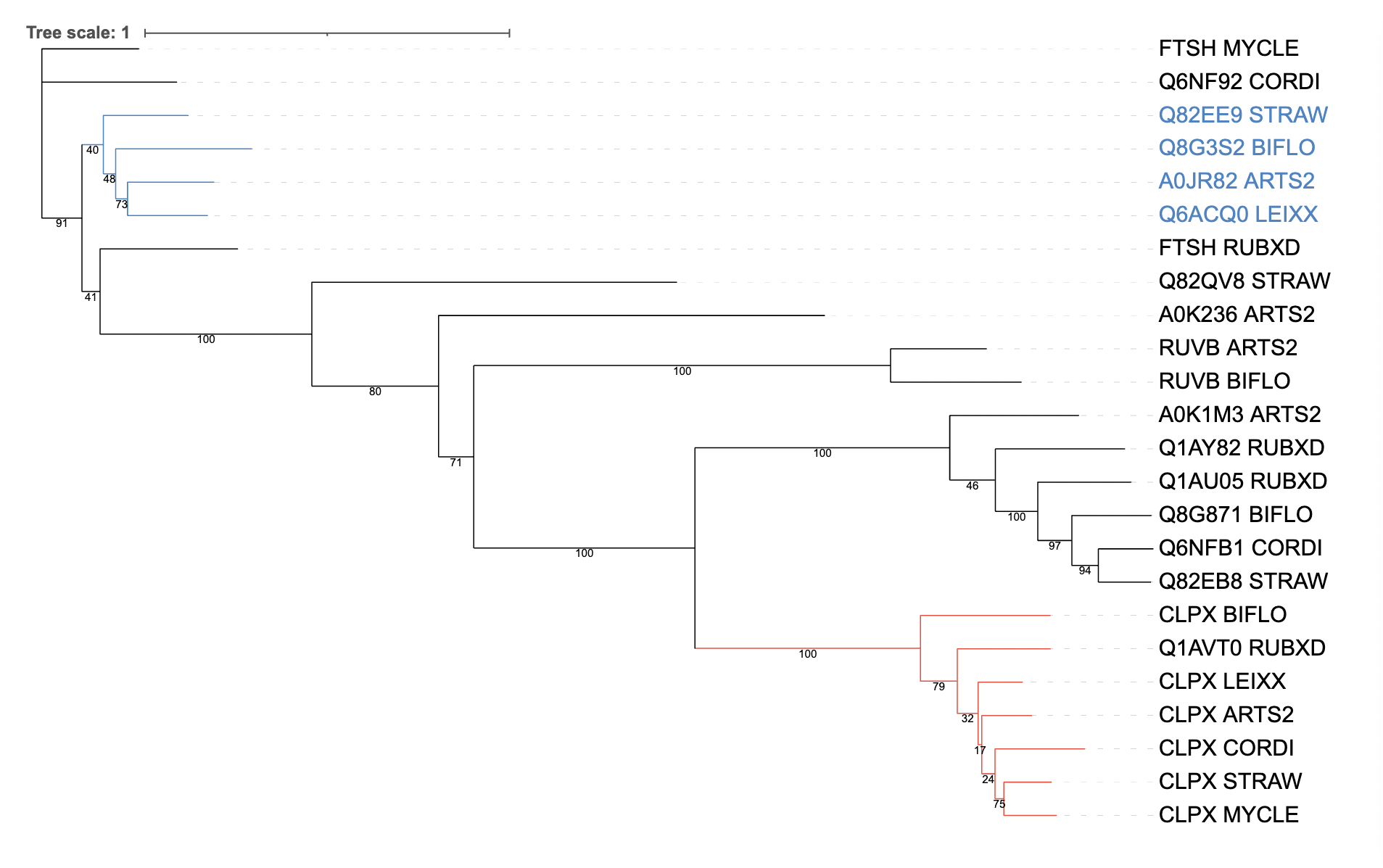
На рис.3 ортологичные группы объединены треугольниками.
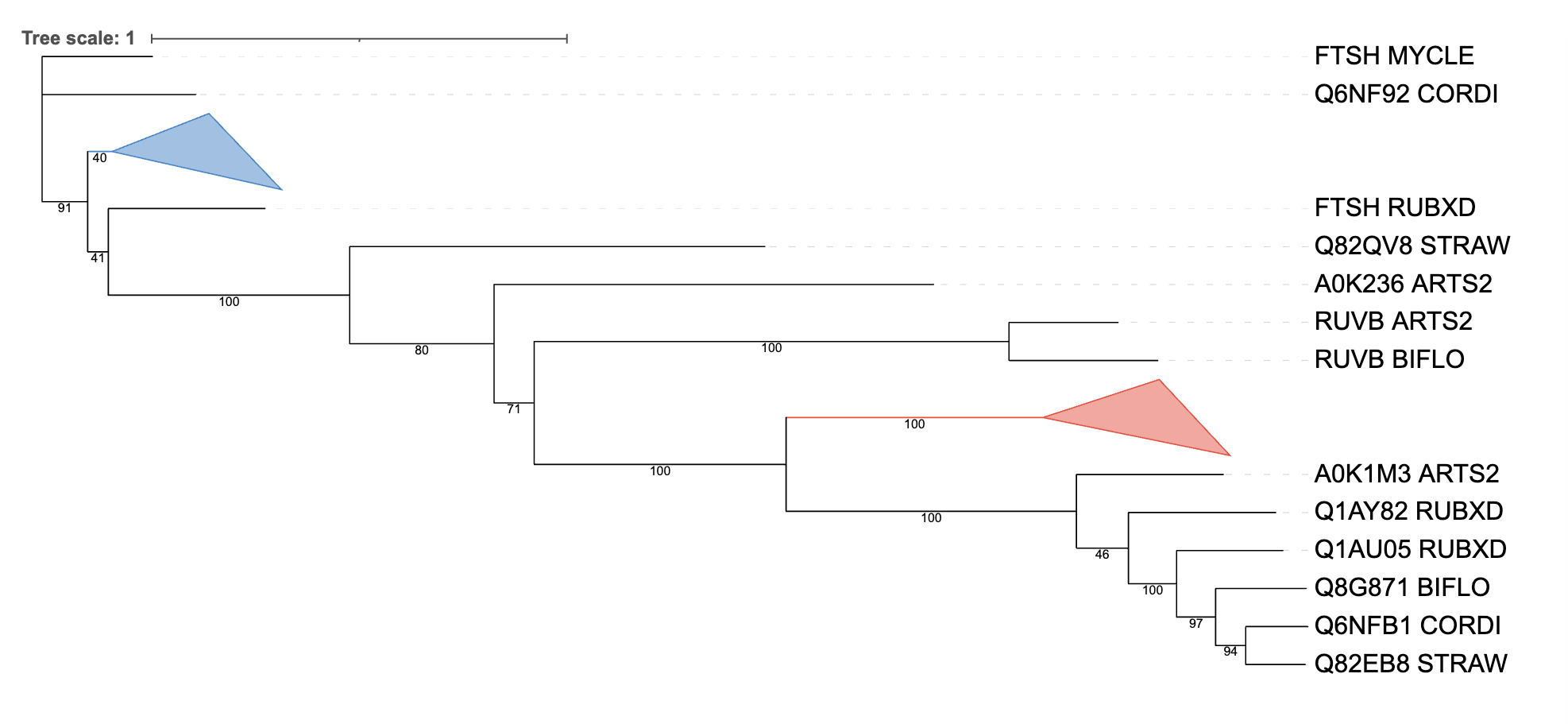
Среди обеих групп: АТФ-зависимых Clp протеаз и АТФ-зависимых цинковых металлопротеаз - выделяется ветвь {ARTS2, LEIXX}, соответствующая эталонному дереву. Синяя группа хорошо соответствует филогении бактерий, отделяя ветвь, которая тоже имеется в эталонном дереве: {ARTS2, LEIXX, BIFLO, STRAW}. В красной же группе имеются ветви, не соответствующие эталону: {MYCLE, STRAW, CORDI}, {MYCLE, STRAW, CORDI, ARTS2, LEIXX}.