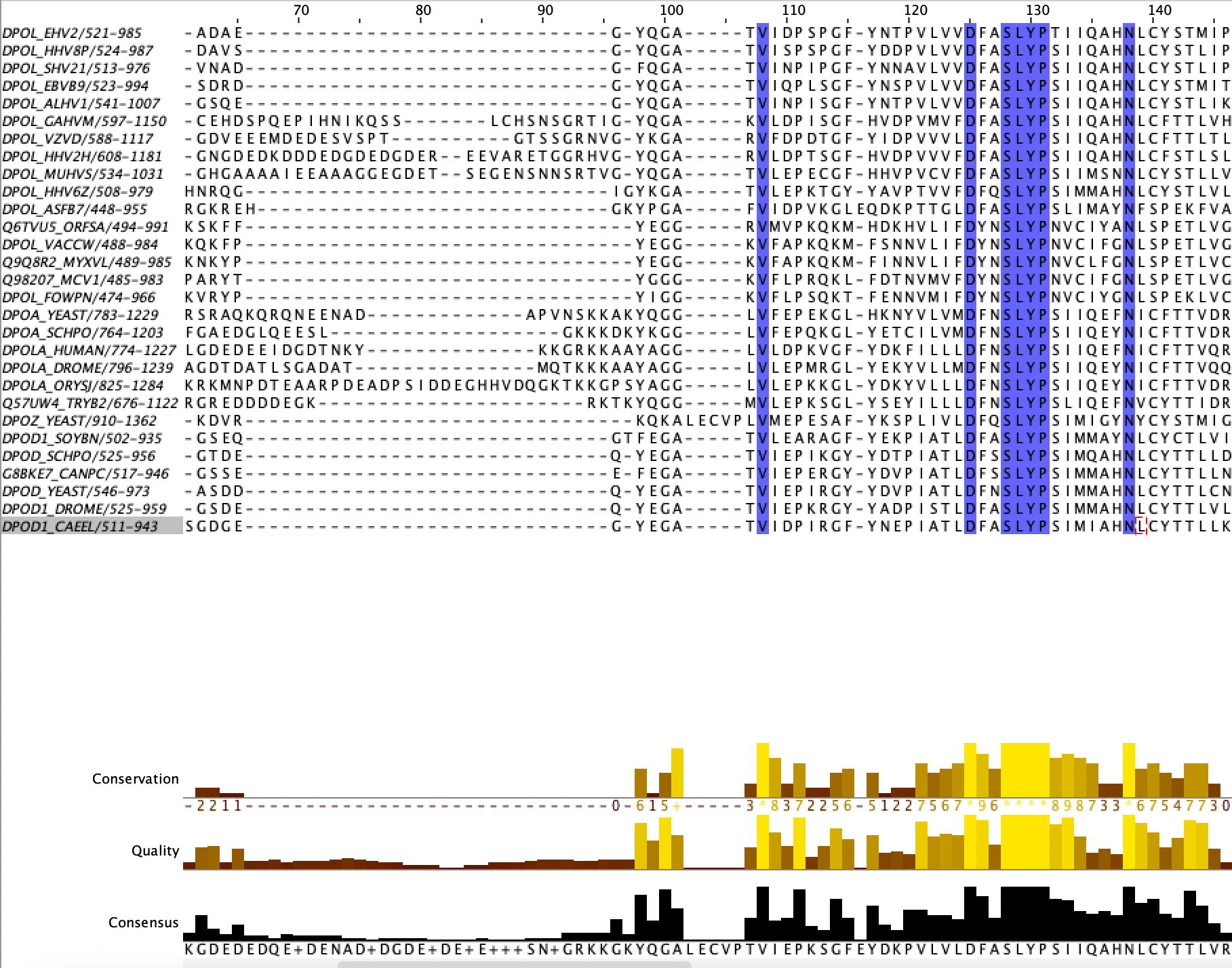
Для выполнения этого задания я выбрала семейство DNA_pol_B (PF00136). Выравнивание seed содержит 29 последовательностей, оно было скачано и открыто в программе Jalview (доступно по ссылке). Далее был выбран консервативный мотив, для которого составлен паттерн: D[FY].SLYP[NST][ILV][CIM].{3}N (Рис.1). С помощью функции Find в Jalview я нашла соответствия паттерна последовательностям выравнивания. Было получено, что во всех 29 последовательностях имеется этот мотив, что указывает на его высокое информационное содержание IC.
Затем по выбранному мотиву был выполнен поиск в базе данных SwissProt на сайте MyHits. Получили 96 находок, все из которых являются ДНК-полимеразами. По-видимому, этот мотив является характерным для данного семейства.

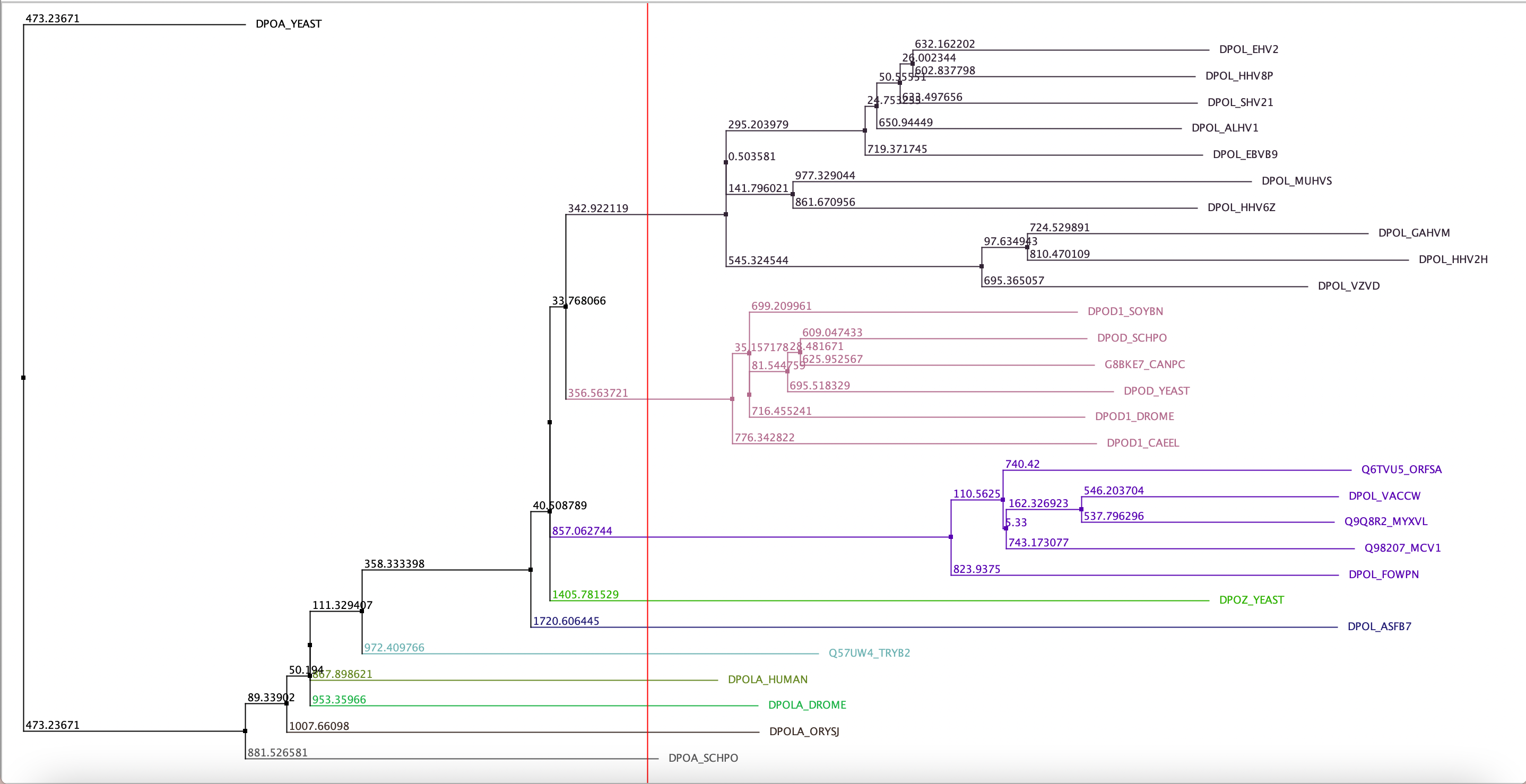
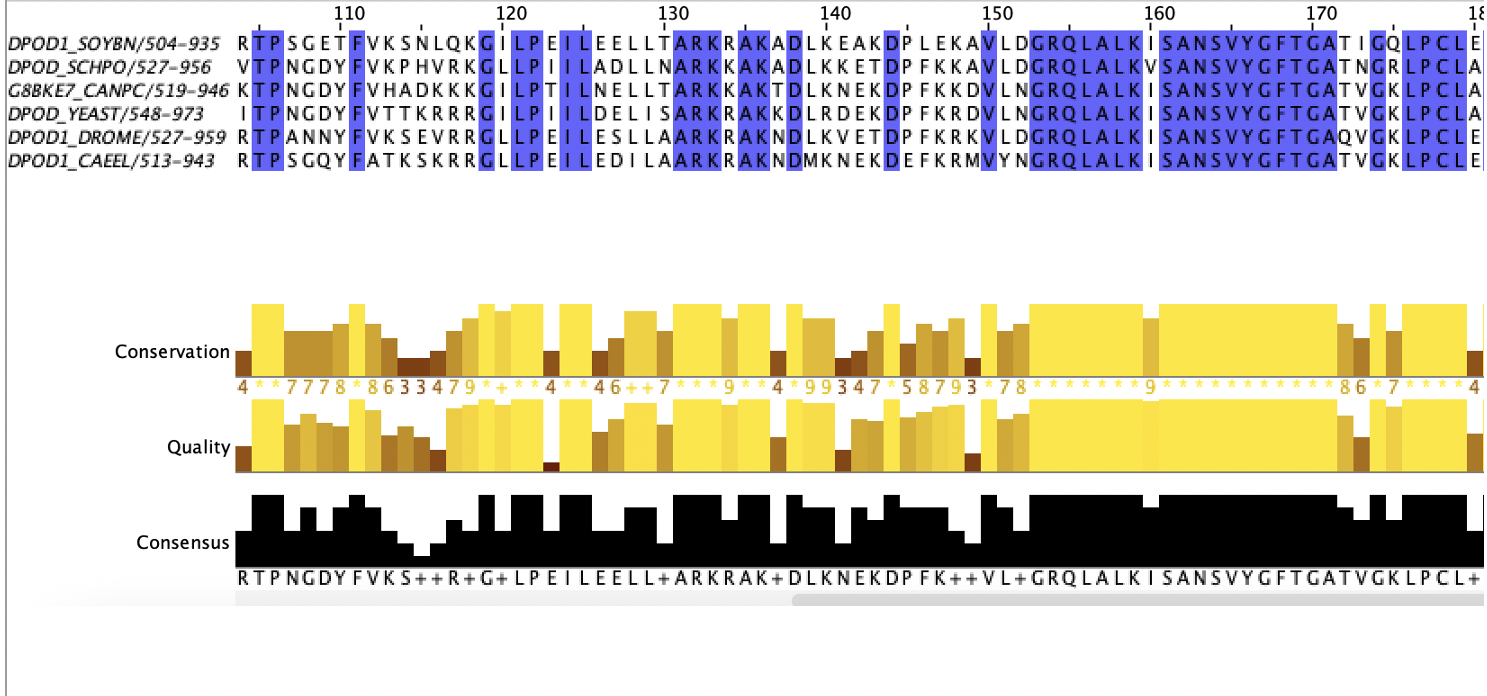
В Jalview было построено филогенетическое дерево методом NJ (Рис.2). Для поиска мотива я выбрала кладу из 6 белков, покрашенную на дереве розовым цветом. Выравнивание данной клады было перенесено в отдельное окно (доступно по ссылке). Далее по выравниванию был найден мотив GRQLALK[IV]SANSVYGFTGA (Рис.3). Затем был выполнен поиск этого мотива внутри клады и по всем последовательностям семейства. Получили, что этот мотив встречается во всех последовательностях клады и не встречается больше нигде в выравнивании. Из этого можно сделать вывод, что выбранный мотив хорошо характеризует данную кладу.
Для выполнения задания был выбран белок C4Z088 - предполагаемый белок-локализатор перегородки MinC, принадлежащий Eubacterium eligens. Это ингибитор клеточного деления, блокирующий образование полярных Z-колец, колеблясь между полюсами клетки и дестабилизируя формирующиеся филаменты FtsZ, прежде чем они сформируют Z-кольцо. Результаты итераций PSI-BLAST с выбранным белком приведены в Таблице 1.
| Номер итерации | Число находок выше порога (0,005) | Идентификатор худшей находки выше порога | E-value этой находки | Идентификатор лучшей находки ниже порога | E-value этой находки |
|---|---|---|---|---|---|
| 1 | 162 | Q2P036.1 | 0.004 | Q5F5V4.1 | 0.005 |
| 2 | 188 | O25693.2 | 2e-09 | - | - |
| 3 | 188 | Q9ZM51.1 | 1e-11 | A7H8E6.1 | 0.012 |
| 4 | 188 | Q9ZM51.1 | 5e-13 | A7H8E6.1 | 0.016 |
Было проведено 4 итерации. Видно, что уже на третьей количество находок перестало изменяться и наблюдается довольно большая разница между e-value худшей правильной находки и лучшей неправильной. Это говорит о том, что, скорее всего, находки составляют семейство гомологичных белков.
Для выполнения данного задания был использован геном бактерии Candidatus Phycosocius spiralis BOTRYCO-1. Ожидаемое число TA с учетом GC-состава равно 21276, а реальное = 14344. Проверка гипотезы осуществлялась с использованием теста хи-квадрат. Полученное p-value = 2.5e-295, что указывает на значимое различие между ожидаемым и реальным числом TA в геноме.