Ресеквенирование. Поиск полиморфизмов у человека.
В практикуме я работала с 4 хромосомой. Для индексирования референса была использована команда hisat2-build chr4.fasta chr4
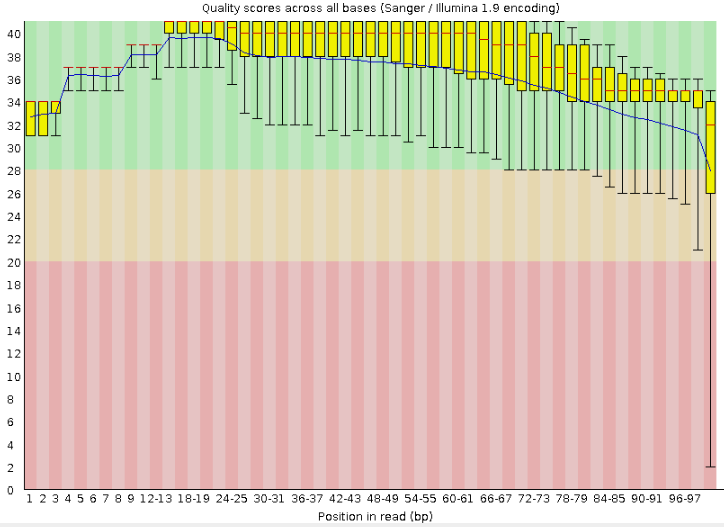
Для анализа качества чтений fastqc chr4.fastq
Для очистки чтений использовалась программа Trimmomatic, вызванная java -jar /nfs/srv/databases/ngs/suvorova/trimmomatic/trimmomatic-0.30.jar SE -phred33 chr4.fastq chr4_trimmed.fastq TRAILING:20 MINLEN:50
Анализ качества чтений после триммированияfastqc chr4_trimmed.fastq
Выравнивание прочтений (триммированных) и референса в формате .sam hisat2 -x chr4 -U chr4_trimmed.fastq -S chr4_aligned.sam --no-softclip --no-spliced-alignment
Перевод файла с результатами выравнивания в бинарный формат .bam samtools view -b chr4_aligned.sam -o chr4_aligned.bam
Сортировка выравнивания чтений с референсом по координате начала чтения в референсе samtools sort chr4_aligned.bam chr4_sorted.bam
Индексирование отсортированного .bam-файла samtools index chr4_sorted.bam
Вывод информации о картировании (нужно узнать число откартированных на референс чтений) samtools flagstat chr4_aligned.sam.
Создание .bcf-файла с полиморфизмами samtools mpileup -uf chr4.fasta chr4_sorted.bam -o chr4_snp.bcf
Создание файла со списком отличий между референсом и чтениями в формате .vcf bcftools call -cv chr4_snp.bcf -o chr4_snp.vcf
Затем были описаны три полиморфизма из .vcf-файла, описание представлено ниже.
Удаление инделей из .vcf-файла (чтобы аннотировать только полученные полиморфизмы и не аннотировать индели)
vcftools --vcf chr4_snp.vcf --remove-indels --recode --out chr4_noindels
Создание входного файла для программы, аннотирующей полиморфизмыconvert2annovar.pl -format vcf4 chr4_noindels.recode.vcf -outfile chr4_snp.avinput
Аннотация полиморфизмов по базе данных RefGene annotate_variation.pl -out chr4_refgene -build hg19 chr4_snp.avinput /nfs/srv/databases/annovar/humandb.old/
Аннотация по базе dbsnp annotate_variation.pl -filter -out chr4_dbsnp -build hg19 -dbtype snp138 chr4_snp.avinput /nfs/srv/databases/annovar/humandb.old/
Аннотация по базе 1000 genomes annotate_variation.pl -filter -dbtype 1000g2014oct_all -buildver hg19 -out chr4_1000genomes chr4_snp.avinput /nfs/srv/databases/annovar/humandb.old/
Аннотация по базе Gwas annotate_variation.pl -regionanno -build hg19 -out chr4_gwas -dbtype gwasCatalog chr4_snp.avinput /nfs/srv/databases/annovar/humandb.old/
Аннотация по базе Clinvar annotate_variation.pl -filter -dbtype clinvar_20150629 -buildver hg19 -out chr4_clinvar chr4_snp.avinput /nfs/srv/databases/annovar/humandb.old/
Анализ качества чтений и их триммирование
Исходно я получила 5810 чтений.

Описание полиморфизмов
Первый полиморфизм.
Координата - 68458937, произошла замена G в референсе на С в чтении, качество чтения 225,009; глубина покрытия 87.
Второй полиморфизм.Координата 88959565, вставка двух нуклеотидов: ATGTGTGTGTGTGTGTGTGT в референсе; ATGTGTGTGTGTGTGTGTGTGT в чтениях;
качество чтения 17,548; глубина покрытия 1.
Третий полиморфизм.Координата 187165891, делеция одного нуклеотида: GTTTTT в референсе; GTTTT в чтениях; качество чьения 3,66479; глубина покрытия 2.
Из 50 полиморфизмов 4 инделя и 46 SNP.
RefGene делит однонуклеотидные полиморфизмы по их расположению в последовательности, 36 из них маркированы как intronic, 2 - downstream,
3 - intergenic, 3 - exonic, 1 - UTR3.
11 однонуклеотидных полиморфизмов произошли в гене STAP1, 13 в гене MEPE, 3 в KIAA0922 (хотя они маркированы как intergenic), 17 в гене KLKB1.
Для 3 замен указан результат, 2 из них являются несинонимичными:
line30 nonsynonymous SNV KLKB1:NM_000892:exon5:c.G428A:p.S143N, chr4 187158034 187158034 G A hom 221.999 . line38 nonsynonymous SNV KLKB1:NM_000892:exon10:c.A1072G:p.T358A, chr4 187172943 187172943 A G het 225.009 .а 1 синонимичной:
line44 synonymous SNV KLKB1:NM_000892:exon15:c.T1761C:p.N587N, chr4 187179210 187179210 T C het 225.009 .
42 из 50 полиморфизмов имеют rs (rs указаны в выходном файле аннотации по базе snp168)
0,396893158 - средняя частота однонуклеотидных замен для 39 полиморфизмов, обнаруженных в базе данных 1000genomes. Самый распространённый полиморфизм имеет частоту 0,91853, это замена аденина на гуанин в 187179486 позиции; самый редкий имеет частоту 0,003794, это тоже замена аденина на гуанин в позиции 68456397.
Согласно базе данных GWAS, замена аденина на гуанин в 68447249 позиции связана с болезнью Паркинсона, замена тимина на цитозин в 88755828 позиции повышает риск сердечнососудистых заболеваний, замена гуанина на аденин в позиции 187149540 влияет на уровень метаболизма, а та же замена в позиции 187158034 ассоциирована с ожирением.
Клиническая аннотация полиморфизмов по базе данных ClinVar: 68447249 ассоциирован с болезнью Паркинсона, 88755828 является фактором риска для сердечнососутистых заболеваний, 187149540 регулирует уровень метаболитов, замена гуанина на аденин в 187158034 (та, которая в GWAS аннотирована как связанная с ожирением) патогенна и ассоциирована с дефицитом прекалликреина (сериновая протеаза, известная как фактор Флетчера).