Сборка de novo генома Buchnera aphidicola с кодом доступа проекта SRR4240380
Пункт 1. Подготовка чтений.
wget ftp://ftp.sra.ebi.ac.uk/vol1/fastq/SRR424/000/SRR4240380/SRR4240380.fastq.gz - команда, использованная для
скачивания fastq-файла с чтениями.
gunzip SRR4240380.fastq.gz - распаковываем архив с чтениями. После распаковки файл имеет размер 550 813 500 байт.
java -jar /nfs/srv/databases/ngs/suvorova/trimmomatic/trimmomatic-0.30.jar SE -phred33 SRR4240380.fastq noad.fastq ILLUMINACLIP:adapters.fasta:2:7:7 -
удаление возможных остатков адаптеров для Illumina. Все 5217318 последовательностей не содержали адаптеров, поэтому не были удалены. Соответственно, размер файла не изменился.
java -jar /nfs/srv/databases/ngs/suvorova/trimmomatic/trimmomatic-0.30.jar SE -phred33 noad.fastq noad_trimmed.fastq TRAILING:20 MINLEN:32 -
обрезка чтений длиной меньше 32 и с качеством ниже 20. Были удалены 250651 (4,8%) чтений и осталось 4966667 (95,2%). После удаления
этих чтений размер файла стал 523 536 678 байт.
Пункт 2. Запуск программ velveth и velvetg, анализ результатов.
velveth . 31 -short -fastq noad_trimmed.fastq - подготовка k-меров длины 31.
velvetg . -cov_cutoff auto - сборка на основе подготовленных k-меров длины 31.
N50 сборки составило 12520, а среднее покрытие 12,72303. Три самых длинных контига имеют длину 25915 (покрытие 27,422072);
23850 (покрытие 24,777358);23807 (покрытие 25,742975). Есть контиги с аномально большим покрытием: так, контиг №11 имеет длину 2106 и покрытие 125,951092.
Контиг №54 имеет длину 934 и покрытие 130,479657. Контиг №77 имеет длину 3 и покрытие 754 (такое большое значение объясняется крайне маленькой длиной контига).
NODE_3_length_25915_cov_27.4220712004 to 11103;613658 to 620926;621055 to 627104 - координаты контига на бактериальной хромосоме,
то есть в результате имеем 3 выравнивания, между двумя последними дистанция маленькая, а вот первый от второго отделают несколько десятков тысяч нуклеотидов.
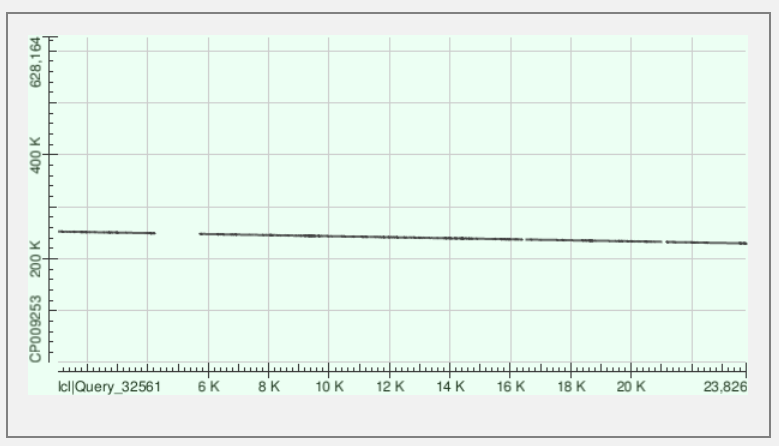
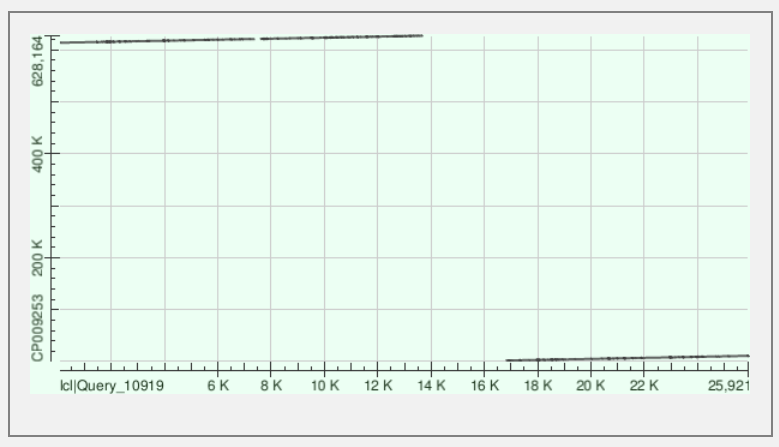
Ниже на первом рисунке видно, как эти три выравнивания распределяются по контигу, то есть какие участки контига выровнялись с бактериальной хромосомой, а на втором рисунке представлена карта локального сходства двух
последовательностей, на которой видно, как выровненные участки расположены на каждой из них.
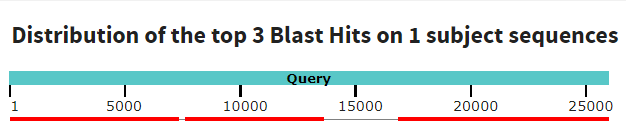
>NODE_20_length_23850_cov_24.777359573092 to 582686 - 7212/9822(73%) нуклеотидов в последовательностях идентичны, 461/9822(4%)
гэпов в выравнивании, цепи для обеих последовательностей прямые; 584329 to 587055 - координаты второго выравнивания, расположенного через
2000 нуклеотидов бактериальной хромосомы от первого с числом совпавших нуклеотидов 2100/2777(76%) и количеством гэпов 108/2777(3%); 593743 to 594099 -
координаты на бактериальной хромосоме последнего выровненого участка контига, в выравнивании 289/359(81%) идентичных нуклеотидов и 4/359(1%) гэпов,
оно имеет длину всего 359 нуклеотидов. Рисунки изображают то же, что и для предыдущего контига.
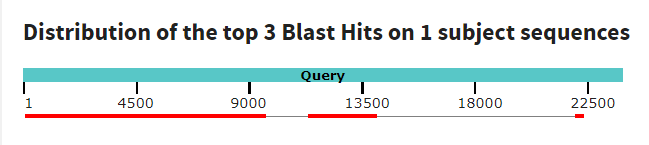
>NODE_20_length_23850_cov_24.777359236918 to 247596 - координаты первого выравнивания с процентом идентичности 8182/10884(75%) и 391/10884(3%)
гэпами, причем выравнивается прямая последовательность контига с обратной цепью бактериальной хромосомы; координаты второго выравнивания
232358 to 236859, в нём 3466/4581(76%) совпавших нуклеотидов и 130/4581(2%) гэпов, в нём тоже противоположные цепи; 229411 to 232057 - координаты
третьего выравнивания с числом совпадений 2156/2685(80%) и числом гэпов 71/2685(2%); координаты четвертого выравнивания:
248967 to 252161, в нём 2527/3246(78%) совпадений и 94/3246(2%) гэпов.