Паралоги и визуализация
Задание 1. Определение таксономии и функции прочтённой последовательности из практикума 6.
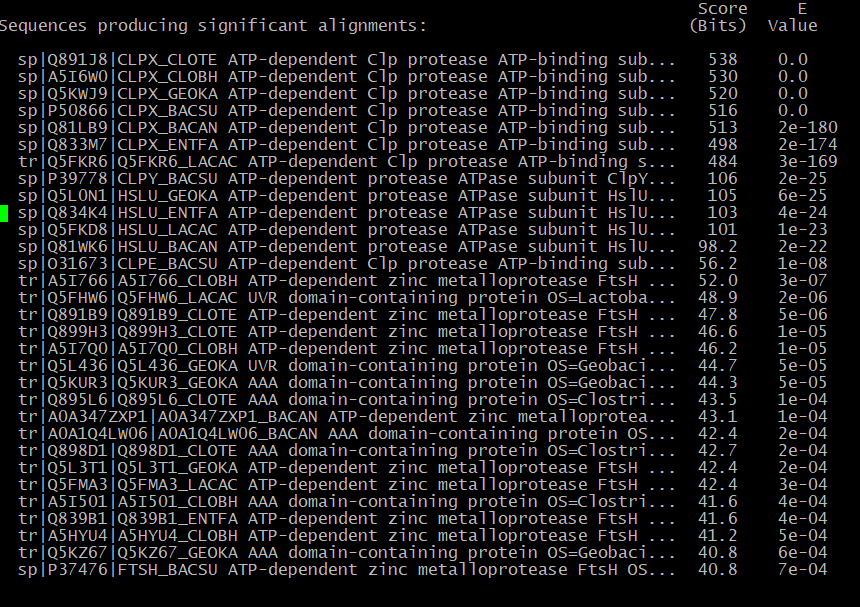
Поиск при помощи blastp по протеомам бактерий, выбранных в первом практикуме, гомологов заданного белка CLPX_ECOLI выдал несколько последовательностей. Находки из выдачи представлены на рисунке ниже.
Задание 2. Реконструкция и визуализация.
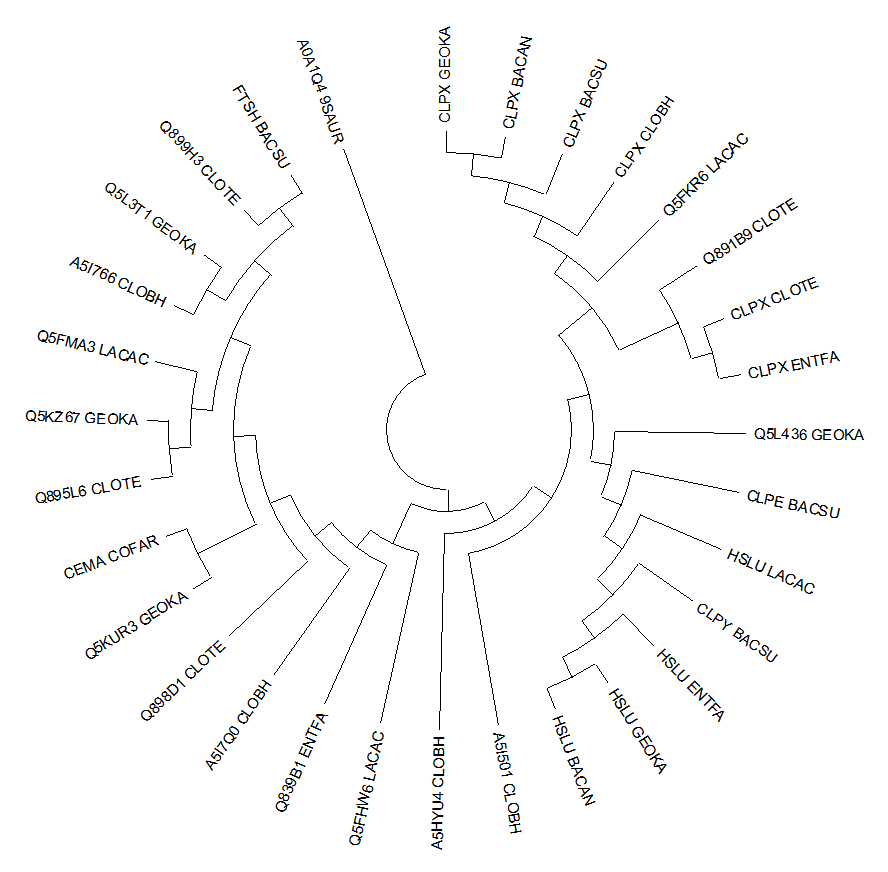
Множественное выравнивание было получено в программе MEGA, куда последовательности были импортированы. Затем
в той же программе с помощью метода Maximum Likelihood было построено филогенетическое дерево в соответствии с полученным
выравниванием. Дерево было визуализировано в программе MEGA, результаты визуализации представлены на рисунке ниже.
(((((((((sp|Q5KWJ9|CLPX_GEOKA,sp|Q81LB9|CLPX_BACAN),sp|P50866|CLPX_BACSU),sp|A5I6W0|CLPX_CLOBH),tr|Q5FKR6|Q5FKR6_LACAC),(tr|Q891B9|Q891B9_CLOTE,(sp|Q891J8|CLPX_CLOTE,sp|Q833M7|CLPX_ENTFA))),(tr|Q5L436|Q5L436_GEOKA,(sp|O31673|CLPE_BACSU,(sp|Q5FKD8|HSLU_LACAC,(sp|P39778|CLPY_BACSU,(sp|Q834K4|HSLU_ENTFA,(sp|Q5L0N1|HSLU_GEOKA,sp|Q81WK6|HSLU_BACAN))))))),tr|A5I501|A5I501_CLOBH),tr|A5HYU4|A5HYU4_CLOBH),(tr|Q5FHW6|Q5FHW6_LACAC,(tr|Q839B1|Q839B1_ENTFA,(tr|A5I7Q0|A5I7Q0_CLOBH,(tr|Q898D1|Q898D1_CLOTE,((tr|Q5KUR3|Q5KUR3_GEOKA,sp|A0A347|CEMA_COFAR),(((tr|Q895L6|Q895L6_CLOTE,tr|Q5KZ67|Q5KZ67_GEOKA),tr|Q5FMA3|Q5FMA3_LACAC),((tr|A5I766|A5I766_CLOBH,tr|Q5L3T1|Q5L3T1_GEOKA),(tr|Q899H3|Q899H3_CLOTE,sp|P37476|FTSH_BACSU)))))))),tr|A0A1Q4|A0A1Q4_9SAUR);
Полученное дерево в формате Newick.
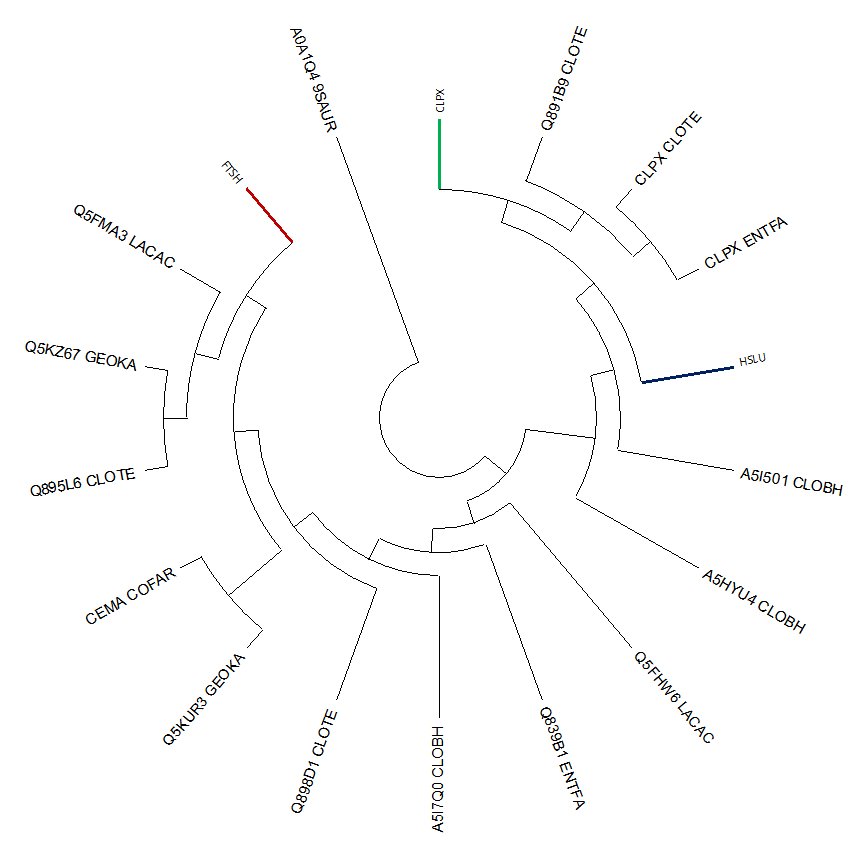
Пары паралогов: CLPX_CLOTE и Q891B9_CLOTE, CLPE_BACSU и CLPY_BACSU, A5HYU4_CLOBH и A5I501_CLOBH.
Пары ортологов: CLPX_GEOKA и CLPX_BACAN, FTSH_BACSU и Q899H3_CLOTE, HSLU_ENTFA и HSLU_GEOKA.
Ниже представлено первое изображение дерева с покрашенными в различные цвета
ортологичными группами.
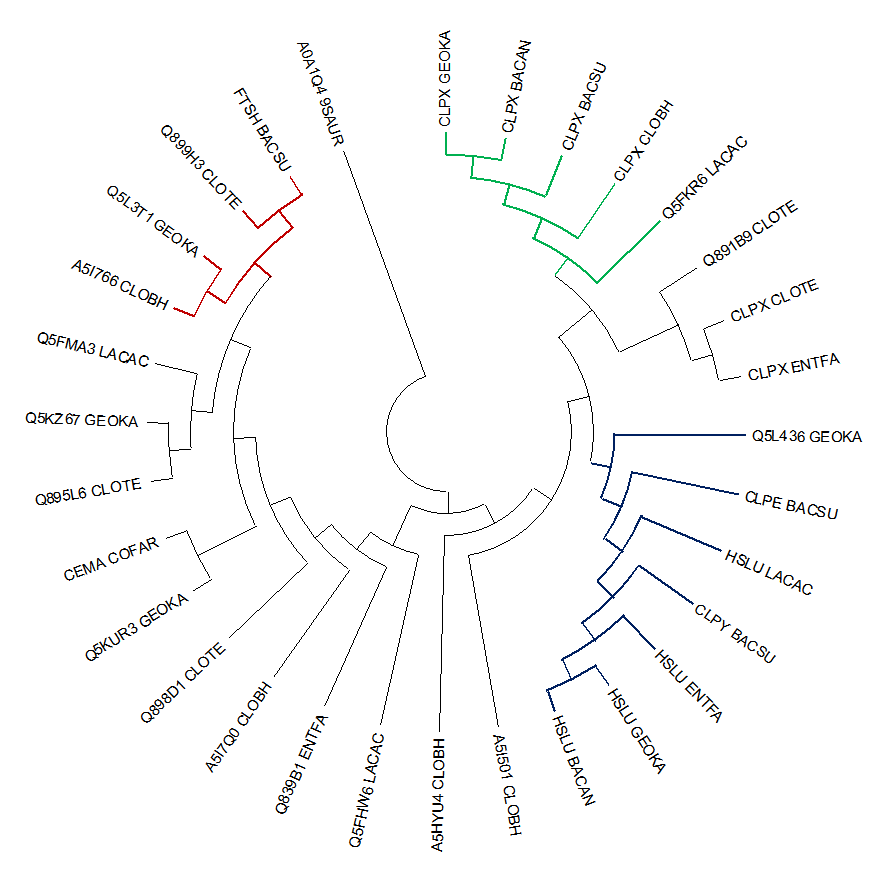
Все ортологичные группы, содержащие больше трёх последовательностей, объединены на рисунке ниже - второе изображение дерева.
Таких группы оказалось три:
CLPXATP-dependent Clp protease ATP-binding subunit ClpX.
В неё входят собственно белки с мнемоникой CLPX, найденные у всех организмов, кроме CLOTE,
ENTFA, и LACAC, которому принадлежит белок Q5FKR6, тоже вошедший в эту ортологическую группу.
Реконструированное дерево близко к филогенетическому дереву самих организмов.
Отличия только в том, что организмы BACAN и BACSU состоят в более тесном родстве
друг с другом, чем с GEOKA (на реконструированном дереве BACAN И GEOKA ближайшие ветви).
HSLUАTP-dependent protease ATPase subunit HslU.
В неё вошли белки HSLU, CLPY, CLPE, Q5L436, принадлежащие бактериям GEOKA, BACSU, LACAC, ENTFA, BACAN.
реконструированное дерево не соответствует дереву самих организмов,
потому что в эту группу входят несколько паралогов и в ней объединены белки с разной мнемоникой, которые у близких организмов могут существенно отличаться (например, когда-то они были паралогами, до разделения видов).
FTSHATP-dependent zinc metalloprotease.
В группу вошли 4 белка, FTSH, Q899H3, Q5l3T1, A5I766. Они принадлежат CLOBH, GEOKA, CLOTE, BACSU.
Реконструированное дерево белков не совпадает с деревом бактерий, поскольку сами белки кажутся сильно отличающимися друг от друга.