Задание 1
Файлы
Оба файла, с хроматограммой прямой и обратной цепи, были открыты в программе Chromas Lite.
Обратная цепь была изменена на комплементарную и перевернута (опция reverse complement), чтобы можно было сравнивать последовательности обеих цепей.
С помощью опции find последовательности были выровнены относительно друг друга.
Затем были определены границы нечитаемых 5'- и 3'-участков в каждой последовательности. Координаты определялись по прямой цепи.
Необходимо заметить, что после удаления нечитаемых участков нумерация нуклеотидов автоматически изменилась, так что приведенные координаты соответствуют исходным файлам, но не соответствуют файлам, полученным в ходе дальнейшей работы.
Границы нечитаемых участков:
Прямая цепь
Начало прочтения: 0-98
Конец прочтения: 680 - до конца
Обратная цепь
Начало прочтения:0-152
Конец прочтения: не представлен на прямой цепи
Далее нечитаемые 5'- и 3'- концы были удалены.
Характеристика качества хроматограмм
Прямая цепь
Обратная цепь
Редактирование последовательностей
Для начала следует привести пример хорошего участка, прочтение которого однозначно:
>
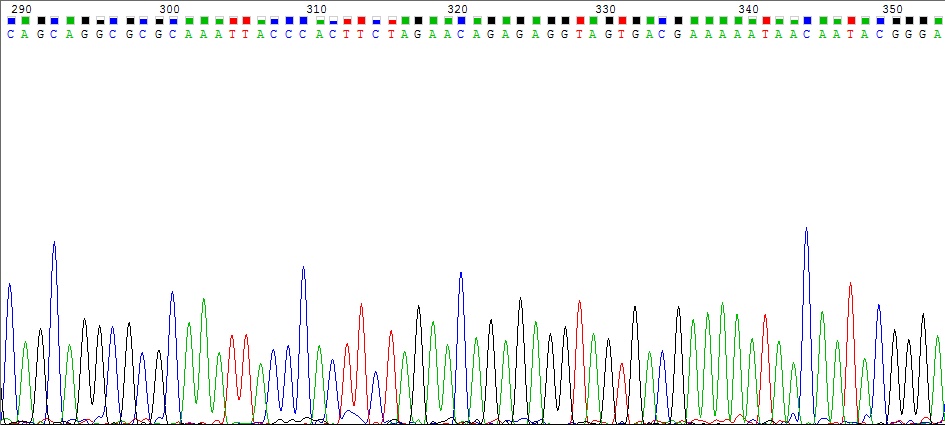
Рисунок 1"Хороший" участок хроматограммы. Прямая цепь наверху, обратная внизу. Видно, что quality values высокие.
Редактирование прямой цепи
Проблема 1. Наложение пиков
Программа не смогла прочесть 315 нуклеотид в прямой цепи. Последовательность однозначно восстанавливается по обратной, в которой проблем в данном месте нет.
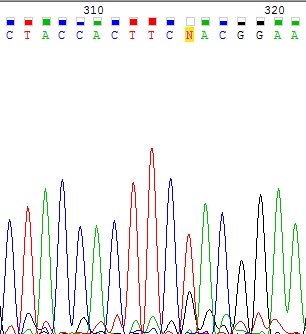
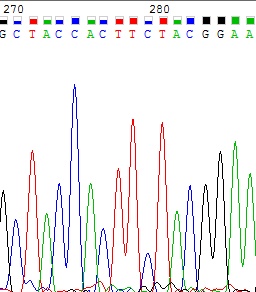
Рисунок 2 Прямая цепь вверху, обратная - внизу.
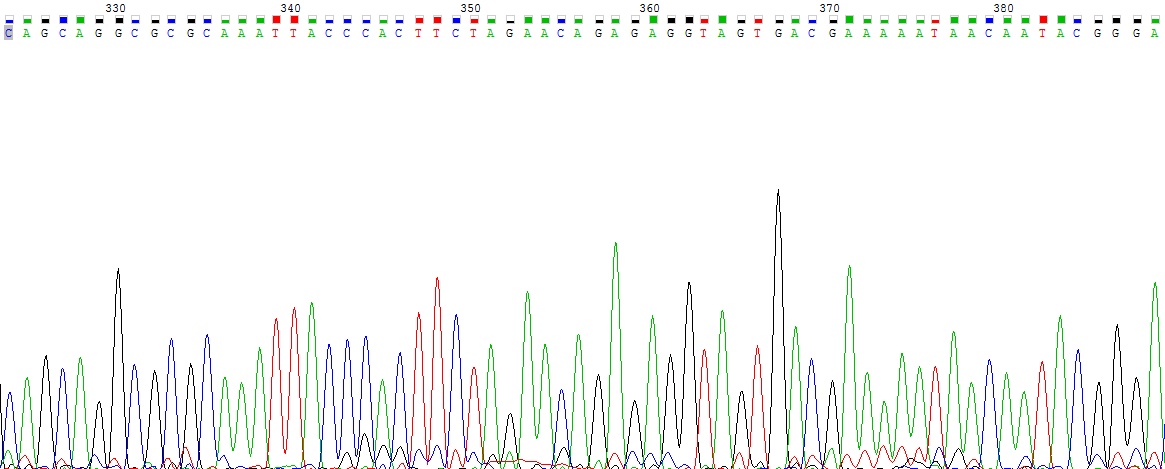
Проблема 2. Сильный шум и вновь наложение пиков.
На представленном участке прямой последовательности наблюдается большая высота пиков и их наложение друг на друга. Сопоставляя участок с участком обратной последовательности, видим, что программа прочла все верно.
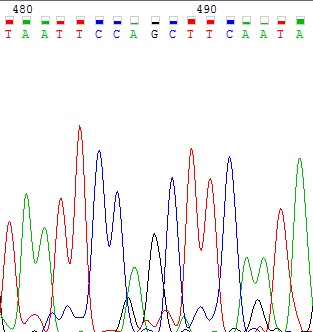
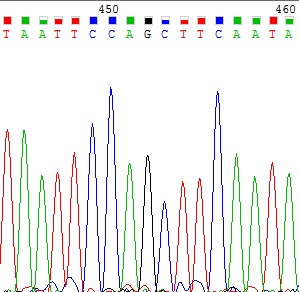
Рисунок 3 Прямая цепь вверху, обратная - внизу.
Проблема 3. Шум выходит на уровень сигнала.
Программа не смогла прочесть 504 нуклеотид прямой цепи. Он восстанавливается при сопоставлении с обратной.
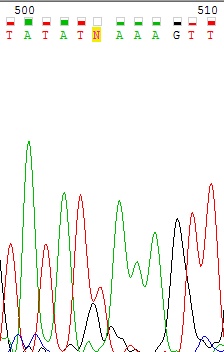
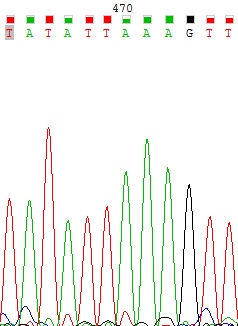
Рисунок 4 Прямая цепь вверху, обратная - внизу.
Редактирование обратной цепи
Проблема 4. Сильный шум на фоне слабого сигнала.
Программа не смогла прочесть 24 нуклеотид обратной цепи. Он восстанавливается при сопоставлении с прямой.
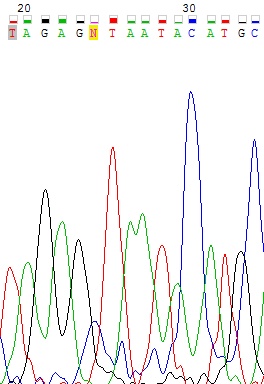
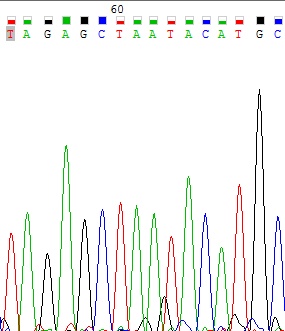
Рисунок 4 Обратная цепь вверху, прямая - внизу.
Задание 2
Требовалось привести пример нечитаемой хроматограммы
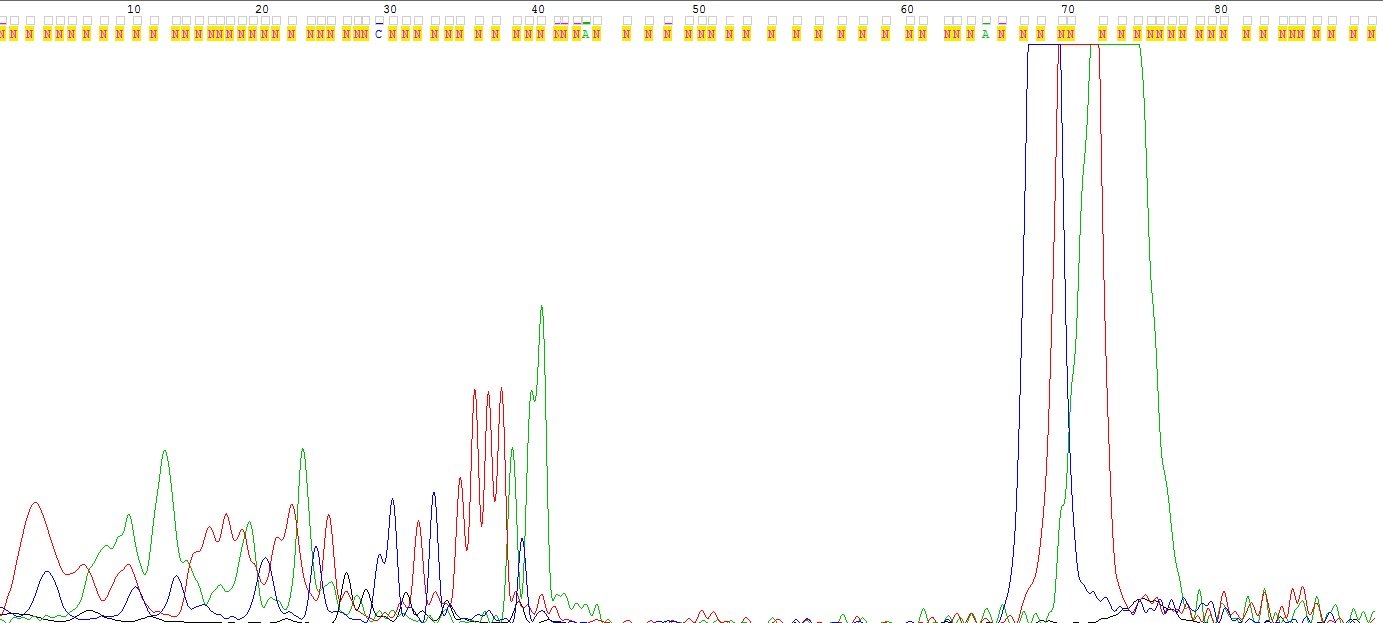
Рисунок 5 Участок хроматограммы, в котором программа не смогла прочесть практически ни один нуклеотид. Взят файл WS2943_SP6R.ab1
© Козлова Анастасия, 2015