Эволюционные домены. Pfam, InterPro.
- Опиcание доменной структуры C0QAT7_DESAH по данным Pfam.
- Представленность домена PF02110 в организмах разных видов
- Представленность доменов белка B5CLH5_9FIRM в белках Bacillus subtilis Так как в белке C0QAT7_DESAH представлен только один тип доменов (PF02110) и количество белков с ним у Bacillus subtilis равно 1, я решил выбрать другой белок (B5CLH5_9FIRM), содержащий помимо данного домена (PF02110) и другие: TMP-TENI (PF02581), Phos_pyr_kin (PF08543).
- Доменные перестройки
- В белках часто встречается разрыв в домене PF02110 (HK) C0QAT7_DESAH:
- Наиболее частно домен PF02110 (HK) можно обнаружить в сочетании с доменом PF02581 (TMP-TENI) THI4_SCHPO:
- Домен PF02110 (HK) встречается как на N-конце так и на C-конце B5CLH5_9FIRM :
- Ссылка на выравнивание-затравку(seed) seed.msf
- Сравнение и описание мотивов в разных БД(для белка THIM_BACSU)
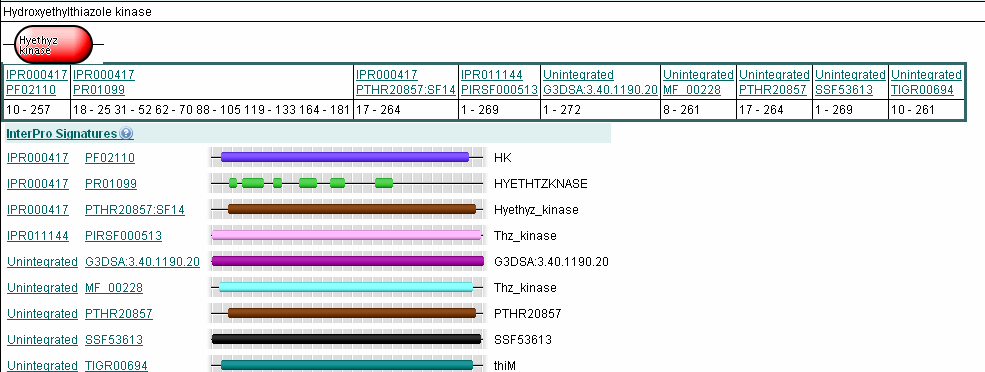
- Самый короткий мотив - HYETHTZKNASE (PR01099). Найден в SPRINT
- Cамый длинный мотив - G3DSA:3.40.1190.20 (G3DSA:3.40.1190.20).Описан в CATH protein structure classification. Тип распохнающего правила - CATH Code
- Структурные подписи интегрированы в InterPro:

-
Да отличаются:
Для PF02110 (Pfam)БД №№ а.о. Pfam 14-256 SPRINT 18 - 25, 31 - 52, 62 - 70, 88 - 105, 119 - 133, 164 - 181 PANTHER 17 - 264 PIRSF 1 - 269 CATH 1-272 ExPASy 8 - 261 SuperFamily 1-269 HMM 10-261
Cхема из Pfam: |
|||||
| На схеме отмечены Pfam домены, найденный в белке C0QAT7_DESAH |
|||||
| № | Pfam AC | Pfam ID | Полное название семейства доменов |
Положение в последовательности белка C0QAT7_DESAH | Клан |
| 1. | PF02110 | HK | Семейство гидрокситиазол киназ. Семейство доменов названо по названию фермента гидрокситиазол киназы, который катализирует реакцию синтеза тиамин пирофосфата. | 32–179 | Это семейство является членом клана рибокиназ (CL0118), который содержит 5 семейств ADP_PFK_GK, Carb_kinase, HK, PfkB, Phos_pyr_kin |
| 2. | 200–296 | ||||
|
Таксон
|
Количество белков с доменом PF02110
|
|
| Эукариоты | Зеленые растения | 13 |
| Грибы | 57 | |
| Животные | - или 7(смотря, что пониать под термином животные, если считать что альвеолята животные, то их 7) | |
| Остальные эукариоты | 7(альвеолята:инфузории, споровики, динофлагеляты) | |
| Бактерии | 598 | |
| Археи | 28 | |
| № | PFAM ID | Bacillus subtilis |
| 1. | HK | THIM_BACSU |
| 2. | TMP-TENI | THIE_BACSU
TENI_BACSU |
| 3. | Phos_pyr_kin | THID_BACSU O31620_BACSU |

B5CLH5_9FIRM:


Q67T51_SYMTH:


Q1E9G5_COCIM:
