- dna_set.spt-скрипт задающий множества атомов:
- ДНК-белковые контакты
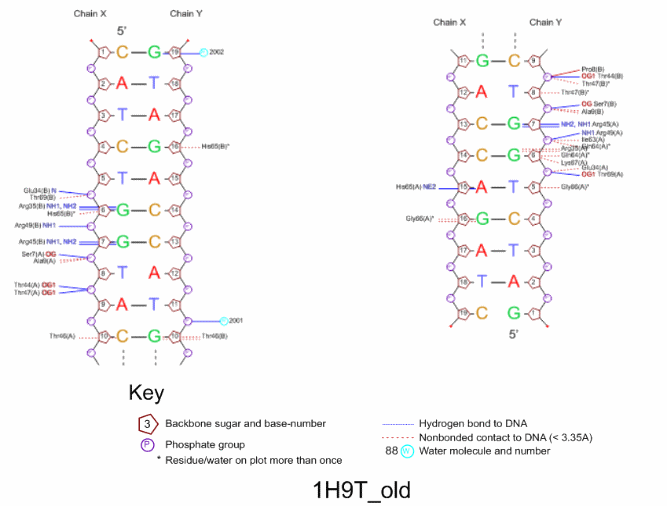
- Популярная схема ДНК-белковых контактов: nucplot.ps - популярная схема ДНК-белковых контактов
- На данной схеме можно обнаружить большое количество аминокислотных остатков с двумя контактами с белком. Например, это Arg35(A), Thr46(B) и др. (В следующем задании будем рассматривать именно Thr46(B))
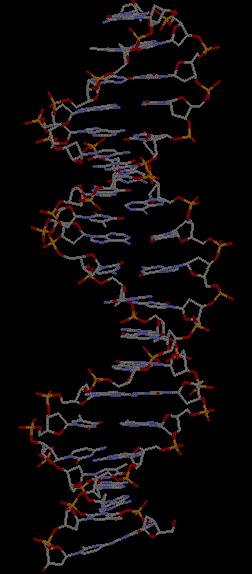
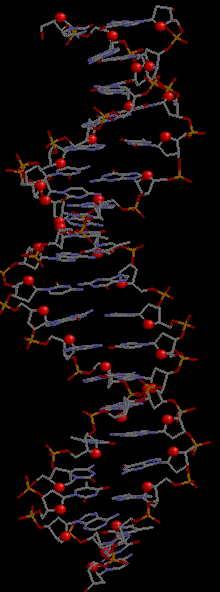
set1 - атомы кислорода в рибозе,
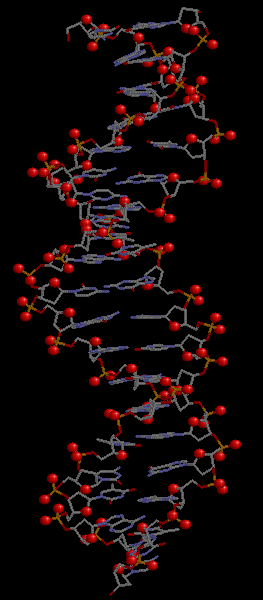
set2 - атомы кислорода в остатке фосфорной кислоты,
set3 - атомы азота в азотистых основаниях
dna_set_visual.spt - последовательно генерирует изображение ДНК с выделенными различными множествами атомов (set1, set2, set3).
 |
 |
 |
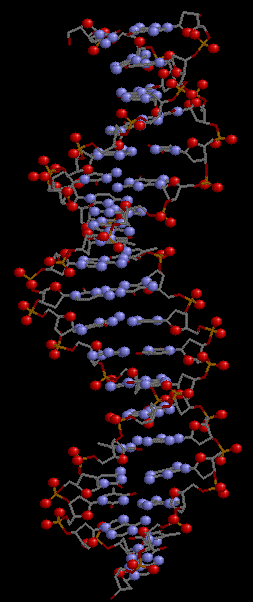 |
| ДНК в остовной моделе | + выделены атомы set1 | + выделены атомы set2 | + выделены атомы set3 |
| Контакты атомов белка с | Полярные | Неполярные | Всего |
| остатками 2'-дезоксирибозы | 8 | 32 | 40 |
| остатками фосфорной кислоты | 19 | 14 | 33 |
| остатками азотистых оснований со стороны большой бороздки | 9 | 9 | 18 |
| остатками азотистых оснований со стороны малой бороздки | 1 | 2 | 3 |
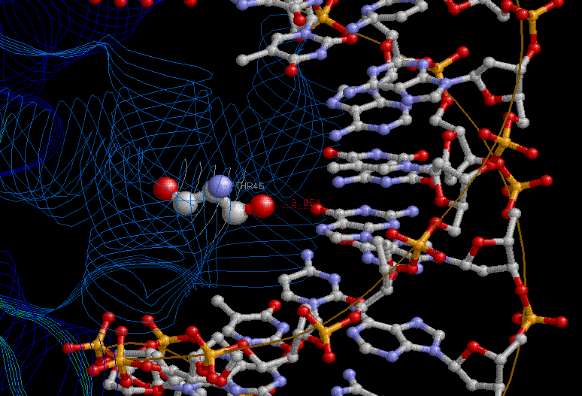
В шарнирной моделе ДНК, белок в ленточной моделе, Thr46 - в шарнирной моделе, с раскраской по атомам.
Я считаю, что наибольший вклад в распознавание последовательности ДНК вносит HIS65:A. Я так считаю, т.к. этот аминокислотный остаток заходит в малую бороздку и его поверхность лучше остальных а.о. взаимодействует с ДНК(см.ниже иллюстрации):
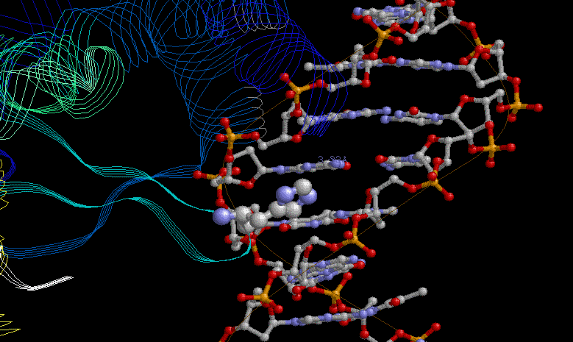
В шарнирной моделе ДНК, белок в ленточной моделе, His65 - в шарнирной моделе, с раскраской по атомам.
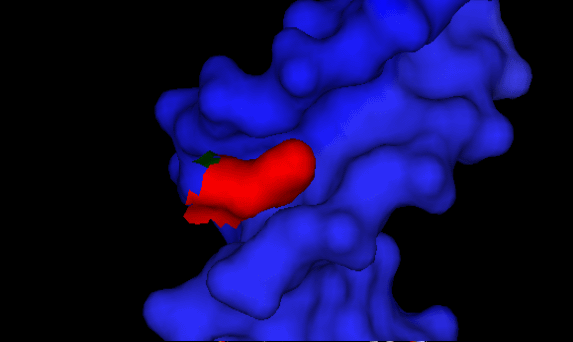
ДНК - поверхность синего цвета, His65 - поверхность красного цвета.
- Предсказание вторичной структуры тРНК путем поиска инвертированных повторов Получение вторичной структуры с помощью программы einverted весьма затруднительно. При длительном подборе параметров наилучшим вариантом стал следующий вызов программы:
- Предcказание вторичной структуры по алгоритму Зукера Для получения предсказания вторичной структуры тРНК по алгоритму Зукера, была использована программа mfold. Сначала была использована команда со стандартными значениями параметров (P=5):
штраф за гэп 12
минимальный вес 10
вес совпадения 5
вес несовпадения -6
mfold SEQ='2cv1_nucl.fasta'Затем я менял параметр P, придавая ему значения 10,15,20. Например:
mfold SEQ='2cv1_nucl.fasta' P=10Но при увеличении значения параметра P появлялись структуры, всё больше отличающиеся от реальной. При стандартном значении была получена структура, наиболее близкая к реальной:
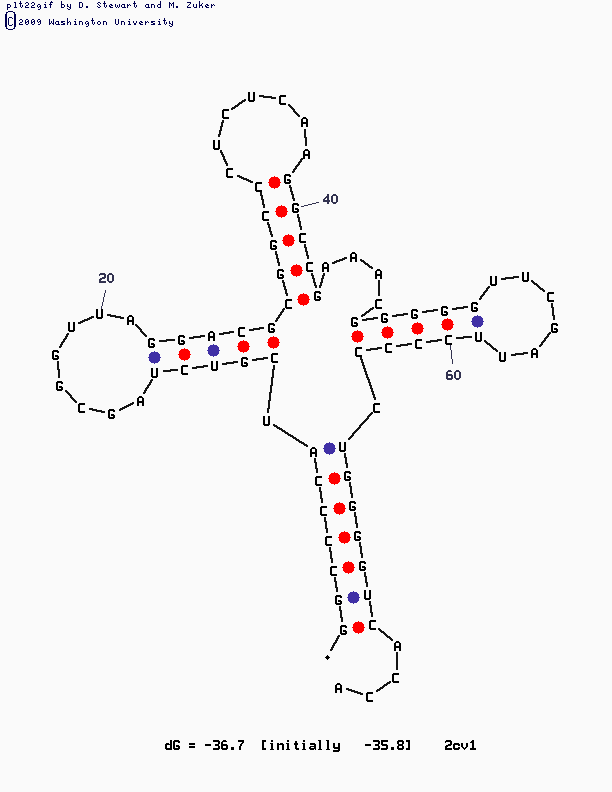 Реальная и предсказанная вторичная структура тРНК из файла 2cv1.pdb
Реальная и предсказанная вторичная структура тРНК из файла 2cv1.pdb
| Участок структуры | Позиции в структуре (по результатам find_pair) |
Результаты предсказания с помощью einverted | Результаты предсказания по алгоритму Зукера |
| Акцепторный стебель | 5' 501-507 3' 5' 566-572 3' Всего 7 пар |
предсказано 5 пар из 7 реальных | предсказано 7 пар из 7 реальных |
| D-стебель | 5' 510-513 3' 5' 522-525 3' Всего 4 пар |
предсказано 0 пар | предсказано 4 пары из 4 реальных и лишняя |
| T-стебель | 5' 549-553 3' 5' 561-565 3' Всего 5 пар |
предсказано 0 пар | предсказано 5 пар, но со сдвигом на 1 нуклеотид |
| Антикодоновый стебель | 5' 526-532 3' 5' 538-544 3' Всего 7 пар |
предсказано 0 пар | предсказано 5 пар из 7 реальных |
| Общее число канонических пар нуклеотидов | 23 | 5 | 22 |
©Анисенко Андрей