EC (классификация ферментов). KEGG (Метаболические пути)
Для выполнения задания был выбран белок PPNK_ECOLI- EC код фермента EC=2.7.1.23 - код белка PPNK_ECOLI, где:
- Определение метаболических путей, в которых учатсвует белок PPNK_ECOLI Локус гена белка PPNK_ECOLI - b2615. Для этого гена были найдены следующие метаболические пути:
- Структурные формулы из базы данных KEGG
- Метаболические пути между L-серином и L-цистеином Выбранная цепочка ферментативных реакций:
- Существование цепочки реакций L-серин → L-цистеин в других организмах
- Сравнение ферментов из далеких организмов
- Найдем с помощью SRS ферменты с ЕС кодом 4.2.1.24 у человека и археи Archaeoglobus fulgidus.
Для того, чтобы провести поиск необходимо снять опцию маски ( Use wildcards ), чтобы при поиске EC белка находились лишь ферменты с EC=4.2.1.24, и не находились ферменты с похожими EC (например, 4.2.1.240 и тд). Для упрощения запроса воспользуемся тем, что для белков приняты короткие имена. ID белков человека заканчиваются на _HUMAN, белки археи Archaeoglobus fulgidus - на _ARCFU. Для отсева идентичных последовательностей воспользуемся ccылкой UNIREF100. В результате, запрос для поиска в БД UniProt выглядит так:([uniprot-ECNumber:4.2.1.24] & ([uniprot-ID:*_HUMAN] | [uniprot-ID:*_ARCFU]))
Получили 4 белка, все относятся к классу дегидрогеназ дельта-амино-левулиновой кислоты. 1 из них относится к археям - HEM2_ARCFU (O28305), а 3 найдены у человека (причем все они представляют один белок, в последовательности это одни участки только с немного различающимися длинами) - выбираем, несмотря на это, белок HEM2_HUMAN (P13716), потому что последовательности остальных входят в последвательность первого(B7Z3I9_HUMAN; Q6ZMU0_HUMAN).
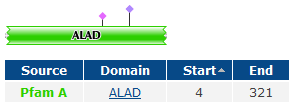
- Сравнение доменной организации (по PFAM) белков HEM2_HUMAN, B7Z3I9_HUMAN, Q6ZMU0_HUMAN и HEM2_ARCFU.
Для этого используем режим SW_InterProMatches (для просмотра находок). - Определение % совпадения последовательностей гомологичных доменов (ALAD) из археи (HEM2_ARCFU) и человека (HEM2_HUMAN).
- Нахождение лучшего ортолога для белков HEM2_ARCFU и HEM2_HUMAN.
Выполняем поиск ортологов с помощью инструментов KEGG.Найдем документ KEGG с описанием гена нужного белка по схеме, описанной в упр.2. На найденной страничке выбираем кнопку "Ortholog", а в открывшемся окне - опцию Best-best (bidirectional best hit),указываем нужную группу организмов и выбираем кнопку "GO".
Для белка HEM2_HUMAN название гена ALAD; для HEM2_ARCFU - hemB. Соответсвующие id в KEGG hsa:210 и afu:AF1974.
Ген ортолог для HEM2_HUMAN среди архей:
Ген: mka:MK0198 Организм: Methanopyrus kandleri Процент идентичности: 0.469 Перекрытие последовательностей: 309
Ген ортолог для HEM2_ARCFU среди эукариот:
Ген: pti:PHATRDRAFT_50723 Организм: Phaeodactylum tricornutum Процент идентичности: 0.489 Перекрытие последовательностей: 321
2 - семесйство трансфераз (transferase)
2.7 - трансферазы фосфор-содержащих групп (Transferring phosphorus-containing groups)
2.7.1 - фосфотрансеразы с гидроксильной группой в качестве акцептора (Phosphotransferases with an alcohol group as acceptor)
2.7.1.23 -НАД+ киназа (NAD+ kinase )
Данный фермент осуществляет следующую реакцию:
ATP + NAD+ = ADP + NADP+
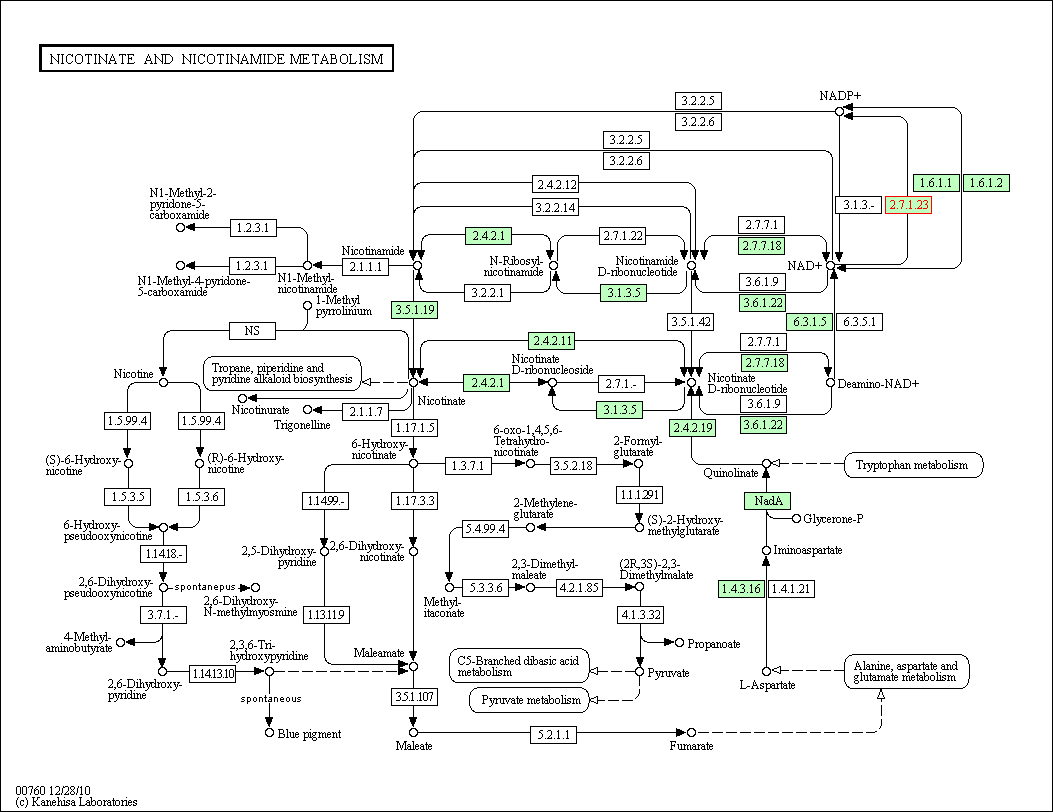
Ниже приведен путь синтеза НАДФ+, красным кругом выделена реакция, осуществляемая ферментом PPNK_ECOLI

eco00760 - метаболизм никотината и никотинамида (Nicotinate and nicotinamide metabolism)
eco01100 - метаболические пути (Metabolic pathways)
|
Название соединения:
L-Серин (L-Serine) L-2-Амино-3-гидроксипропионовая кислота(L-2-Amino-3-hydroxypropionic acid) L-3-Гидроксиаланин(L-3-Hydroxy-alanine) Идентификатор: C00065 Структурная формула: 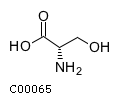
|
Название соединения:
L-Цистеин(L-Cysteine) L-2-Амино-3-меркаптопропионовая кислота(L-2-Amino-3-mercaptopropionic acid) Идентификатор: C00097 Структурная формула: 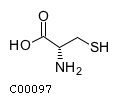
|
путь:метаболизм серина, треонина и глицина
цепочка:L-серин → L-цистеин
К сожалению или к счасть( это уже для кого как) не было найдено ни одного интермедиата. С картой можно осзнакомиться на ссайте в файле - map.png
| Организм | Возможна ли цепочка реакций | Обоснование |
| Escherichia coli K-12 MG1655 | да | есть фермент, неоходимый для осуществления цепочки EC 4.2.1.22 |
| Archaeoglobus fulgidus | да | есть фермент, неоходимый для осуществления цепочки EC 4.2.1.22 |
| Arabidopsis thaliana (thale cress) | да | есть фермент, неоходимый для осуществления цепочки EC 4.2.1.22 |
| Homo sapiens | да | есть фермент, неоходимый для осуществления цепочки EC 4.2.1.22 |
B7Z3I9_HUMAN - домен хоть и выделен, но не отмечен.
Q6ZMU0_HUMAN - менее похож на белок из Archaeoglobus fulgidus (немного длиннее и у него есть участок low_complexity (низкого сродства)).
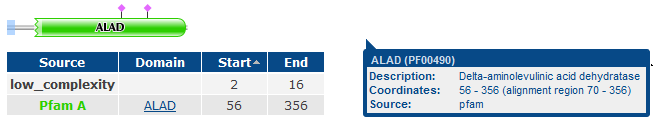
HEM2_HUMAN (P13716)
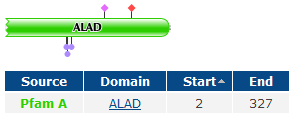
HEM2_ARCFU (O28305)
Воспользуемся программой needle для создания выравнивания. Получили процент сходства 61.4%.

Белок в организме человека соответсвует белку в археи с относительно большим процентом идентичности, белки выполняют одинаковые функции в столь разных и далеких организмах. Аналогичные выводы можно сделать, во время рассмотрения белка HEM2_ARCFU и его ортолога.
©Анисенко Андрей