Трансмембранные белки
-
Для выполнения данного задания были выбраны следующие альфа-спиральные трансмембранные белки:
- Трансмембранные участки белка P06795 (MDR1_MOUSE) Помимо белков которые предлагались в задании в выравнивание были добавлены гомологи данного белка: Q8R427 (Q8R427_RAT), P43245 (MDR1_RAT).
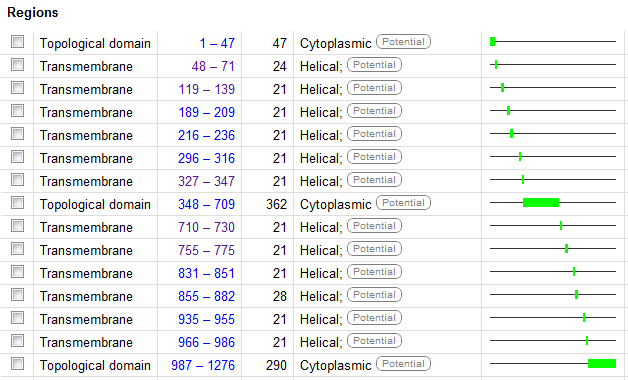
- UNIPROT В ниже приведенной таблице представлены координаты трансмемьанных и цитоплазматических учасктов белка MDR1_MOUSE:
- TMHMM а следующие данные были получены при помощи прогаммы предсказания трансмембранных спиралей по последовательности (сервис TMHMM )
- PDBTM В базе данных PDBTM была найдена запись 3G5U. Ниже приведена иллюстрация расположеня трансмембранных участков данног белка:
- Cравнение предсказания при помощи программы TMHMM c предсказанием по гомологу Стоит отметить, что данная программа (TMHMM) по всей видимости считает, что все альфа спирали погруженные в билипидный слой перпендикулярны ему, т.к. длины всех участков погруженных в мембрану равны 22, т.е. видимо, данная программа ищет участки последовательности наиболее гидрофобные и при этом соответствующие длинне 22 а.о. на один трансмембранный элемент.
| альфа-спиральные трансмембранные белки | Бета-баррели | ||
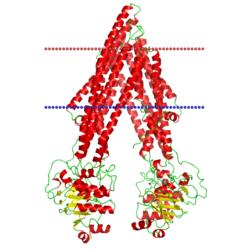
| археородпсин-1
(Halobacterium sp., PDB код 1uaz) |
 |
деацетилаза липида А PagL
(Pseudomonas aeruginosa, PDB код 2erv) |
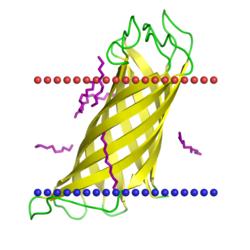
|
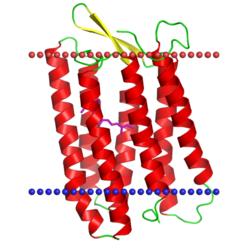
| сенсорный белок kdpD
(Escherichia coli, PDB код 2ksf) | 
| автотранспортер NalP
(Neisseria meningitidis, PDB код 1uyn) |
|
| фумарат редуктаза
(Wolinella succinogenes, PDB код 2bs2) | 
| фосфолипаза А наружней мембраны
(Escherichia coli, PDB код 1qd5) |
|
| М2 канал вируса гриппа В
(Influenza virus, PDB код 2kix) | 
| альфа гемолизин
(Staphylococcus aureus, PDB код 7ahl) |
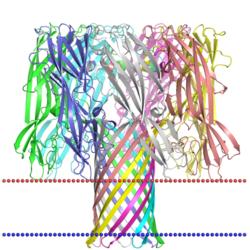
|
| калиевый канал KcsA
(Streptomyces lividans, PDB код 3eff, полноразмерный белок) | 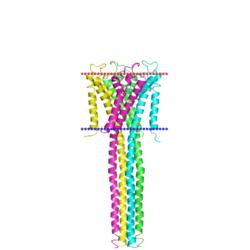
| VDAC-1 канал
(Homo sapiens, PDB код 2k4t) |
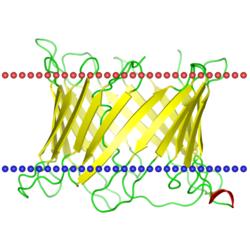
|
| PDB код | Число трансмембранных цепей | Тип
(спираль, баррель) |
Число трансмембранных участков в цепи | Число остатков в одном трансмембранном участке
(среднее, минимальное, максимальное) |
(*) Толщина мембраны в ангстремах
(расстояние между атомами на границах трансмембранных участков |
| 1uaz | 1 (A) | спираль | 7 | C=21; min=18; max=23 | 31,8±1,3Å |
| 2ksf | 1 (A) | спираль | 4 | C=16; min=9; max=21 | 26,2±4Å |
| 2bs2 | 2 (C, F) | спираль | 10 | C=22; min=20; max=24 | 31,2±1,2Å |
| 2kix | 4 (A, B, C, D) | спираль | 4 | C=24; min=23; max=24 | 31,8±1,2Å |
| 3eff | 4 (K, L, M, N) | спираль | 12 | C=20; min=12; max=24 | 31,8±1,3Å |
| 2erv | 1 (A) | баррель | 8 | C=11; min=7; max=12 | 24,7±1,8Å |
| 1uyn | 1 (X) | баррель | 12 | C=9 min=8; max=11 | 25,3±1,3Å |
| 1qd5 | 1 (A) | баррель | 12 | C=9; min=5; max=12 | 24,6±1,2Å |
| 7ahl | 7 (A, B, C, D, E, F, G) | баррель | 14 | min=7; max=8, других участков нет | 23,5±0,9Å |
| 2k4t | 1 (A) | баррель | 19 | min=6; max=8, примерно с одинаковой вероятностью встречаются участки длины 7 и 8 | 22,0±1,5Å |
Из таблицы видно, что толщина мембраны для альфа-спиральных трансмембранных белков как правило равна 31,8Å, а в случае бета-баррелей - 24,0Å (И в связи с этим количество аминокислотных остатков, приходящихся на один трансмембранный участок, для бета-баррелей значительно ниже, чем в случае альфа-спиральных белков.
sp_P06795_MDR1_MOUSE TMHMM2.0 inside 1 47 sp_P06795_MDR1_MOUSE TMHMM2.0 TMhelix 48 70 sp_P06795_MDR1_MOUSE TMHMM2.0 outside 71 114 sp_P06795_MDR1_MOUSE TMHMM2.0 TMhelix 115 137 sp_P06795_MDR1_MOUSE TMHMM2.0 inside 138 188 sp_P06795_MDR1_MOUSE TMHMM2.0 TMhelix 189 211 sp_P06795_MDR1_MOUSE TMHMM2.0 outside 212 215 sp_P06795_MDR1_MOUSE TMHMM2.0 TMhelix 216 238 sp_P06795_MDR1_MOUSE TMHMM2.0 inside 239 293 sp_P06795_MDR1_MOUSE TMHMM2.0 TMhelix 294 316 sp_P06795_MDR1_MOUSE TMHMM2.0 outside 317 707 sp_P06795_MDR1_MOUSE TMHMM2.0 TMhelix 708 730 sp_P06795_MDR1_MOUSE TMHMM2.0 inside 731 750 sp_P06795_MDR1_MOUSE TMHMM2.0 TMhelix 751 773 sp_P06795_MDR1_MOUSE TMHMM2.0 outside 774 849 sp_P06795_MDR1_MOUSE TMHMM2.0 TMhelix 850 872 sp_P06795_MDR1_MOUSE TMHMM2.0 inside 873 934 sp_P06795_MDR1_MOUSE TMHMM2.0 TMhelix 935 957 sp_P06795_MDR1_MOUSE TMHMM2.0 outside 958 971 sp_P06795_MDR1_MOUSE TMHMM2.0 TMhelix 972 994 sp_P06795_MDR1_MOUSE TMHMM2.0 inside 995 1276
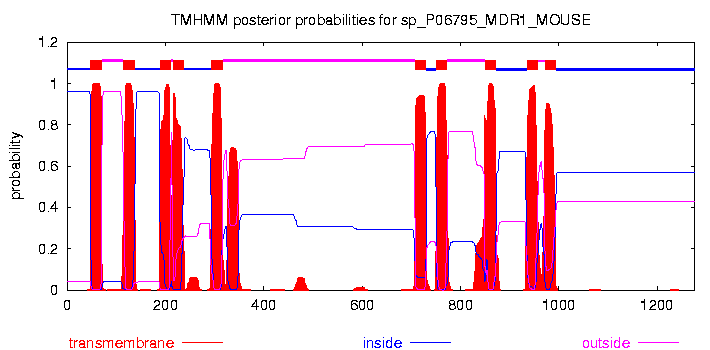
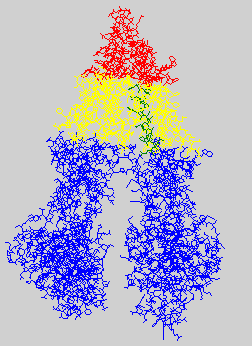
и соответствие а.о. участкам:
1-32 неизвестный 33-44 цитоплазматический 45-68 тм 69-113 внешнемембранный 114-132 тм 133-183 циоплазматический 184-203 тм 204-214 внешнемембранный 215-234 тм 235-290 цитоплазматический 291-310 тм 311-331 внешнемембранный 332-349 тм 350-626 цитоплазматический 627-683 неизвестно 684-707 цитоплазматический 708-728 тм 729-752 внешнемембранный 753-773 тм 774-828 цитоплазматический 829-847 тм 848-851 внешнемембранный 852-872 тм 873-932 цитоплазматический 933-953 тм 954-972 внешнемембранный 973-990 тм 991-1271 цитоплазматический 1272-1284 неизвестноВ данном выравнивании (данный белок и белок гомолог, для которого существует рентгеноструктурный анализ) Gene3.msf зеленым цветом покрашены а.о. соответсвующие трансмембарнным спиралям.
в данном выравнивании all.msf представлены выровненные последовательности всех белков и плюс включены строки с описанием структуры данных белков. Желтым цветом отмечены а.о., которые соответсвуют трансмембранным элементам и совпали во всех случаях предсказания.
| Число а.к. остатков | |
| Всего а.к. остатков в последовательности | 1276 |
| Остатки, предсказанные TMHMM как локализованные в мембране (всего) | 220 |
| Правильно предсказали - совпадают с 3D предсказанием (true positives, TP) | 165 |
| Предсказали не то, что нужно (а.о. предсказаны как мембранные, а по данным 3D таковыми не являются, false positives, FP) | 55 |
| Правильно не предсказали ( не предсказаны, и по данным 3D не находятся в мембране, true negatives, TN) | 979 |
| Не предсказали то, что нужно (остатки по данным 3D находятся в мембране, false negatives, FN) | 77 |
| Чувствительность (sensivity) = TP / (TP+FN) | 0,681818182 |
| Специфичность (specificity) = TN / (TN+FP) | 0,946808511 |
| Точность(precision) = TP /(TP+FP) | 0,75 |
| Сверхпредсказание = FP/ (FP+TP) | 0,25 |
| Недопредсказание = FN / (TN+FN) | 0,072916667 |