Пакет Pftools
Для дальнейшей работы было выбрано выравнивание белков из Firmicutes: RS3_BACAN, RS3_BACSU, RS3_CLOB1, RS3_CLOTE, RS3_ENTFA, RS3_FINM2, RS3_GEOKA, RS3_LACAC, RS3_LACDA, RS3_LACLM, RS3_LISMO, RS3_PEDPA, RS3_STAA1, RS3_STAES, RS3_STRP1, RS3_STRPN, RS3_THETN, полученное при выполнении предыдущих заданий.- Подготовка файла в формате msf
- PFW Программа pfw позволяет вычислять веса индивидуальных последовательностей во множественном выравнивании, используя метод Сиббала и Аргоза. используя запрос pfw part.noreturn > part_pfw.msf. Ниже приведен участок файла до применения данной команды:
- PFMAKE Программа pfmake создает prosite-овский профиль, используя в качестве входного файла - взвешенное выравнивание. При помощи запроса pfmake part_pfw.msf /usr/share/pftools23/blosum62.cmp > part_pfmake.prf был получен файл part_pfmake.prf.
- Подготовка последовательностей для поиска При помощи запроса seqret sw-org:bacteria bacteria.fasta был получен файл, содержащий все последовательности из бактерий.
- Нормировка профиля На первом этапе был сгенерирован банк, того же размера, что и bacteria.fasta.
- Поиск по профилю При помощи запроса pfsearch -C1.15 -f part_pfmake.prf bacteria.fasta > part.pfsearch было найдено 2366 последовательности part.pfsearch
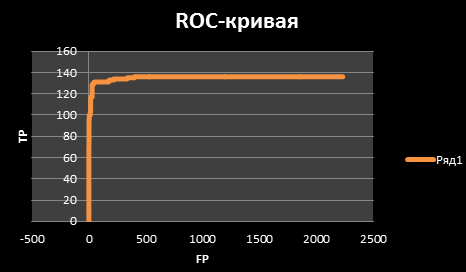
- Оценка профиля Число верных находок ("True positive hits", TP)=136 Число ложных находок ("False positive hits", FP)=2230 Число ненайденных белков подсемейства (ложноотрицательных результатов, "False negatives", FN)=0 Чувствительность TP/(TP+FN)=1 Селективность TP/(TP+FP)=0,057
Т.к. пакет Pftools не работает с файлами, имеющими конец строки, принятый в Windows, я поменял признаки конца строки на UNIХ-овые используя команду noreturn -in part.msf -out part.noreturn.
Name: RS3_THETN Len: 246 Check: 4841 Weight: 1.00 Name: RS3_CLOB1 Len: 246 Check: 6023 Weight: 1.00 Name: RS3_CLOTE Len: 246 Check: 6185 Weight: 1.00 Name: RS3_FINM2 Len: 246 Check: 1233 Weight: 1.00 Name: RS3_LACAC Len: 246 Check: 6704 Weight: 1.00 Name: RS3_LACDA Len: 246 Check: 3672 Weight: 1.00 Name: RS3_STAA1 Len: 246 Check: 1549 Weight: 1.00 Name: RS3_STAES Len: 246 Check: 1525 Weight: 1.00 Name: RS3_PEDPA Len: 246 Check: 2217 Weight: 1.00 Name: RS3_ENTFA Len: 246 Check: 1518 Weight: 1.00 Name: RS3_LACLM Len: 246 Check: 2337 Weight: 1.00 Name: RS3_STRP1 Len: 246 Check: 1733 Weight: 1.00 Name: RS3_STRPN Len: 246 Check: 556 Weight: 1.00 Name: RS3_BACAN Len: 246 Check: 5134 Weight: 1.00 Name: RS3_LISMO Len: 246 Check: 5014 Weight: 1.00 Name: RS3_BACSU Len: 246 Check: 6904 Weight: 1.00 Name: RS3_GEOKA Len: 246 Check: 3821 Weight: 1.00
Как видим все последовательности имеют вес 1.00.
И участок файла после работы данной программы:
Name: RS3_BACAN Len: 24 Check: 2649 Weight: 0.0270 Name: RS3_BACSU Len: 24 Check: 2532 Weight: 0.0179 Name: RS3_CLOB1 Len: 24 Check: 2412 Weight: 0.1014 Name: RS3_CLOTE Len: 24 Check: 2806 Weight: 0.1152 Name: RS3_ENTFA Len: 24 Check: 2522 Weight: 0.0142 Name: RS3_FINM2 Len: 24 Check: 3155 Weight: 0.1509 Name: RS3_GEOKA Len: 24 Check: 2649 Weight: 0.0270 Name: RS3_LACAC Len: 24 Check: 2431 Weight: 0.0365 Name: RS3_LACDA Len: 24 Check: 2447 Weight: 0.0693 Name: RS3_LACLM Len: 24 Check: 2908 Weight: 0.1004 Name: RS3_LISMO Len: 24 Check: 2532 Weight: 0.0179 Name: RS3_PEDPA Len: 24 Check: 2522 Weight: 0.0142 Name: RS3_STAA1 Len: 24 Check: 2335 Weight: 0.0515 Name: RS3_STAES Len: 24 Check: 2335 Weight: 0.0515 Name: RS3_STRP1 Len: 24 Check: 2674 Weight: 0.0590 Name: RS3_STRPN Len: 24 Check: 2714 Weight: 0.0546 Name: RS3_THETN Len: 24 Check: 2374 Weight: 0.1015
Как видим программа переписала веса последовательностям. part_pfw.msf
Далее был проведен "фальшивый поиск", для получения веса профиля на случайных последовательностях - pfsearch -C0.0 -f part_pfmake.prf shuffled.fasta | sort -n > scores.txt
А затем и сама нормировка - pfscale scores.txt part_pfmake.prf > scaled.prf
scaled.prf
При этом изменились поля: MA /NORMALIZATION, MA /CUT_OFF.
Но к сожалению при выполнении следующего пункта при использовании нормированного профиля возникала ошибка поэтому в дальнешем использовал ненормированный профиль.
Ниже приведена ROC-кривая:
©Анисенко Андрей