Алгоритмы реконтрукции филогении
- Укоренение в среднюю точку Мы можем укоренить дерево, если в нем отсутсвует корень и указаны длины ветвей (поэтому методы UPGMA и максимальной экономии не пододят. Первый из них выдает уже укорененное дерево, а второй - не выдает длины ветвей) . Данным требованиям удовлетворяют деревья, полученные при помощи программы fneighbor, реализующей алгоритм Neighbor-Joining и UPGMA.
- Использование внешней группы В последовательность белков группы RPOZ был добавлен данный белок из E.coli. Данная операция была осуществлена посредством следующего запроса seqret sw:rpoz_ecoli stdout >> sequences.fasta. Затем при помощи программы MUSCLE построено выравнивание новых последовательностей. Далее из названий последовательностей в выравнивании удалил все, за исключение мнемоники организма. Конечный вариант выравнивания подал на вход программе fprotpars. После выполнения выше описанных операций при помощи программы RETREE получил дерево с аут-группой:
- Бутстрэп При помощи программы fconsense (с предварительным созданием бутстрэпов и создание деревьев) было получено дерево:
((((CLOTE:4.89902,LACDA:4.36664):5.46257,THETN:2.44099):2.63350, PEDPA:-0.57832):9.04740,((FINM2:2.89136,STAA1:0.09228):0.52254,STAES:-0.68757):6.58025,BACAN:-5.40084); - скобочная формула дерева, полученного данной программой.
была запущена программа retree:
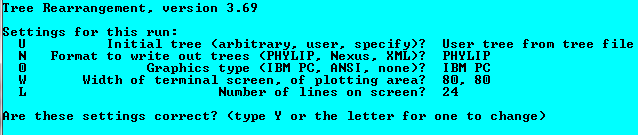
На вопрос программы ответил Y, после чего ввел M. Было получено дерево:
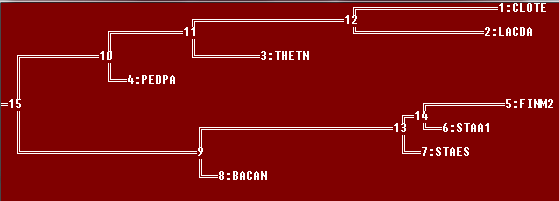
Укоренение произошло в ветвь {CLOTE,LACDA,THETN,PEDPA} против {FINM2,STAA1,STAES,BACAN}. В правильном дереве было укоренение в ветвь {CLOTE, FINM2, THETN} против {PEDPA, LACDA, STAES, STAA1, BACAN}
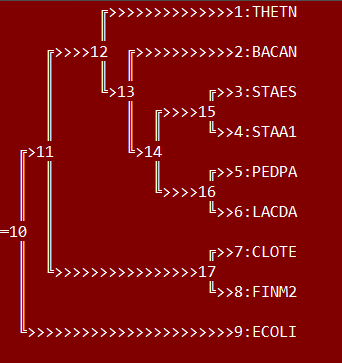
как видно, дерево укоренилось (если отбросить внешнюю группу) по ветви {CLOTE, FINM2} против {THETN, PEDPA, LACDA, STAES, STAA1, BACAN}. Данное укоренение больше похоже на правильное укоренение, ветви отличаются только по элементу THETN.
+-----------------------------------------------THETN
|
| +-------STAA1
| +--99.0-|
| +--27.7-| +-------STAES
| | |
| +--31.3-| +---------------BACAN
| | |
| | | +-------LACDA
| +--35.7-| +----------55.4-|
| | | +-------PEDPA
+-------| |
| +-------------------------------FINM2
|
+---------------------------------------CLOTE
Скобочная формула дерева: (((((STAA1,STAES),BACAN),(LACDA,PEDPA),FINM2),CLOTE),THETN);.В дереве, созданном программой fprotpars без применения bootstrap - 1 общая ветвь с правильным деревом:
{THETN, PEDPA, LACDA, BACAN, CLOTE, FINM2} vs {STAES, STAA1}
В дереве, созданном программой fprotpars c применения bootstrap - 4 общие ветви с правильным деревом:
{PEDPA, LACDA, STAES, STAA1, BACAN} vs {CLOTE, FINM2, THETN}
{THETN, PEDPA, LACDA, BACAN, CLOTE, FINM2} vs {STAES, STAA1}
{PEDPA, LACDA} vs {STAES, STAA1, BACAN, CLOTE,FINM2 THETN}
{THETN, PEDPA, LACDA, CLOTE, FINM2} vs {STAES, STAA1, BACAN}.
Таким образом, после применения bootstrap'а качество реконструкции филогении улучшилось.
Не получила поддержку тривиальная ветвь: {CLOTE,FINM2} vs {THETN, PEDPA, LACDA, STAES, STAA1, BACAN}
©Анисенко Андрей