Предсказание парных выравниваний
1. выравнивание последних двух последовательностей из http://kodomo.fbb.msu.ru/FBB/year_13/term2/aln2/aln_10/aln_10.fasta.
Выравнивание было загружено в Jalview с помощью ручного ввода текста с двумя последними последовательностями (представлено на Рисунке 1). Скачать проект можно ЗДЕСЬ.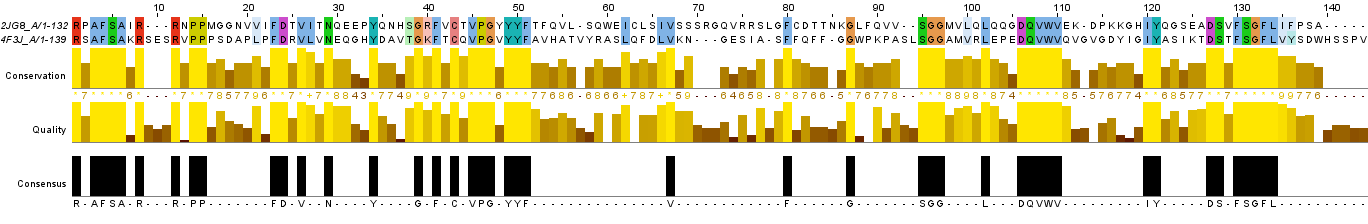
Рис.1. Выравнивание.
2. Глобальное выравнивание последних двух последовательностей в формате fasta.
Выранивание проводилось следующим образом: с помошью программы needle в putty два файла с последовательностями компонуются в один - выравнивание. Две нужных последовательности выглядят так:>2JG8_A/1-132 RPAFSAIRRNPPMGGNVVIFDTVITNQEEPYQNHSGRFVCTVPGYYYFTFQVLSQWEICLSIVSSSRGQVRRSLGFCDTTNKGLFQV VSGGMVLQLQQGDQVWVEKDPKKGHIYQGSEADSVFSGFLIFPSA
>4F3J_A/1-139 RSAFSAKRSESRVPPPSDAPLPFDRVLVNEQGHYDAVTGKFTCQVPGVYYFAVHATVYRASLQFDLVKN GESIASFFQFFGGWPKPASLSGGAMVRLEPEDQVWVQVGVGDYIGIYASIKTDSTFSGFLVYSDWHSSPV
Были сделаны два файла с этими последовательностями: one.fasta и two.fasta. На выходе был получен ФАЙЛ. Открыв его в Jalview получаем выравнивание, показаное на Рисунке 2.
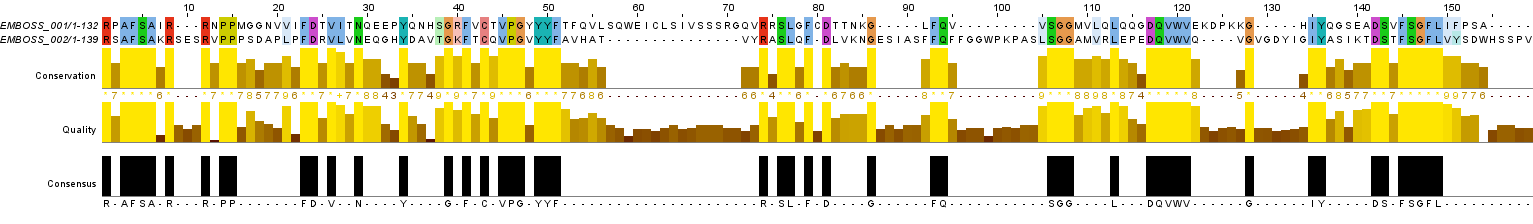
Рис.2. Глобальное выравнивание.
3. Выравнивание последних двух последовательностей в формате fasta.
Выранивание проводилось аналогично пункту 2, , с теми же файлами, но с помошью программы water. На выходе был получен ФАЙЛ. Открыв его в Jalview получаем выравнивание, показаное на Рисунке 3.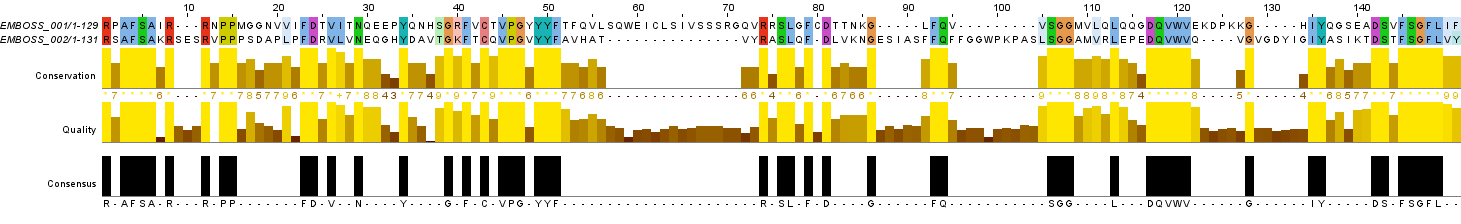
Рис.3. Выравнивание. Water
4. Получение выравнивания двух заведомо негомологичных последовательностей
Выравнивание между моим белком glr4197 и белком
одного из студентов superoxide dismutase [Thermosynechococcus elongatus BP-1] . Были
использованны программы needle и water. Файлы соответствующих выравниваний:
needle.fasta
water.fasta;
и в форматах
по умолчанию:
needle
water

Рис.4.Выравнивание негомологичных последовательностей, полученное при помощи needle.
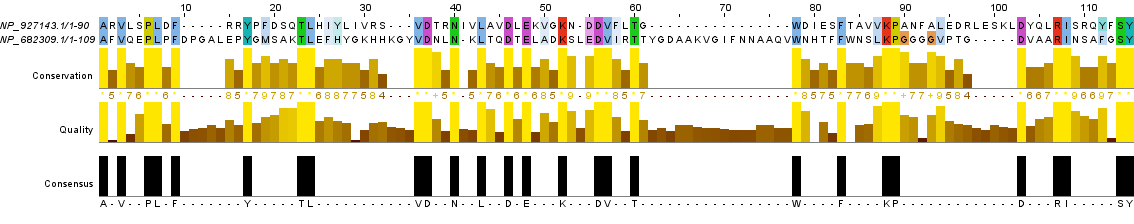
Рис.5.Выравнивание негомологичных последовательностей, полученное при помощи water.
5. Tаблица параметров выравниваний 1-4
| Длина выравнивания | Число и % совпадений | Число и % сходных остатков | Число и % гэпов (в обеих цепях) | ||
| 1 | начальное выравнивание | 144 | 44 = 30.5% | 62(+-3) = 40.9% - 45.1% | 17 = 5.9% |
| 2 | глобальное выравнивание (needle) | 159 | 51 = 32% | 77 = 48.5% | 47 = 14.8% |
| 3 | локальное выравнивание (water) | 151 | 51 = 33.8% | 72 = 47.7% | 42 = 13.9% |
| 4 | выравнивание негомологичных белков(глобальное) | 240 | 30 = 12.5% | 60(+-3) = 23.75%-26.25% | 151 = 31.4% |
| 5 | выравнивание негомологичных белков(локальное) | 127 | 27 = 21.26% | 45(+-3) = 33.0% - 37.8% | 50 = 19.7% |
6. Участок различия в двух выравниваниях
Для поиска отличного участка два выравнивания(исходное и глобальное) были совмещенны, для лучшего совмещения были добаленны гэпы.Проект совмещенного выравнивания можно скачать здесь. На рисунке видно отличный участок - это значительная неокрашенная область с 57го по 107ой аминоксислотный остаток.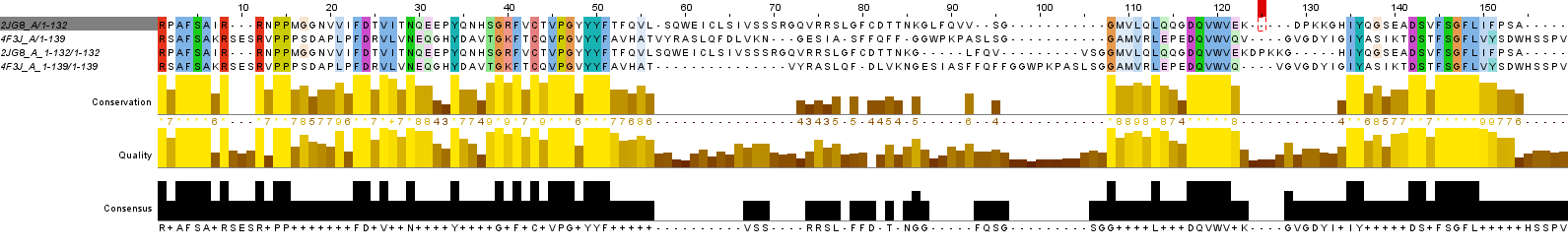
Рис.5.Сравнение выравниваний.
7. Проверка правильности выравниваний (из первых пунктов)
При помощи sup_check был получен скрипт, позволяющий оценить взаимное пространственное расположение аминокислотных остатков в двух последовательностях друг относительно друга. Проект выравнивания(исходного) можно скачать здесь. На рисунке ниже представленно именно это выравнивание. Здесь обнаружилось 6 ошибок первого рода(я приняла участки за структурносходные, но на самом деле это оказалось не так). Ошибок второго рода(т.е. я не поверила в структурную схожесть остатков, но при взгляде в RasMol обнаружилось обратное) значительно больше - 17._pr13.png)
Рис.6.Проверка выравнивания двух последовательностей из исходного выравнивания.
Для сравнения глобального выравнивания пришлось немного изменить файл sup_check.ini(поменять название последовательностей, с которыми программа работает и изменить название выдаваемого скрипта). Проект выравнивания можно скачать здесь.Здесь было обнаруженно 3 ошибки первого рода и столько же ошибок второго рода. Оба показателя лучше чем при проверке исходного выравнивания, таким образом, глобальное выравнивание оказалось лучше._pr10.png)
Рис.7.Проверка глобального выравниванияэтих же двух последовательностей.
| © Амосова Алена. 2014 год | Дата последнего изменения: 13 марта 2014. |