Выравнивание и гомология.
Для выполнения заданий первого практикума по выравниваниям, я выбрала файл aln1_5.fa. Выравнивание последовательностей – это метод, позволяющий изобразить их предполагаемое сходство. Для этого используется программа JalView. На Рисунке 1 изображено выравнивание полностью. Тип раскраски выравнивания – Clustalx, консервативность - 70%.
Рис.1. Полное выравнивание.
На Рисунке 2 и Рисунке 3 два участка, на которых для каждой колонки можно ожидать гомологию между остатками из всех последовательностей.

Рис.2. Участок с предполагаемой гомологией
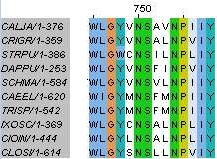
Рис.3. Другой участок с предполагаемой гомологией
Далее мне нужно было найти участок, на котором остатки двух последовательностей предположительно гомологичны, а остатки всех остальных – нет. Для этого я построила дерево последовательности, которое показано на Рисунке 4.
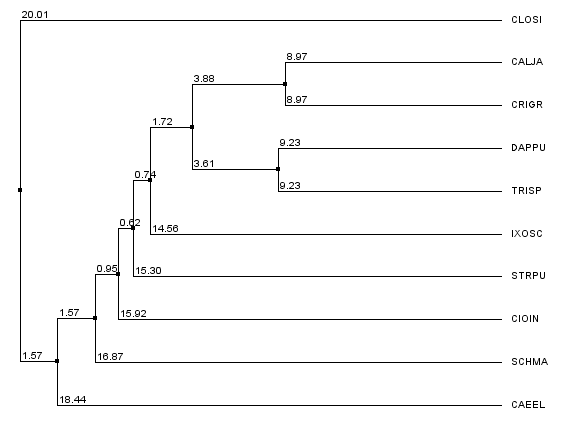
Рис.4. Дерево построенное в программе JalWiew
Последовательности CALJA и CRIGR достаточно близки и их выравнивание явно содержит довольно много похожих участков. На Рисунке 5 показан участок, где эти две последовательности, отдельно окрашенные в цвет Clustalx с консервативностью 70%, гомологичны между собой, и не гомологичны другим последовательностям (столбцы 100-108).

Рис.5. Две наиболее гомологичные последовательности
Для первого блока (см. Рисунок 2), состоящей из 21 колонки, характерно наличие восьми абсолютно функционально консервативных позиций (что составляет 38%) и 11 позиций функционально консервативных на 70% (это 52% от всех). На Рисунке 6 представлен участок между двумя вертикальными блоками (выделен красным – 29 столбцов). Под выравниванием есть строка Conservation, по которой можно определить, что гэпов 10 (на гэпы указывают прочерки). В процентах это 34%.
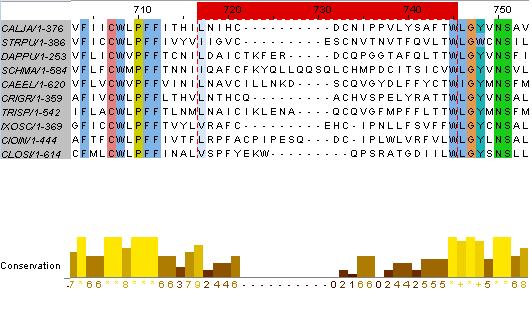
Рис.6. Две наиболее гомологичные последовательности
2.Ручное выравнивание
С помощью команды «File > Add Sequence > from URL…» к исходным последовательностям была добавлена ещё одна (seqdump1_5). Для нового выравнивания была установлена окраска, описанная выше, с тем же процентом консервативности. Для поиска одинаковых участков можно воспользоваться поиском «File > Add Sequence > from URL», но, случайно передвигая добавленную последовательность, мне удалось найти участок, гомологичный изображённому на Рисунке 3. Результат можно видеть на Рисунке 7.
Рис.7. Ручное выравнивание
Выравнивание полностью можно посмотреть на Рисунке 8.

Рис.8. Полное выравнивание вручную.
Для поиска консенсусной последовательности (это последовательность из самых встречаемых букв из каждого столбца) я воспользовалась программой cons на сервере http://emboss.bioinformatics.nl/ . Полученный на выходе файл содержит последо вательность:
>consensus GNVLVxvAVxRxKxLQtxtNYfIVSLAVADLLVAIIVMPxYAVYxxLExVNgKxWxLGxV ICDIYVAlDVLcCTASILNLcaISIDRYLAVTxPVxYxxxxRxRNxKRVxLMIaaVWVLS xVISxPLLfGLNxxxxxxNxxxxxxxxxxxxxxxxxxxxxsxxxxCxIxxNxdFVIYSSI xSFYIPxiIMLxIYxRIYLxLRxxxxxxxRRxRRxxxxxxxxxxxxxxxxxxxxxxxxxx xxxxxxxxxxxxksxRsxxxxxxmxxxxxxxxxxxxxxxxxxcxxxxxxxxxxxthxeDx xxxxxxxxxxxxxxxxxxxxxnxxxxxxxxxxxxxxxxxxxxxxxxxxxxxxxxxxxxxs xxxxxxsxsLnxxxsPxxxxxExkkxxxxxpxxxxxxxxxxxxxxxxxxxxxxxxxxxxx xxxxxxxxxxxSsxnLSLxEpsxxxxxxxxxxxxxxxxxxxxxxxxxxsxxTxDSxxxxs xxxxxxxxxxxxxxxxxxxxxxxxxxxxxxxxxxxxxxxxxxxxxxxxxxxxxxxxxxxx xNxxxxxxxxxxxxxxxxxxxxxxxxxxxxxxxxxxxxxxxxxxxxxxxxxxxxxsPRxx kxxxxxxxxxxEvQxmxnxxxRxSxxSxxxxxxxxxxxxxxxxxxxxxxxxxxxxxxxxx xxxxxxxxxxxxxLrRRxxxKISxkxxxREKKATKTLAIVLGVFLiCWLPFFIvNILNax CxxxxxxxxxxxCxVxxxLFxIxTWLGYVNSxLNPIIY
Его можно скачать ЗДЕСЬ .
LOGO последовательности – это последовательность, где каждая буква имеет разный размер - в зависимости от встречаемости в столбце. Лого блока, изображенного на Рисунке 7, можно посмотреть на Рисунке 9.

Рис.9. LOGO-последовательность
3.Выравнивание" заведомо негомологичных белков
Я выбрала случайным образом 5 белков, с которыми работали мои однокурсники, и выполнила выравнивание. Как и ожидалось, гомологичных участков почти не было. Выравнивание представлено на Рисунке 10.
Рис.10. Полное выравнивание негомологичных белков.
С помощью дерева (см. Рисунок 11) я смогла определить два самых близких организма и окрасила их в цвет той же консервативности отдельно. На Рисунке 12 выравнивание представлено полностью.
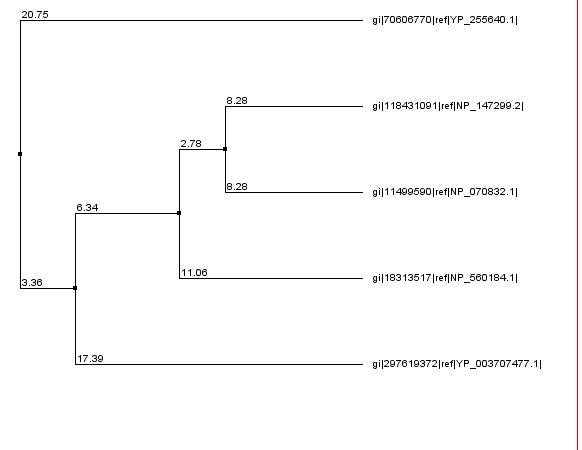
Рис.11. Дерево негмологичных белков.

Рис.12. Полное выравнивание негомологичных белков.
| © Амосова Алена. 2014 год | Дата последнего изменения: 13 марта 2014. |