Предсказание вторичной структуры заданной тРНК
С помощью программы einverted, были предсказаны комплементарные взаимодействия в тРНК из предыдущего задания. Если запустить программу со стандартными параметрами, она ничего не запишет в выходной файл, но изменив параметры на Gap penalty 10, Minimum score threshold 20, Match score 6, Mismatch score -4,я получила файл с таким результатом:
SEQUENCE: Score 42: 10/12 ( 83%) matches, 1 gaps
22 gagcgccaga-ct 33
|| | ||||| ||
48 ctggaggtctaga 36
Получился слишком короткий участок. Поставив немного другие условия Gap penalty 10, Minimum score threshold 15, Match score 10, Mismatch score -4, я получила:
SEQUENCE: Score 120: 22/32 ( 68%) matches, 6 gaps
1 gcgga-t-t-tagctcagttggga-gagcgccaga-ct 33
||| | | | | | || | | || | ||||| ||
72 cgcttaagacacctag-cttgtgtcctggaggtctaga 36
Далее с помощью программы mfold из пакета EMBOSS, которая реализует алгоритм Зукера, была предсказана вторичная структура тРНК. Лучшая из полученных структур (Р=10, номер _2) показана на рис.1:
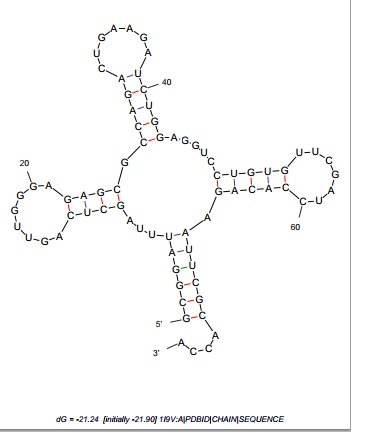
Рис.1. тРНК, вторичная структура.
Должна сказать, что при Р=20 и Р=30 это тоже лучшая структура. Таблица сравнения предсказанных структур и реальной:
| Участок структуры | Позиции в структуре (по результатам find_pair) | Результаты предсказания einverted | Результаты предсказания по алгоритму Зукера |
| Акцепторный стебель | 5'-1-7-3' 5'-66-72-3' всего семь пар |
7 | 6 |
| D-стебель | 5'-49-53-3' 5'-61-65-3' всего пять пар |
2 | 4 |
| T-стебель | 5'-40-44-3' 5'-26-30-3' всего пять пар |
- | 5 |
| Антикодоновый стебель | 5'-10-13-3' 5'-22-25-3' всего четыре пары |
4 | 5 |
| Общее число канонических пар нуклеотидов | 20 | 22 | 18 |
Поиск ДНК-белковых контактов в заданной структуре
Для поистка контактов использовался СКРИПТ| Контакты атомов белка с | Полярные | Неполярные | Всего |
| остатками 2'-дезоксирибозы | 11 | 52 | 63 |
| остатками фосфорной кислоты | 28 | 0 | 28 |
| остатками азотистых оснований со стороны большой бороздки | 5 | 12 | 17 |
| остатками азотистых оснований со стороны малой бороздки | 5 | 13 | 18 |
Можно заметить, что большинство контактов приходится на сахарофосфатный остов. Используем следующие команды для получения изображений:
remediator --old "1MDM.pdb" > "1MDM_old.pdb"
nucplot 1MDM_old.pdb
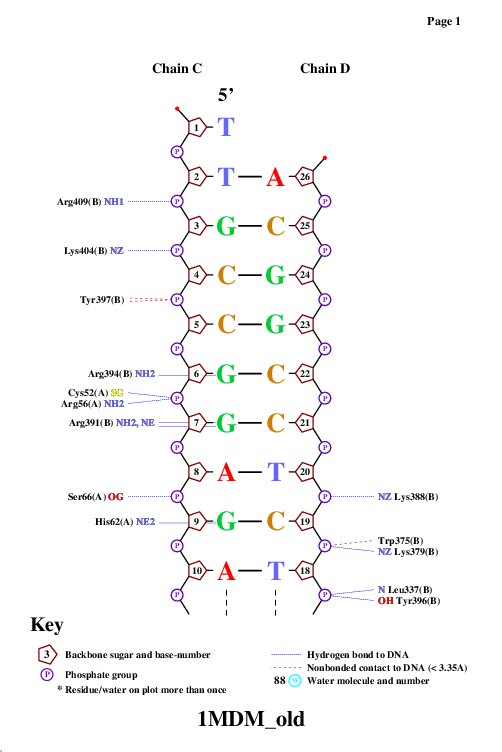
Рис.2.
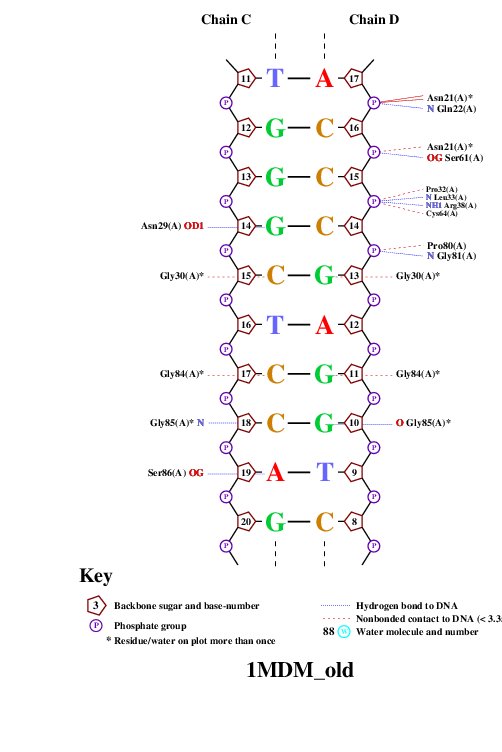
Рис.3.
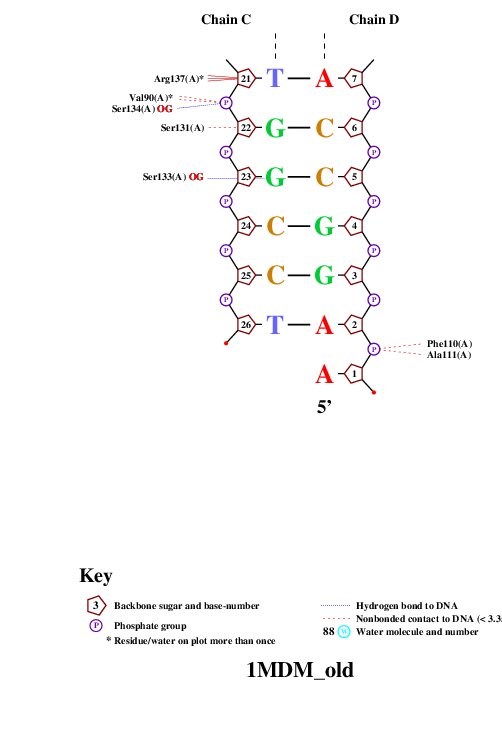
Рис.4.
Аминокислотных остатков с наибольшим числом указанных на схеме контактов с ДНК несколько: остатки Gly85, Gly30, Asn21, Arg391 и Gly84. Все они образуют по 2 контакта.
Пример связи:
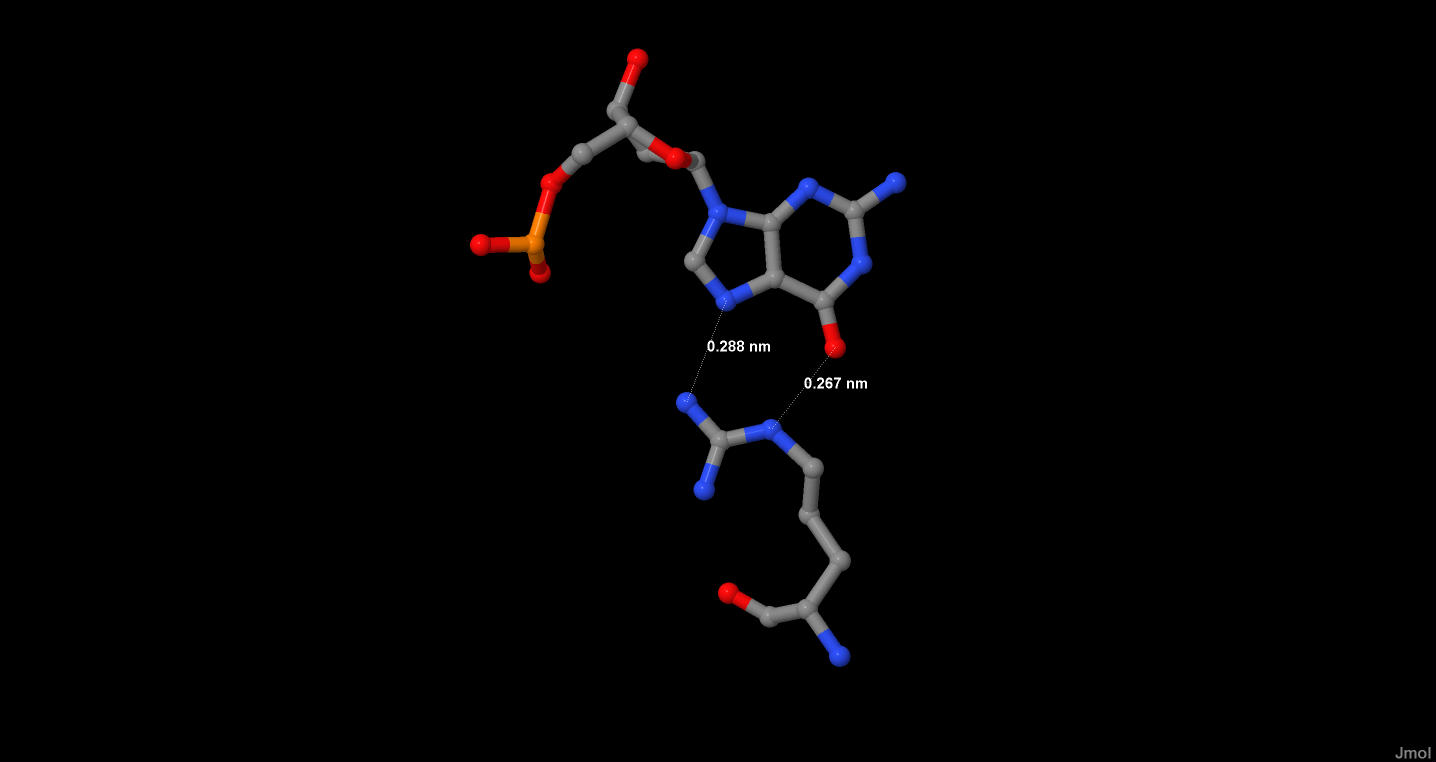
Рис.5.
Сверху показан G7, снизу аргинин391. Также можно увидеть связи между ними.
| © Амосова Алена. 2013 год | Дата последнего изменения: 21 декабря 2013. |