Четвертое занятие
Для выравнивания были получены последовательности 16S рибосомальной РНК каждой из бактерий из базы полных геномов NCBI . Нужные последовательности лежат в файле с расширением .frn. По Ссылке можно скачать файл с полученными последовательностями. Последовательности были выровнены (Muscle) - Скачать Файл с выравниванием был импортирован в программу MEGA и для построения дерева был выберан метод Lest Maximum Likelyhood Tree. Дерево не совпадает с правильным - 3 ветви реконструированы неправильно (см. Рис.1). Это связано с тем, что выравнивания нуклеотидных последовательностей сами по себе менее достоверны, чем выравнивания соответствующих белков (из-за того что нуклеотидные последовательности состоят только из 4-х букв, вероятность случайного совпадения позиций намного больше, чем для белковых последовательностей).
Паралоги
Два гомологичных белка будем называть ортологами, если они из разных организмов и разделение их общего предка на линии, ведущие к ним, произошло в результате видообразования. А два гомологичных белка из одного организма называются паралогами. Чтобы найти в моих бактериях достоверные гомологи белка CLPX_BACSU, было построено дерево этих гомологов. Фаил с полными протеомами моих бактерий можно скачать по Ссылке. Был проведен поиск с помощью локального бласта гомологов с E-value = 0,001 по протеомам моих бактерий. Считая дерево реконструированным верно, укажите несколько пар паралогов и одну-две группы попарно ортологичных белков. Приведите примеры отражённых на дереве эволюционных событий двух типов: 1) дупликация гена; 2) разделение путей эволюции белков в результате видообразования.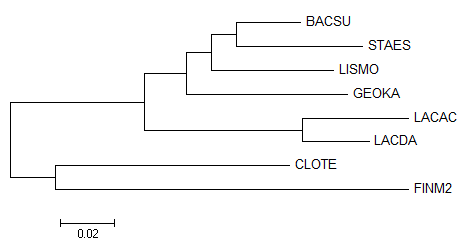
Рис.1 Филогенетическое дерево, построенное по нуклеотидным последовательностям.
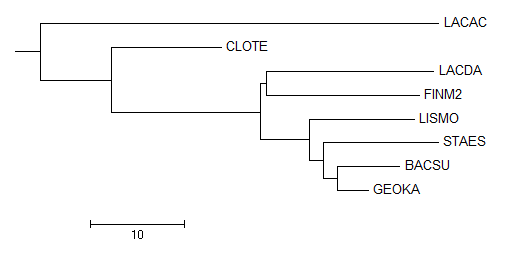
Рис.2 Филогенетическое дерево, укорененное в среднюю точку.
| © Амосова Алена. 2013 год | Дата последнего изменения: 21 декабря 2013. |