Практикум №10. Выравнивание геномов.
Для данного практикума я выбрала организмы рода Bacillus . Последовательность генома я нашла на странице геномов NCBI. Там я воспользовалась сервисом "Browse by Organism". Ввела в окно поиска: "Bacillus" и выбрала 2 вида - Bacillus subtilis - сенная палочка, грамположительная бактерия[1] (AL009126.3) и Bacillus safensis - грамположительная мезофильная гетеротрофная бактерия[2] (CP043404.1). При выборе учитывала, что сборка генома должна быть не ниже хромосомного. Выравнивание делала по 1-ой (и единственной) хромосоме организма.
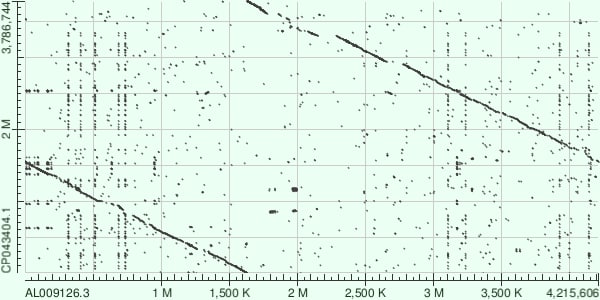
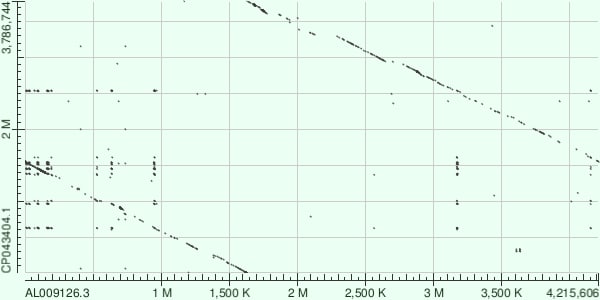
На карте локального сходства видно, что у двух видов начальная точка отсчета (то есть где как бы разрывается кольцевой геном) находится в разных частях ДНК. Это видно из-за того, что в точке отсчета на карте локального сходства не выявлены совпадения.
Выравниваются участки 0 - 1650К хромосомы B.subtilis c и участками 1600К - 0 B.safensis; участки 1650К - 4215К У B.subtilis c и участками 3786К - 1500К B.safensis. Произошла инверсия - поворот участка хромосомы на 180 градусов. В целом, можно сказать, что геномы выровнились неплохо, но имеется ряд делеции/вставок. К примеру, на участке 2150К - 2300K хромосомы B.subtilis произошла вставка.
Если сравнивать результаты алгоритмов blastn и megablast, то можно заметить, что megablast выявил меньше соотвествий по сревнению с blastn (у megablast длина слова, следовательно, меньше совпадений).
Литература
[1]Bacillus Subtilis P.J. Piggot, in Encyclopedia of Microbiology (Third Edition), 2009
[2]Bacillus safensis sp. nov., isolated from spacecraft and assembly-facility surfaces