Практикум №15. Сборка de novo.
В рамках данного практикума мы выполняем задания по сборке генома de novo. Для этого в таблице напротив своей фамилии я нашла код доступа проекта по секвенированию бактерии Buchnera aphidicola.
Далее скачиваю чтения командой:
wget ftp://ftp.sra.ebi.ac.uk/vol1/fastq/SRR424/001/SRR4240361/SRR4240361.fastq.gz
Подготовка чтений командой trimmomatic
Я получила файл all_adapters.fasta объединением файлов с адаптерами.
Далее проводилась обрезка адапретов командой:
java -jar /usr/share/java/trimmomatic.jar SE -threads 20 SRR4240361.fastq.gz SRR4240361_adapttrim.fastq.gz ILLUMINACLIP:all_adapters.fasta:2:7:7 2> log_adapttrim.txt
Далее из файла log_adapttrrim.txt выяснено, что адаптеры составляли 0,47% всех последовательностей чтений.
Затем необходимо было удалить с правых концов чтений нуклеотиды с качеством ниже 20 и оставить только такие чтения, длина которых не меньше 32 нуклеотидов. Для этого использовала команду:
java -jar /usr/share/java/trimmomatic.jar SE -threads 20 SRR4240361_adapttrim.fastq.gz SRR4240361_trim.fastq.gz TRAILING:20 MINLEN:32 2>log_trim.txt
Проанализировав файл log_trim.txt, сделала вывод, что на данном этапе было удалено 403754 (5.58%) чтений. Вес исходного файла 193M, после очистки адаптеров - 192M, после удаления части чтений - 178М.
Подготовка k-меров длины 31.
Команда, использованная на это этапе:
velveth kmers 31 -short -fastq.gz SRR4240361_trim.fastq.gz
где 31 - длина К-меров, kmers - папка, куда перемещаются К-меры.
Cборка генома
Далее была произведена сборка геномов на основе 31-меров. Команда:
velvetg kmers
Анализ качества сборки
Используя файл Log, установила, что N50 сборки = 25683
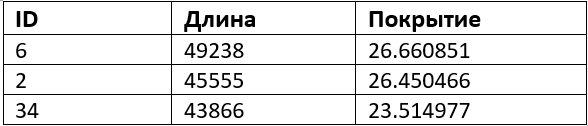
С помощью команды sort -r -n -k 2 stats.txt | less установила длины трёх самых длинных контигов и их покрытие (Результат команды в Таблице 1.)
Есть контиги с аномально большим покрытием, но их длина <<31. К примеру, у контига длиной 1 (ID 62) покрытие составляет 212829.0. Есть и контиги с аномально маленьким покрытием. Например, контиг длиной 63 (ID 169) имеет покрытие 2.952381.
Далее я сравнивала каждый из трех самых длинных контингов с хромосомой Buchnera aphidicola (GenBank/EMBL AC — CP009253). Ниже приведены результаты программы megablast и поясление к ним.
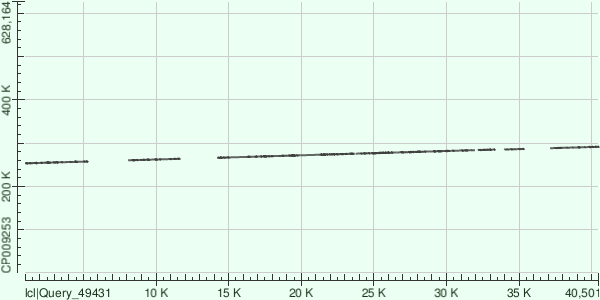
Пояснения к Рис.1.
Фрагмент контига 50-12790 выровнился на участок хромосомы с координатами 127845-140555.
Фрагмент контига 25809-338903 выровнился на участок хромосомы с координатами 153752-161738.
Фрагмент контига 16429-23828 выровнился на участок хромосомы с координатами 144368-151796.
Фрагмент контига 34098-38958 выровнился на участок хромосомы с координатами 161898-166752
Фрагмент контига 38989-45432 выровнился на участок хромосомы с координатами 166750-173180
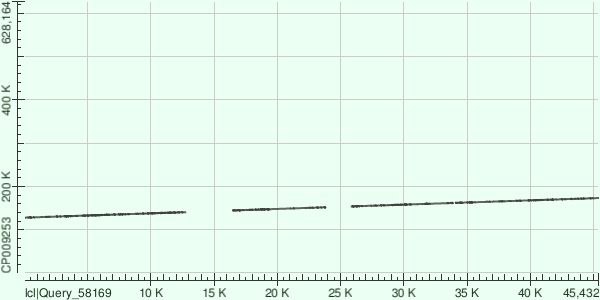
Пояснение к Рис.2.
Фрагмент контига 10984-23268 выровнился на участок хромосомы с координатами 467412-474667
Фрагмент контига 18327-23268 выровнился на участок хромосомы с координатами 462496-467421
Фрагмент контига 40383-43410 выровнился на участок хромосомы с координатами 442877-445895
Фрагмент контига 977-5299 выровнился на участок хромосомы с координатами 253223-257546
Фрагмент контига 34011-35345 выровнился на участок хромосомы с координатами 285200-286535
Фрагмент контига 32205-33314 выровнился на участок хромосомы с координатами 283963-285070
Стоит отметить, что контиг картировался на хромосому в обратном направлении.
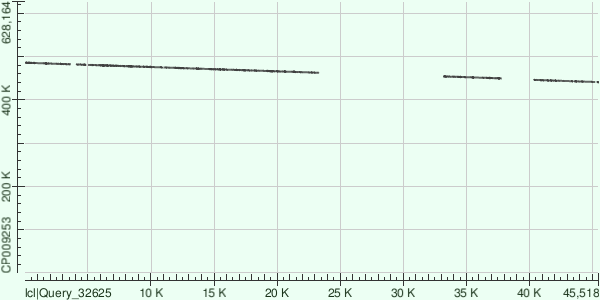
Пояснение к Рис.3.
Фрагмент контига 14198-23677 выровнился на участок хромосомы с координатами 266073-275551
Фрагмент контига 23736-31957 выровнился на участок хромосомы с координатами 275566-283706
Фрагмент контига 8077-11648 выровнился на участок хромосомы с координатами 260224-263784
Фрагмент контига 37135-40501 выровнился на участок хромосомы с координатами 288181-291560
Фрагмент контига 37135-40501 выровнился на участок хромосомы с координатами 288181-291560
На DotPlotе видно, что произошла делеция (разрыв, так называемой линии)