Практикум №2
Задание №1
С помощью программы fiber пакета 3DNA я построила A-, B- и Z-форму дуплекса ДНК, последовательность одной из нитей которого представляет собой 5 раз повторенную последовательность "gatс". Для создания файлов были использованы следующие команды:
- fiber -seq=GATCGATCGATCGATCGATC -a gatc-a.pdb
- fiber -seq=GATCGATCGATCGATCGATC -b gatc-b.pdb
- fiber -z gatc-z.pdb.
Ссылка на сгенерированные файлы ниже:
Задание №2
В данном задании нам нужно было сравнить сгенерированнцю структуру одной из 3-х форм ДНК со структурой той же формы, полученной экспериментальными данными. Для этого я использовала B-форму ДНК.
В ходе выполнения задания мне удалось научиться различать большую и маленькую бороздку ДНК. Заданное мне основание - цитозин - изображено на Рис.1. Я выбрала тот, который находится в положении 13 на B цепи экспериментальной модели. Для создания изображения я использовала программу MarvinSketch.
Атомы, смотрящие в сторону большой бороздки, я раскрасила красным цветом, а атомы, смотрящие в сторону малой, - синим.
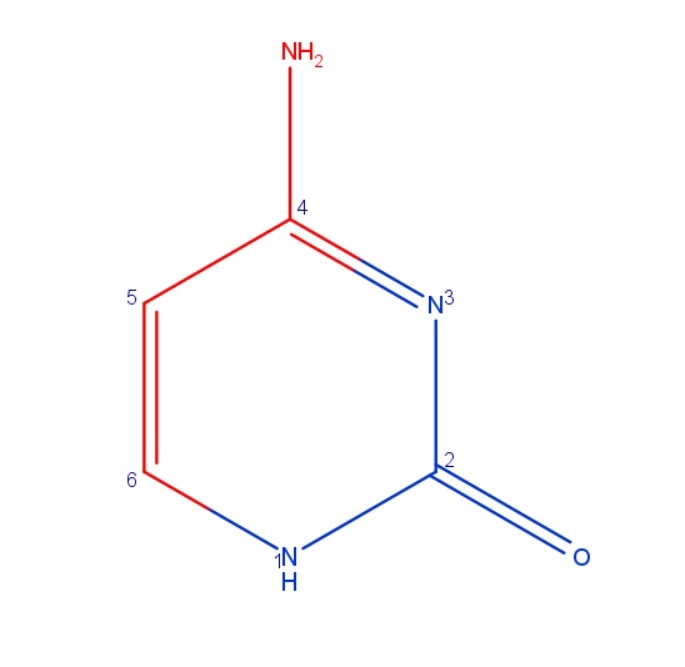
- В сторону малой бороздки обращены атомы: C13.N1, C13.C2, C13.O2, C13.N3
- В сторону большой бороздки обращены атомы: С13.C4, С13.N4, С13.C5, С13.C6
| Форма ДНК | A | B | Z |
|---|---|---|---|
| Тип спирали | Правая | Правая | Левая |
| Шаг спирали (Å) | 28,03 | 33,75 | 42,34 |
| Число оснований на виток | 11 пар | 10 пар | 12 пар |
| Ширина большой бороздки(Å) | 16,81(A10:A-G33:B) | 17,21(G9:A - G29:B) | 18,3(C20:A - C58:B) |
| Ширина малой бороздки(Å) | 7,98(T11:A - C24:B) | 11,69(A14:A - T31:B) | 7,20(G13:A - G31:B) |
Задание №3
В этом задании нам необходимо определить параметры структур нуклеиновых кислот с помощью программ пакета 3DNA. Для его выполнения я использовала программы find_pair и analyze.
Я использовала структуру из PDB с ID: 1ffy. Так как пакет 3DNA работает только со старым форматом PDB, то для начала нужно перевести файл из старого формата в новый с помощью команды:
! remediator --old ''1ffy.pdb'' > ''1ffy_old.pdb
С помощью программы find_pair определяю спаренные основания и положения спиралей в структуре, переправляю результат работы предыдущей программы на вход программе analyze:
find_pair -t 1ffy_old.pdb stdout | analyze
В результате получается ряд файлов. Для дальнейшего анализ буду использовать 1ffy_old.out и stacking.pdb
Нужно сравнить значение торсионных углов модели (данные из презентации Рис.2.) с теоретическими (Рис.3.)

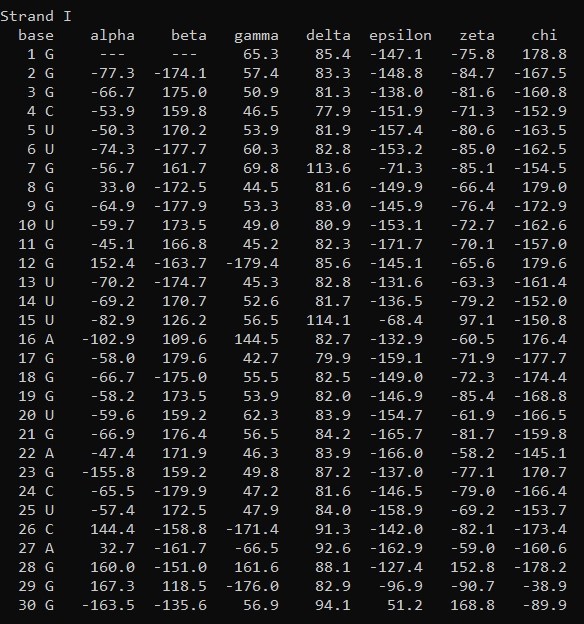
Так как значения α,β,γ углов у А-днк и В-днк схожи, то буду оценивать схожесть δ,ε,ζ,χ углов.
- δ близок к значению А-днк
- ε ближе к В-днк
- ζ одинаково похож и на А-днк, и на В-днк
- χ ближе к А-днк
Мне кажется, что можно сделать вывод о том, что тяжи РНК больше похожи на А-днк.
Далее я рассматривала водородные связи. (Рис.4.)
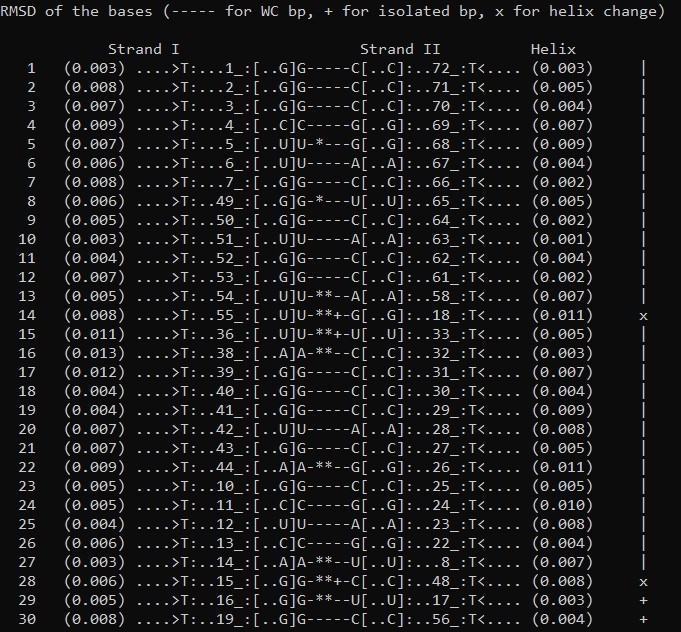
Неканонические пары:
Координаты стеблей:
19G-56C - водородная связь в тРНК, стабилизирующая ее третичную структуру (то есть это комплементарная пара, которая не имеет отношения к стеблям)