Практикум №8
Задание 1.
Подходящий контиг (с CDS) оказался не принадлежащим ни к какой хромосоме (unplaced scaffold).
Характеристики выбранного контига:
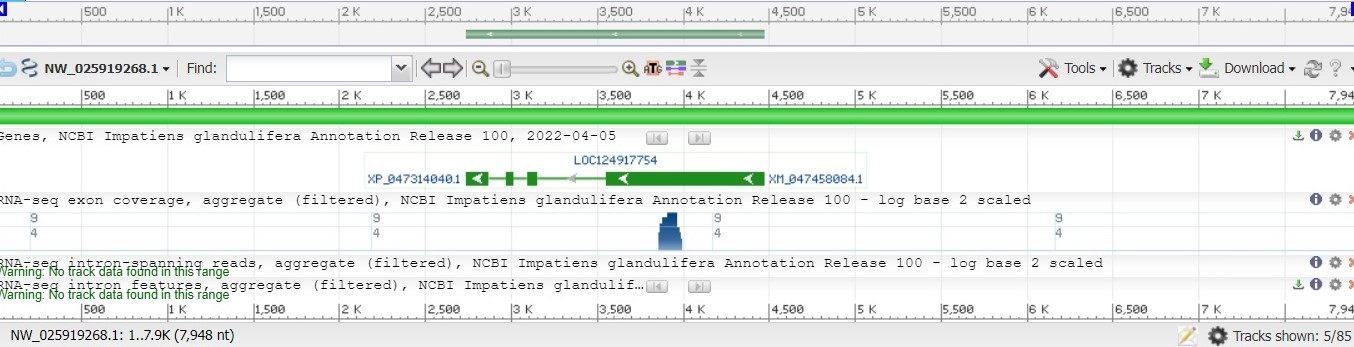
В выбранном контиге есть один CDS, состоящий из 4-х экзонов (отмечены прямоугольниками) и 3-х интронов (отмечены линиями). Получается, что на данном контиге один ген. Ген расположен на "-" цепи ДНК.
Ссылка на последовательность контигаЯ выполнила поиск BLAST по эукариотическим последовательностях для данного контига, при этом исключив класс Magnoliopsida.
1. blastn. Поиск выдал 99 находок. Для поиска я использовала стандартную длину слова - 11, остальные параметры не меняла. Первый десяток находок были из неродственных организмов - среди них не было даже ни одного растения. Мне кажется, что все находки случайны.
2. megablast. Поиск выдал 4 находки. Интересно, что все находки в одном организме, но на разных хромосомах.
3. blastx. Поиск производился в базе данных RefSeq для белков. Изначально делала поиск с исключением класса Mangoliopsida - ничего не нашлось. Я решила попробовать изменить длину слова с 6-ти до 2-х, но и это не помогло. Поэтому я решила не исключать таксоны вообще и задать длину слова - 3. Поиск выдал 14 находок - 11 принадлежат самому организму, 3 - другим.
4. tblastx. Поиск ничего не нашел, а точнее указал на ошибку - нехватку вычислительных ресурсов для выполнения запроса.
Примеры использования алгоритмов:
blastn используется для поиска гомологов нуклеотидной последовательности в нуклеотидной базе данных. Позволяет сравнивать геномы неблизкородственных организмов, а также устанавливать, к какому организму принадлежит тот или иной фрагмент генома. megablast можно использовать, когда мы уверены, что наша последовательность найдется в каким-то конкретном организме. Это актуально, когда поиск проводится внутри одного и того же рода. blastx используется, например, для аннотации последовательности, так как последовательность переводится из нуклеотидной в аминокислотную и поиск проводится по белковой базе данных. tblastx используется, когда у нас имеется последовательность и мы хотим поискать гомолога в таких геномах, в которых еще не предсказаны гены.
Задание 2. Поиск в геноме эукариота гены основных рибосомальных РНК по далекому гомологу
Для начала проиндексирую последовательность генома моего организма. Чтобы не мучиться с длинным названием, я переименовала свою последовательность как genomee.fna
makeblastdb -in genomee.fna -dbtype nucl
Далее я провела локальный поиск BLAST отдельно для каждой рРНК по полученной базе данных с помощью blastn. Использовала его, тк ведем поиск по последовательностям, а не по белкам и организмы неродственные. Использовала команды:
blastn -task blastn -query 16s.fna -db genomee.fna -out 16s.out -outfmt 7 -evalue 0.0001
blastn -task blastn -query 23s.fna -db genomee.fna -out 23s.out -outfmt 7 -evalue 0.0001
Я уменьшила E-value до 0.0001 в обоих запросах. На вход подавались последовательности 16s.fna и 23s.fna, которые являются последовательностями рибосомальных РНК 16S и 23S E.coli, соответсвенно. 16S РНК принадлежит малой субъединице рРНК, 23S - большой.
Для 16S получилось 147 находок, а у 23S - 207 находок. Для последовательности 16S рРНК найдены следующие гомологи: два гена в ДНК хлоропласта 16S рРНК, что вполне естественно, ведь в хлоропластах находятся прокариотические рибосомы. Так как результат поиска выдал много находок, то я решила проверить десяток лучших. По результатам проверки могу сделать вывод, что не нашла среди них какие-то аннотированные гены (в том числе гены рРНК). Для 23S рРНК также найден гомолог - ген 16S рРНК в хлоропластной ДНК. В этом случае ситуация аналогичная - я проверила еще несколько лучших находок - аннотированных генов среди них нет.