Практикум №2. Практикум по визуализации деревьев.
1. Составление списка гомологичных белков, включающих паралоги
Для выполнения данного задания я скопировала в свою директорию протеомы выбранных мною бактерий. Затем объединила их в один файл - all.fasta
cat *.fasta > all.fasta
Далее создала локальную базу данных и произвела поиск гомологов командами:
makeblastdb -in all.fasta -dbtype prot -out db.fasta
blastp -query clpx.fasta -db db.fasta -out blastp.out -evalue 0.001 -outfmt 7
Получены следующие находки:
Score E
Sequences producing significant alignments: (Bits) Value
sp|A1SME0|CLPX_NOCSJ ATP-dependent Clp protease ATP-binding subun... 528 0.0
sp|Q9CBY6|CLPX_MYCLE ATP-dependent Clp protease ATP-binding subun... 519 0.0
sp|Q8FN57|CLPX_COREF ATP-dependent Clp protease ATP-binding subun... 518 0.0
sp|A0JXL2|CLPX_ARTS2 ATP-dependent Clp protease ATP-binding subun... 516 0.0
tr|Q47MU4|Q47MU4_THEFY ATP-dependent Clp protease ATP-binding sub... 516 0.0
sp|Q6AFZ6|CLPX_LEIXX ATP-dependent Clp protease ATP-binding subun... 509 0.0
sp|Q8G5R1|CLPX_BIFLO ATP-dependent Clp protease ATP-binding subun... 432 7e-150
tr|A0K1M3|A0K1M3_ARTS2 ATPase AAA-2 domain protein OS=Arthrobacte... 54.3 4e-08
tr|Q8G871|Q8G871_BIFLO Protease OS=Bifidobacterium longum (strain... 51.2 5e-07
tr|Q8FMH5|Q8FMH5_COREF Putative endopeptidase Clp ATP-binding cha... 47.0 9e-06
tr|Q47MZ2|Q47MZ2_THEFY ATPase OS=Thermobifida fusca (strain YX) O... 43.9 9e-05
sp|Q8G6B7|RUVB_BIFLO Holliday junction ATP-dependent DNA helicase... 42.7 1e-04
tr|Q6ACQ0|Q6ACQ0_LEIXX ATP-dependent zinc metalloprotease FtsH OS... 43.1 1e-04
tr|Q8G3S2|Q8G3S2_BIFLO ATP-dependent zinc metalloprotease FtsH OS... 43.1 1e-04
sp|A0JXB1|RUVB_ARTS2 Holliday junction ATP-dependent DNA helicase... 42.4 2e-04
tr|A1SDV1|A1SDV1_NOCSJ ATP-dependent zinc metalloprotease FtsH OS... 42.0 3e-04
tr|Q47KU4|Q47KU4_THEFY ATP-dependent zinc metalloprotease FtsH OS... 42.0 4e-04
tr|A0JR82|A0JR82_ARTS2 ATP-dependent zinc metalloprotease FtsH OS... 41.6 4e-04
tr|A0K236|A0K236_ARTS2 AAA ATPase, central domain protein OS=Arth... 41.6 4e-04
sp|Q9CD58|FTSH_MYCLE ATP-dependent zinc metalloprotease FtsH OS=M... 40.8 8e-04
sp|A1SJA7|RUVB_NOCSJ Holliday junction ATP-dependent DNA helicase... 40.4 8e-04
sp|P24428|CLPC_MYCLE Probable ATP-dependent Clp protease ATP-bind... 40.4 0.001
tr|Q8FMG2|Q8FMG2_COREF ATP-dependent zinc metalloprotease FtsH OS... 40.4 0.001
Реконструкция и визуализация
Реконструировала дерево найденных гомологов программой FastME c параметрами: "Gamma distributed rates across sites" — No, "Starting tree" — BIONJ, "No refinement", 100 бутстреп реплик. Текстовая выдача
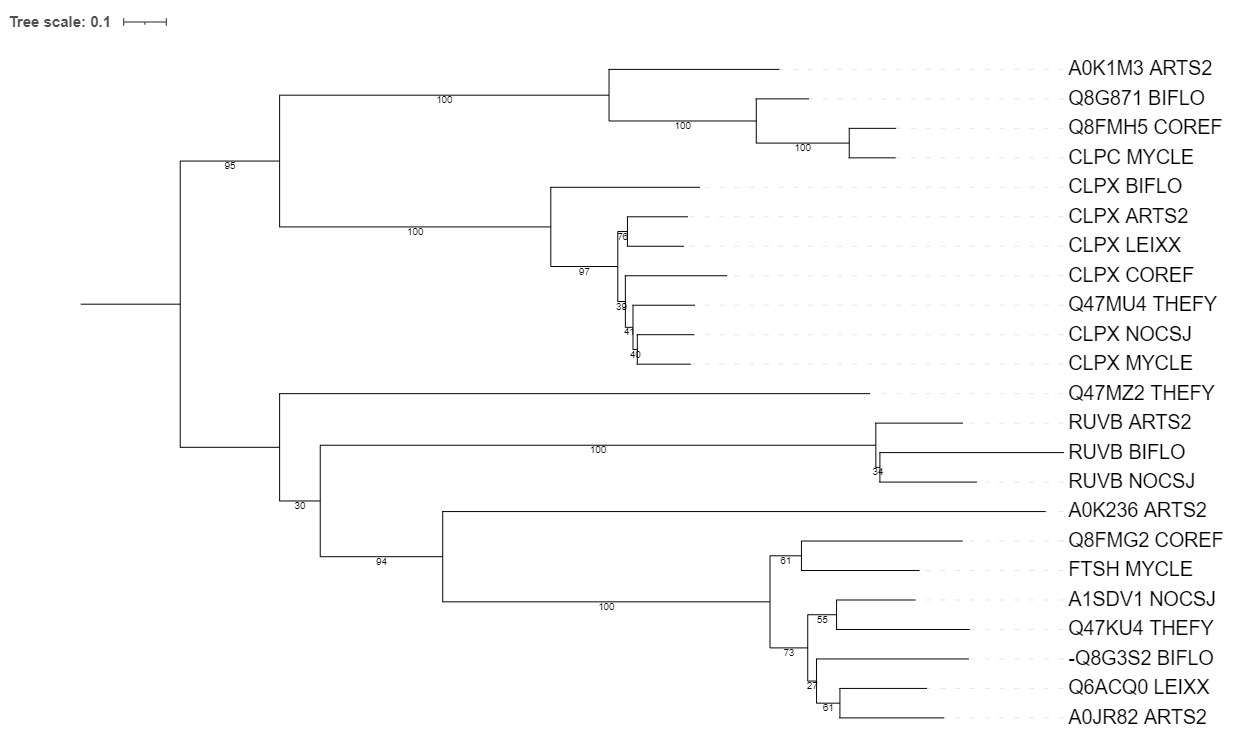
Сделала укоренение дерева в среднюю точку. Исходя из изображения, можно выделить следующие ортологи: CLPX_ARTS2 и CLPX_LEIXX, CLPX_NOCSJ и CLPX_MYCLE, RUVB_ARTS2 и RUVB_NOCSJ. Паралоги - A0JR82_ARTS2 и A0K236_ARTS2, Q8G871_BIFLO и CLPX_BIFLO, CLPC_MYCLE и CLPX_MYCLE.
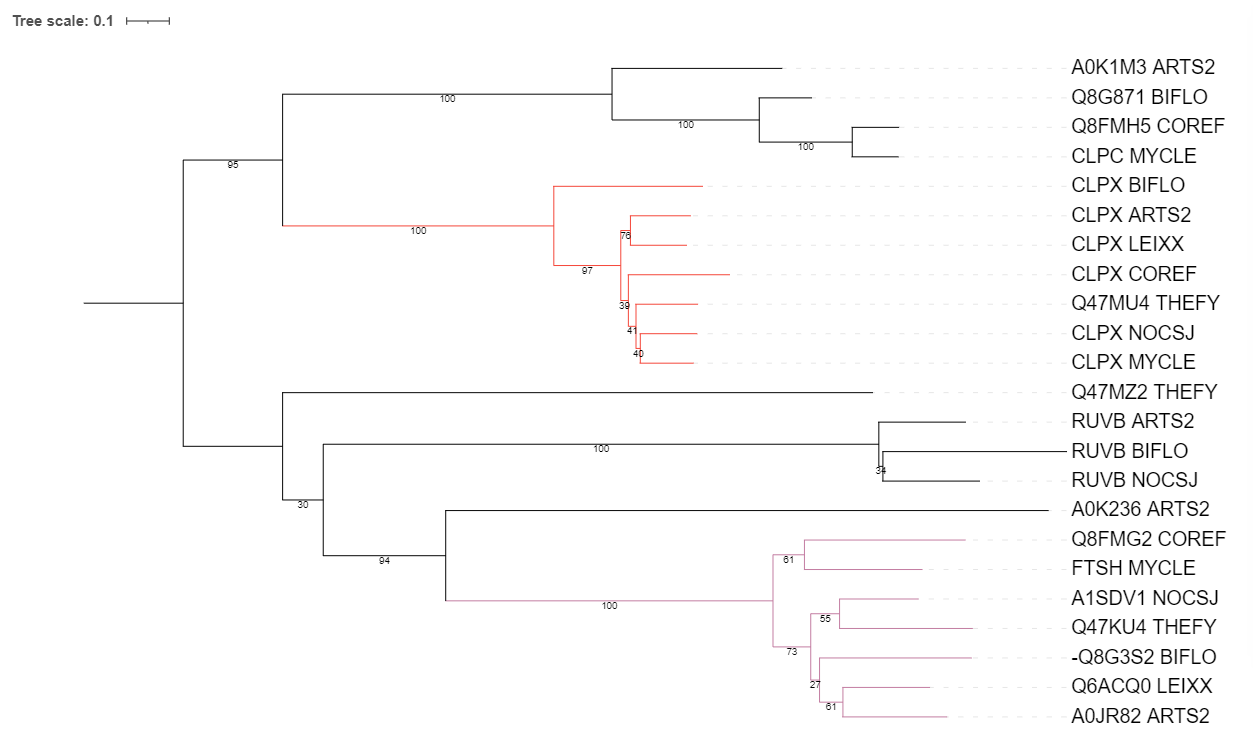
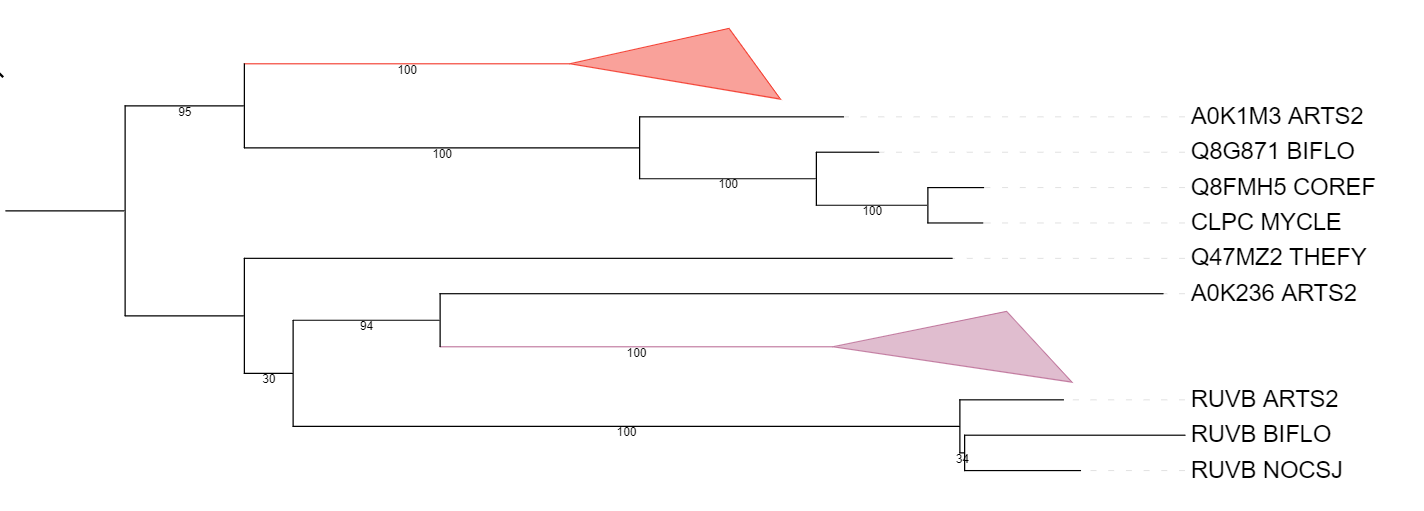
В обоих группах представлены белки из всех выбранных мною бактерий. Группа АТФ-зависимых Clp протеаз очень похожа на эталонное дерево бактерий, отличается только положение ветви бактерии MYCLE. Группа АТФ-зависимых цинковых металлопротеаз существенно отличается от эталонного дерева.