A- и В- формы ДНК. Структура РНК
Моделирование A-, B- и Z-формы дуплекса ДНК с помощью пакета 3DNA
- Запускаем программу Рutty и, используя протокол ssh, подключаемся к серверу kodomo.cmm.msu.ru.
- Используем свой логин и пароль.
- С помощью команды cd term3/block1/pr2 переходим в рабочую директорию.
- Введем следующие команды, что бы указать путь к 3DNA:
- export PATH=${PATH}:/home/preps/golovin/progs/X3DNA/bin
- export X3DNA=/home/preps/golovin/progs/X3DNA
- C помощью программы fiber пакета 3DNA построим A-, B- и Z-форму дуплекса ДНК, последовательность одной из нитей которого представляет собой 5 раз повторенную последовательность "gatc". Структуры сохраним в файлах gatc-a.pdb, gatc-b.pdb и gatc-z.pdb, соответственно, с помощью следующих команд:
- fiber -a gatc-a.pdb
- fiber -b gatc-b.pdb
- fiber -z gatc-z.pdb
Средства JMol для работы со структурами нуклеиновых кислот
Используйя файл А-формы, полученный с помощью программы fiber пакета 3DNA, средствами JMol выделены разные атомы и химические группировки:
- С помощью команд wireframe 50, restrict backone, cartoons off, color magenta выделен сахарофосфатный остов ДНК (рис.1)
- Используя различные цвета и команду wireframe 50 выделены все нуклеотиды (рис.2)
- Выделены только аденины, также при помощи цвета (рис.3)
- C помощью команд select g and *.N7, spacefill 300, label %n%r и цвета был выделен атом N7 во всех гуанинаx (рис.4)
 |
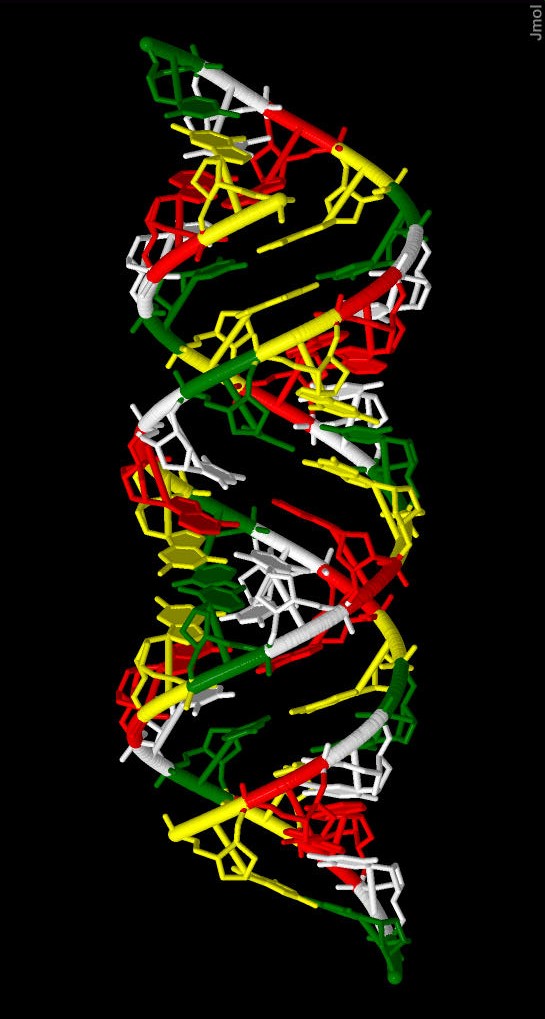 |
Рисунок 1.
Сахарофосфатный остов A-формы ДНК. |
Рисунок 2.
Нуклеотиды A-формы ДНК
(аденин - красный; гуанин - желтый; тимин - белый; цитозин - зеленый).
|
 |
 |
Рисунок 3.
Аденины A-формы ДНК.
Окрашены в розовый. |
Рисунок 4.
Атомы N7 в гуанинаx A-формы ДНК.
Сами гуанины окрашены в голубой, нужные атомы представлены в виде
шариков и выделены розовым, их номера также подписаны. |
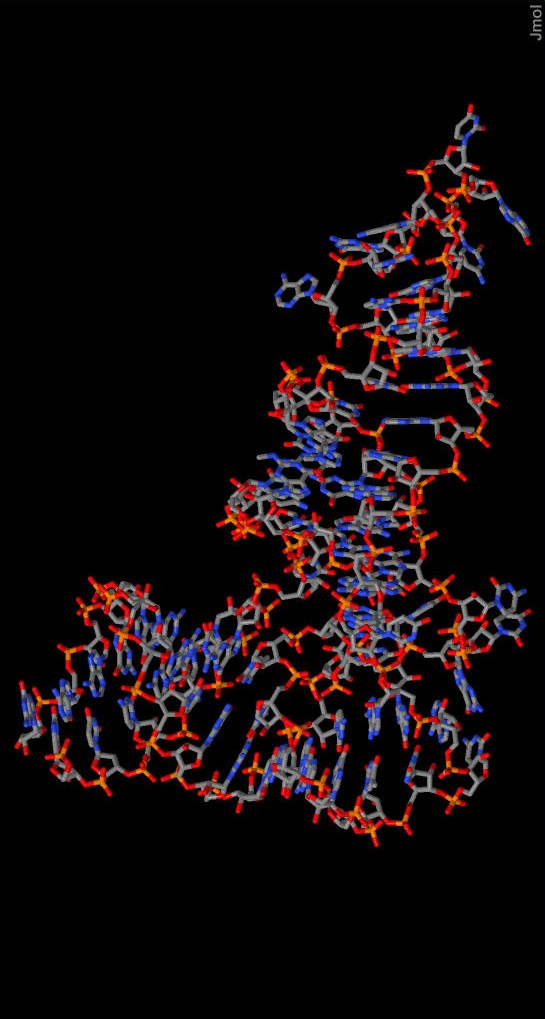
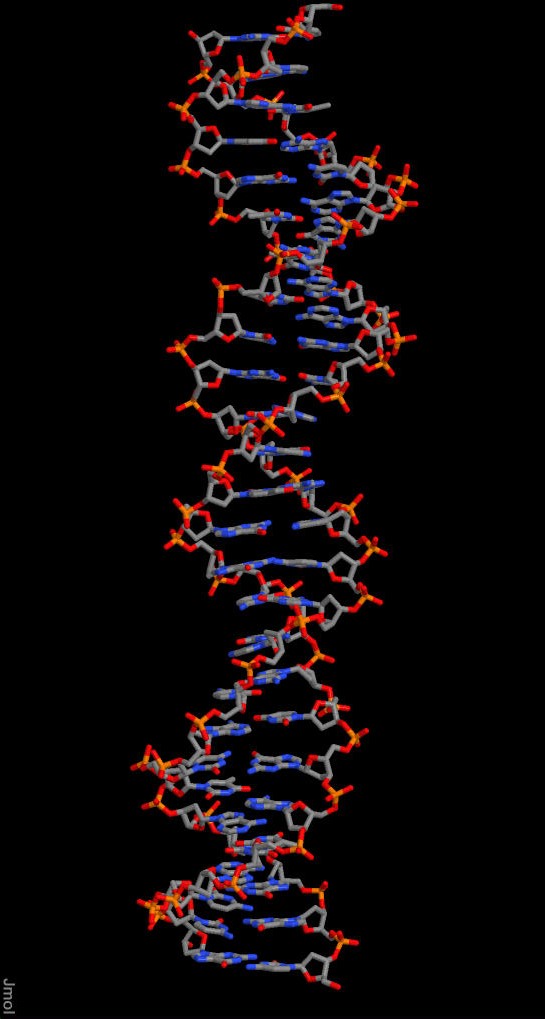
Исследование структур 1f7v и 1rio с помощью Jmol
При помощи идентификаторов PDB были найдены соответствующие структуры на сайте PDB. Для работы был выбран биологический вариант структур. Далее заданные структуры ДНК и РНК были проверены на наличие разрывов, их не выявлено. Результаты работы предствлены на Рис.5,6.
 |
 |
| Рисунок 5. Молекула тРНК |
Рисунок 6. Молекула ДНК |
| Изображения нуклеиновых кислот представлены в проволочной модели и окрашены в стандартные цвета с помощтью команды color cpk. |
Координаты атомов только тРНК и ДНК в отдельных файлах для дальнейшей работы.
Сравнение структур 3-х форм ДНК с помощью средств JMol
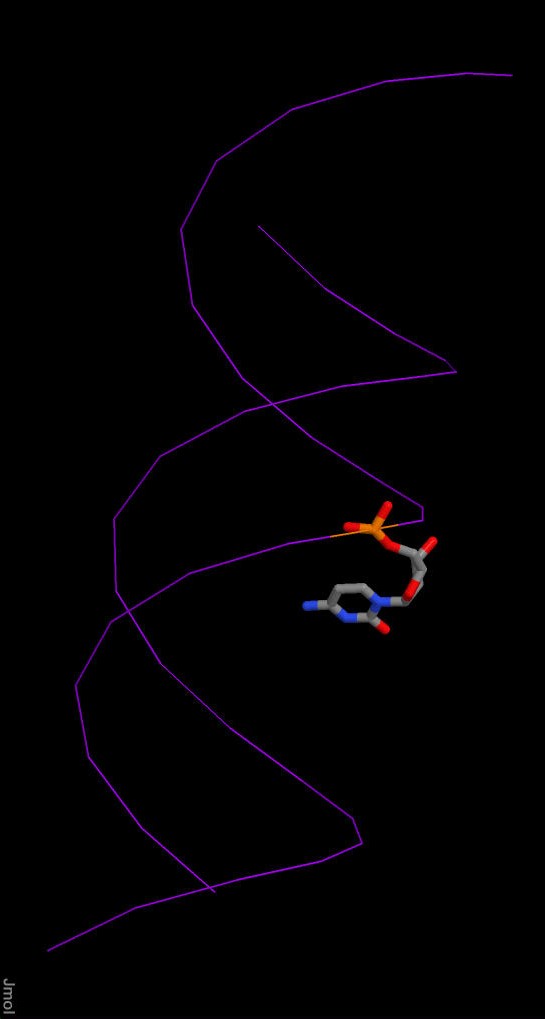
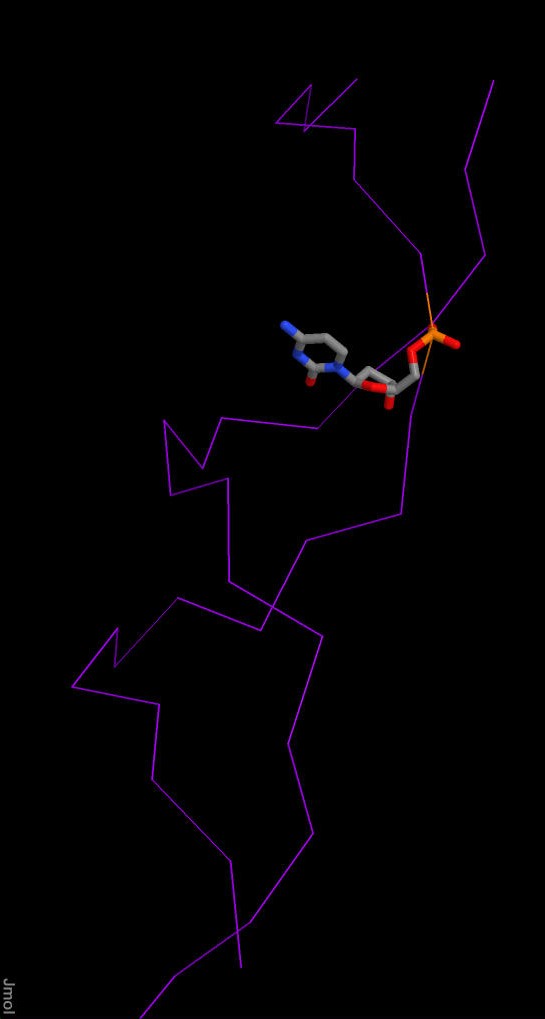
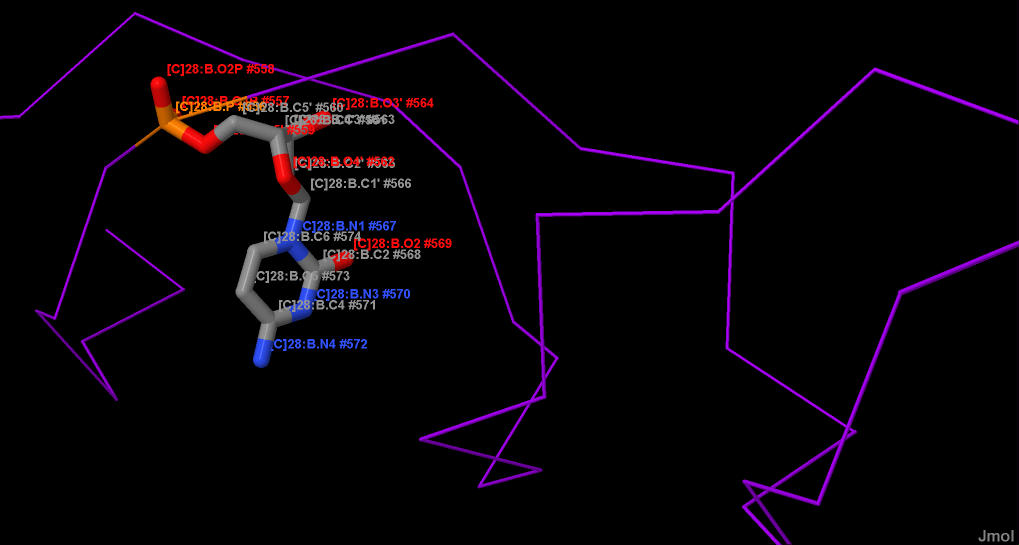
В Jmol были открыты файлы, полученные с помощью программы fiber пакета 3DNA для рассмотрения структуры и визуального определения большо и малой бороздок. Затем для определения, какие атомы основания обращены в сторону большой бороздки, а какие - в сторону малой, был выбран цитозин 28 (Рис.7-15 и Таблица 1).
Таблица 1. Номера атомов цитозина 28, обращенных в сторону большой и малой бороздок.
|
A-форма ДНК |
B-форма ДНК |
Z-форма ДНК |
|
Номера атомов цитозина, обращененных в сторону большой бороздки |
c28.c4, c28.c5, c28.n4 |
c28.c4, c28.c5, c28.n4 |
c28.c4, c28.c5, c28.c6, c28.n3, c28.n4 |
|
Номера атомов цитозина, обращененных в сторону малой бороздки |
c28.c2, c28.n1, c28.o2 |
c28.c2, c28.n1, c28.o2 |
c28.c2, c28.n1, c28.o2 |
|
Остальные атомы цитозина |
c28.n3, c28.c6 |
c28.n3, c28.c6 |
- |
 |
 |
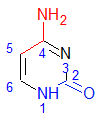 |
Рисунок 7, 8.
Цитозин 28. Целый остов А-формы ДНК и фрагмент структуры. |
Рисунок 9.
Модель цитозина 28, полученное с помощью ChemSketch для А-формы ДНК. Атомы, смотрящие в сторону большой бороздки, выделены красным цветом, в сторону малой - синим. |
 |
 |
 |
Рисунок 10, 11.
Цитозин 28. Целый остов B-формы ДНК и фрагмент структуры. |
Рисунок 12.
Модель цитозина 28, полученное с помощью ChemSketch для B-формы ДНК. Атомы, смотрящие в сторону большой бороздки, выделены красным цветом, в сторону малой - синим. |
 |
 |
 |
Рисунок 13, 14.
Цитозин 28. Целый остов Z-формы ДНК и фрагмент структуры. |
Рисунок 15.
Модель цитозина 28, полученное с помощью ChemSketch для Z-формы ДНК. Атомы, смотрящие в сторону большой бороздки, выделены красным цветом, в сторону малой - синим. |
Оригинальный файл ChemSketch можно скачать здесь.
Сравнение основных спиральных параметров разных форм ДНК
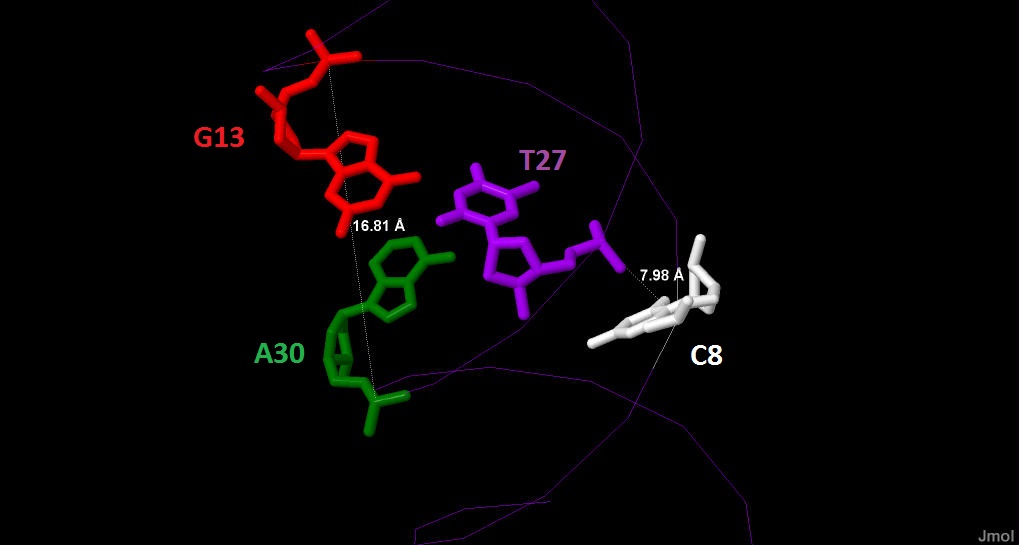
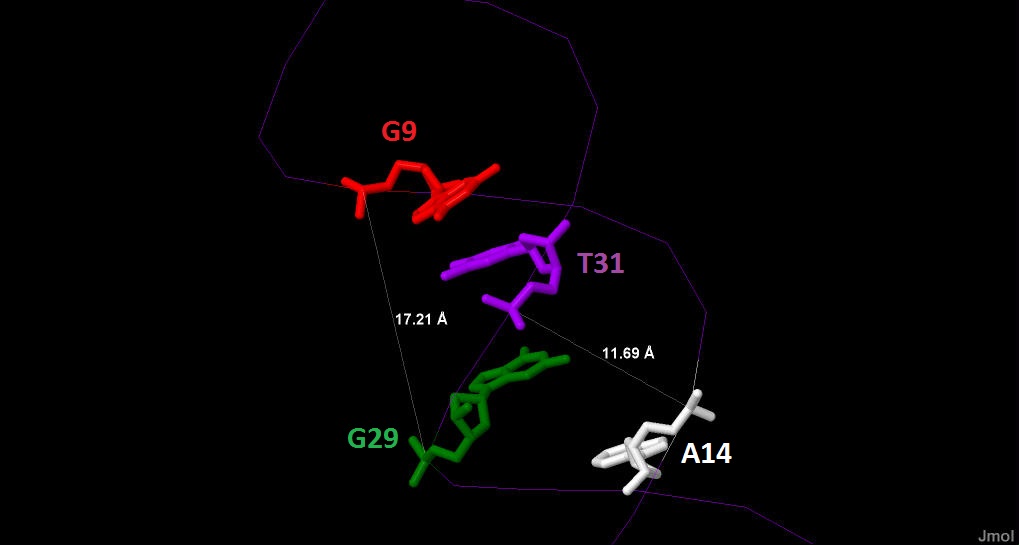
Результаты изучения структур разных форм ДНК с помощью Jmol (Рис. 16-18) внесены в Таблицу 2.
Таблица 2. Сравнение основных спиральных параметров разных форм ДНК.
|
A-форма |
B-форма |
*Z-форма |
| Тип спирали (правая или левая) |
Правая |
Правая |
Левая |
| Шаг спирали (Å) |
28.03 |
33.75 |
43.50 |
| Число оснований на виток |
11 |
10 |
12 |
| Ширина большой бороздки (Å) |
16.81 (от гуанина) |
17.21 (от гуанина) |
18.30 (от цитозина) |
| Ширина малой бороздки (Å) |
7.98 (от цитозина) |
11.69 (от аденина) |
9.87 (от гуанина) |
 |
 |
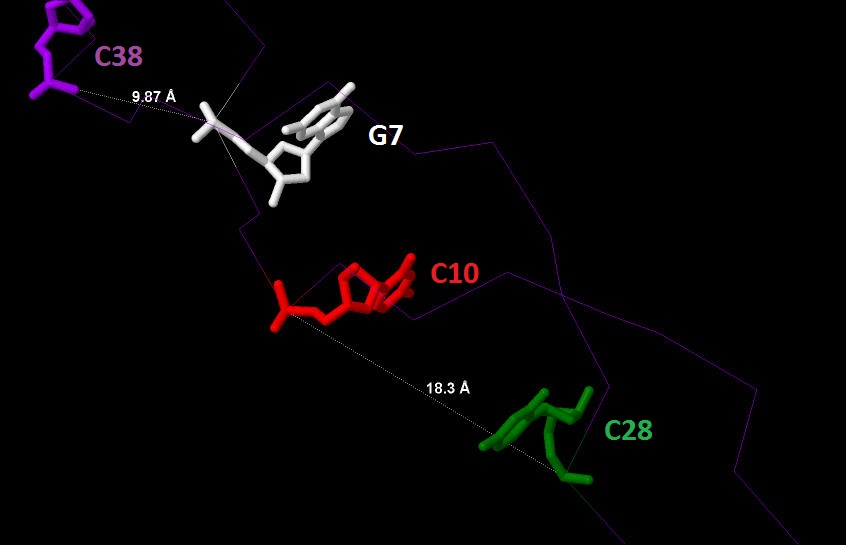 |
Рисунок 16.
Большая и малая борозды А-ДНК, измеренные в Å с помощью Jmol. |
Рисунок 17.
Большая и малая борозды B-ДНК, измеренные в Å с помощью Jmol. |
Рисунок 18.
Большая и малая борозды Z-ДНК, измеренные в Å с помощью Jmol. |
Сравнение торсионных углов в структурах А- и В-форм
С помощью команды Settings->Torsion программы JMol были измерены торсионные углы цитозина. Далее было проведено сравнение значения углов в А- и В-форме с приведенными в презентации.
Таблица 3. Сравнение торсионных углов в структурах А- и В-форм ДНК (углы измерены в градусах).
|
α (P-O5') |
β (O5'-C5') |
γ (C5'-C4') |
δ (C4'-C3') |
ε (C3'-O3') |
ξ (O3'-P |
χ (C1'-N) |
|
A-ДНК |
-51.70 |
174.80 |
41.67 |
79,11 |
-147.78 |
-75,15 |
-157,18 |
|
В-ДНК |
-29.89 |
136.38 |
31.17 |
143.34 |
-140,77 |
-160.52 |
-97.96 |
|
Значения торсионных углов из презентации |
|
A-ДНК |
-62 |
173 |
52 |
88 или 3 |
178 |
-50 |
-160 |
|
В-ДНК |
-63 |
171 |
54 |
123 или 131 |
155 |
-90 |
-117 |
Значения торсионных углов довольно сильно отличаются от приведенных в презентации.
Определение параметров структур нуклеиновых кислот с помощью программ пакета 3DNA
Так как пакет 3DNA работает только со старым форматом PDB, то для перевода файлов gatc-a.pdb, gatc-b.pdb и gatc-z.pdb в старый формат была использована программа remediator, установленная на kodomo. Для этого была введена команда:
- remediator --old ''XXXX.pdb'' > ''XXXX_old.pdb
Для анализа структур нуклеиновых кислот были использованы программы find_pair и analyze. Например, программа find_pair определяет спаренные основания и положения спиралей в структуре, для этого надо ввести команду:
- find_pair -t XXXX.pdb XXXX.fp
Полученные данные необходимы для работы analyze, следовательно, необходимо перенаправить результат работы find_pair на вход программе analyze:
- find_pair -t XXXX.pdb stdout | analyze
В результате был создан ряд файлов с описанием разных параметров структуры, в файле XXXX.out можно найти описание водородных связей, значения всех торсионных углов, ширину малой и большой бороздки и т.д.
При сравнении значенияй соответствующих торсионных углов в структурах A-, B- и Z-форм ДНК, очевидно, что больше всего отличаются значения следующих углов: у A- и B-форм - δ и χ; у A- и Z-форм - α; у B- и Z-форм - α, ξ и χ.
Значения торсионных углов и средние значения каждого из них в структуре 1F7V приведены в файле. Cтруктура 1F7V больше всего похожа на A-форму ДНК, исходя из значений торсионных углов.
В заданной структуре ДНК были определены, с помощью программы Excel, средние значения каждого из торсионных углов (краевые нуклеотиды не рассмотрены), результат можно посмотреть здесь. Cамыми "деформированными" нуклеотидами (с наиболее отклоняющимся значением какого-либо или нескольких торсионных углов) являются t6:t и a22:u, образующие комплементарную пару в ДНК.
Исследование структуры тРНК
Определение структуры водородных связей
- Акцепторный стебель состоит из участка 901-907 и комплементарного ему участка 966-972.
- Т-стебель состоит из участка 949-953 и комплементарного ему участка 961-965.
- D-стебель из участка 910-913 и комплементарного ему участка 922-925.
- Антикодоновый стебель из участка 939-944 и комплементарного ему участка 926-931.
- 9 неканонических пар оснований присутствуют в структуре тРНК, они поддерживают структуру стеблевых дуплексов. Ее также поддерживают 13 пар канонических оснований.
Например, неканоническая пара - B:.913_:[..C]C-*--xC[..C]:.922_:B
Поиск возможных стекинг-взаимодействий
В файле 1f7v_old.out были найдены данные о наибольшей площади перекрывания 2-х пар азотистых оснований, следовательно, возможны следующие стекинг-взаимодействия:
step i1-i2 i1-j2 j1-i2 j1-j2 sum
7 Gc/GC 5.97( 1.41) 0.00( 0.00) 0.00( 0.00) 7.08( 4.30) 13.05( 5.71)
12 Gu/aC 7.08( 1.99) 0.00( 0.00) 0.00( 0.00) 4.95( 1.78) 12.03( 3.77)
13 uP/Ga 5.41( 1.91) 0.00( 0.00) 0.00( 0.00) 7.84( 2.58) 13.25( 4.49)
Далее с помощью следующих команд (где x=7, 12, 13):
- ex_str -x stacking.pdb stepx.pdb
- stack2img -cdolt stepx.pdb stepx.ps
и программы Ghost_View можно получить изображение нескольких наиболее сильных стекинг-взаимодействий (Рис.19-21).
 |
 |
 |
Рисунок 19.
Наиболее сильное стекинг-взаимодействие. Секция 7. |
Рисунок 20.
Наиболее сильное стекинг-взаимодействие. Секция 12. |
Рисунок 21.
Наиболее сильное стекинг-взаимодействие. Секция 13. |
© Novikova Maria, 2013
Последнее обновление: 25.09.2013



















