EMBOSS
Программа getorf пакета EMBOSS
Был создан файл с записью D89965 банка EMBL при помощи команды entret. Запустила программу getorf так, чтобы получить набор трансляций всех открытых рамок данной последовательности, которые определены при использовании стандартного кода и одновременно удовлетворяют следующим условиям:
- Длина не менее 30 аминокислотных остатков
- Начинаются со старт-кодона (или начала последовательности) и заканчиваются стоп-кодоном (или концом последовательности)
Для этого была использована команда:
getorf d89965.entret -minsize 90 -find 1
Был получен следующий результат:
MQFHPRLPAVLQVCAACDRYASLLPAQRRL
>D89965_1 [66 - 155] Rattus norvegicus mRNA for RSS, complete cds.
MQFHPRLPAVLQVCAACDRYASLLPAQRRL
>D89965_2 [56 - 169] Rattus norvegicus mRNA for RSS, complete cds.
MISDAVSSATASSASSLRSMRSVRQSFASSTAALTRWP
>D89965_3 [163 - 432] Rattus norvegicus mRNA for RSS, complete cds.
MALMHFQFTFKQFEQRKSIRSTARKARDDFVVVQTADLFHVAFHYGIAQRGLTITSDDHM
AVTAYAYYSCHELTPWLRIQSTNPVQKYGA
>D89965_4 [218 - 3] (REVERSE SENSE) Rattus norvegicus mRNA for RSS, complete cds.
MLLRCSNCLNVNWKCIRAIWSKPPLSWQKTGVPIACCANLKHCWQSRMKLHRLSSPVTVT
WCSQKTILLLSA
>D89965_5 [294 - 1] (REVERSE SENSE) Rattus norvegicus mRNA for RSS, complete cds.
MKGNVKKVRRLYNDKVIAGFAGGTADAFTLFELFERKLEMHQGHLVKAAVELAKDWRTDR
MLRKLEALLAVADETASLIITGNGDVVQPENDLIAIGS
CDS в транслированном виде выглядит следующим образом:
FT /translation="MALMHFQFTFKQFEQRKSIRSTARKARDDFVVVQTADLFHVAFHY
FT GIAQRGLTITSDDHMAVTAYAYYSCHELTPWLRIQSTNPVQKYGA"
Наиболее всего на CDS с координатами 163..435 похожа запись:
>D89965_3 [163 - 432] Rattus norvegicus mRNA for RSS, complete cds.
MALMHFQFTFKQFEQRKSIRSTARKARDDFVVVQTADLFHVAFHYGIAQRGLTITSDDHM
AVTAYAYYSCHELTPWLRIQSTNPVQKYGA
Последовательность, к которой относится запись D89965 - P0A7B8:
>P0A7B8
MTTIVSVRRNGHVVIAGDGQATLGNTVMKGNVKKVRRLYNDKVIAGFAGGTADAFTLFEL
FERKLEMHQGHLVKAAVELAKDWRTDRMLRKLEALLAVADETASLIITGNGDVVQPENDL
AIGSGGPYAQAAARALLENTELSAREIAEKALDIAGDICIYTNHFHTIEELSYKA
Она выравнивается с пятой найденной последовательностью:
P0A7B8 1 MTTIVSVRRNGHVVIAGDGQATLGNTVMKGNVKKVRRLYNDKVIAGFAGG 50
|||||||||||||||||||||||
D89965_5 1 ---------------------------MKGNVKKVRRLYNDKVIAGFAGG 23
P0A7B8 51 TADAFTLFELFERKLEMHQGHLVKAAVELAKDWRTDRMLRKLEALLAVAD 100
||||||||||||||||||||||||||||||||||||||||||||||||||
D89965_5 24 TADAFTLFELFERKLEMHQGHLVKAAVELAKDWRTDRMLRKLEALLAVAD 73
P0A7B8 101 ETASLIITGNGDVVQPENDLIAIGSGGPYAQAAARALLENTELSAREIAE 150
|||||||||||||||||||||||||
D89965_5 74 ETASLIITGNGDVVQPENDLIAIGS------------------------- 98
P0A7B8 151 KALDIAGDICIYTNHFHTIEELSYKA 176
D89965_5 98 -------------------------- 98
Выводы:
- Программой были найдены пять рамок считывания, из которых наиболее близка к CDS только одна.
- Программа работает хорошо, наиболее подходящая найденная последовательность не отличается от исходной.
- Возможно, причины присутствия большего количества рамок в том, что при прохождении последовательности программа находит старт-кодоны и стоп-кодоны, азотистые основания которых, возможно, принадлежат разным триплетам или находятся в интронах.
- Было обнаружено, что две одинаковые последовательности относятся к разным организмам - кишечная палочка и крыса. Такие результаты могли быть получены из-за загрязнения пробы бактериями.
Файлы-списки
Выполненные команды, для получения файла.
- entret sw:adh*_* adh.fasta
- infoseq -only -usa adh.fasta > 1
- grep -f organisms 1 > 2
- seqret @2 seq.fasta
EnsEMBL
Был проведен поиск информации о гене человека NEK2_HUMAN (AC в Swiss-Prot: P51955), выбранном ранее.
Попытка найти последовательность гена этого белка, используя сервис "BLAST/BLAT", оказалось неудачной. Вместо выдачи была получена вот такая страница:
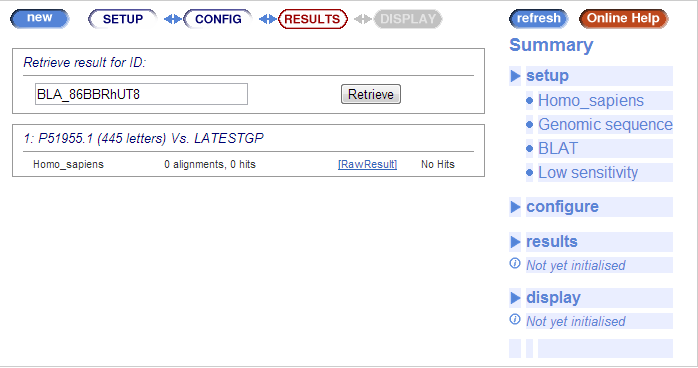
|
|
Рисунок 1. Выдача BLAST/BLAT.
|
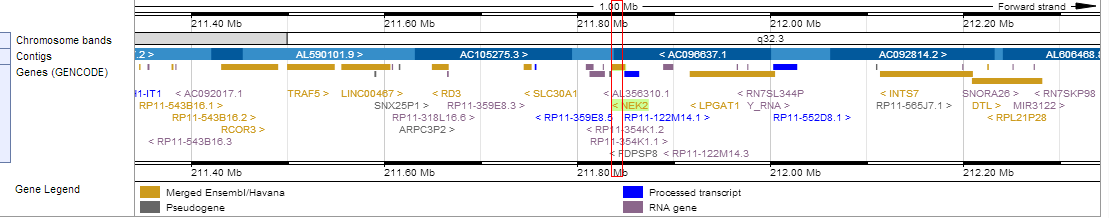
Cо страницы, посвященной гену была открыта страница Region in detail. Некоторая информация раздела выдачи Region in detail (Рис. 2-5):
- Ген находися на 1 хромосоме, координаты гена: 211,836,114-211,848,960. Красной рамочкой обведен участок хромосомы, в котором находится ген (Рис. 2).
- Mожно выделить красной рамочкой любой участок данной хромосомы, тогда информация о "регионе" отобразится ниже.

|
|
Рисунок 2. Расположение гена NEK2 в хромосоме 1.
|
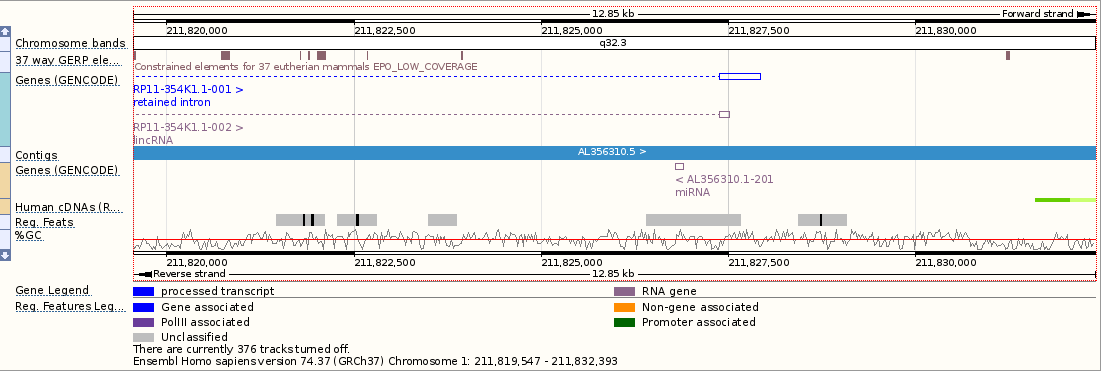
На рисунке 3 представлен "регион", в котором расположен данный ген.
- В строке под названием "Contigs" указаны рядом лежащие гены: АС105275.3 и AC096637.1.
- В строке "Genes" фиолетовым показаны гены, кодирующие РНК; синим - транскрипты, прошедшие процессинг; серым - псевдогены.
- Можно менять параметры выдачи Region in detail: масштаб, проводить поиск только экзонам гена, делать выравнивания с соответствующими участками хромосом разных животных и т.д.
|
|
Рисунок 3. Ген NEK2 в хромосоме 1 и его окружение.
|
|
Рисунок 4. Выровненные гены NEK2 из разных баз.
Серым обозначены гены, кодирующие РНК; синим - транскрипты, прошедшие процессинг.
|

|
Рисунок 5. Cхема расположения интронов и экзонов в гене NEK2 по различным данным.
Красным показаны гены, кодирующие белки; синим - транскрипты, прошедшие процессинг.
|
Кроме выдачи Region in detail есть и другие разделы в EnsEMBL. Например, в разделе "Chromosome summary" можно найти информацию о генах в разных участках хромосомы в целом; в разделе "Whole genome" есть информация о всем геноме.
© Novikova Maria, 2013
Последнее обновление: 17.12.2013





