
На предыдущем занятии методом Neighbour Joining было построено 2 филогенетических дерева. Деревья, реконструированные этим методом, являются неукоренёнными, в отличие от деревьев, реконструированных методом UPGMA (Average Distance в программе JalView).
Чтобы укоренить в среднюю точку дерево, построенное методом Neighbour Joining, можно воспользоваться программой retree пакета PHYLIP. Ниже приведены изображения соответствующих укоренённых деревьев, полученные программой MEGA.
Алгоритм действий:
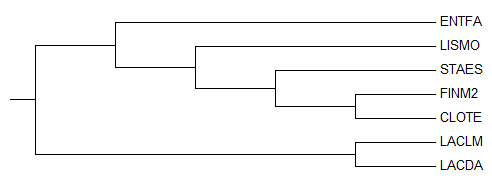
Ниже представлено исходное реконструированное дерево, полученное с помощью программы JalView методом Neighbour Joining Using % Identity, и укоренённое в среднюю точку дерево. Ветвь, в которую произошло укоренение, обозначена на изображении исходного дерева стрелкой. Данное укоренение вряд ли можно считать верным относительно правильного дерева, наиболее приемлемым можно назвать укоренение в тривиальную ветвь {CLOTE,FINM2}.
 |
 |
| Исходное дерево | Укорененное дерево |
Метод максимальной экономии (Maximum parsimony) не реконструирует длины ветвей. Поэтому деревья, построенные этим методом, невозможно укоренить в среднюю точку. Однако можно воспользоваться укоренением с помощью внешней группы.
В качестве внешней группы для реконструкции дерева отобранных бактерий методом максимальной экономии использован белок семейства hslo из кишечной палочки Escherichia coli (hslo_ecoli). Последовательности белков отобранных бактерий (Firmicutes) и последовательность белка кишечной палочки были выровнены вместе: Web Service > Alignment > Muscle with Defaults в JalView, после чего результат выравнивания был импортирован в программу MEGA, при этом имена последовательностей были отредактированы (оставлена только мнемоника видов). Укоренили построенное методом максимальной экономии дерево: Maximum Parsimony в меню Phylogeny, в ветвь, ведущую к ECOLI (можно с помощью команды Subtree > Root с последующим выбором соответствующей ветви). Для получения изображения укоренённого дерева без ECOLI воспользовались кнопкой Show Subtree Separately на левой панели окна MEGA.
 |
| Maximum parsimony с использованием внешней группы |
Полученное укоренение можно считать относительно правильным. Реконструированное методом максимальной экономии с использованием внешней группы дерево отличается от правильного ветвями:
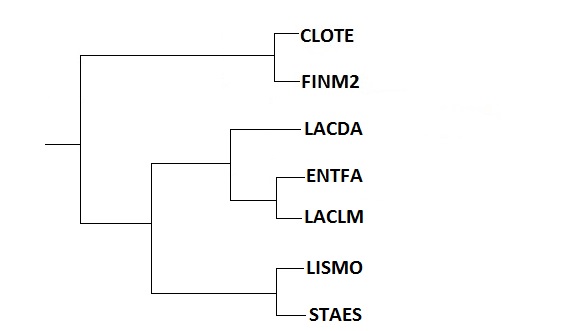
Бутстрэп-анализ филогении белков отобранных бактерий можно провести с использованием одного из методов, доступных в программе MEGA. Например, ниже приведены изображения деревьев, полученные в результате бутстрэп-анализа белков с использованием метода UPGMA (число реплик было установлено равным 100). Как видно, деревья, названные Original tree и Bootstrap consensus tree, очень похожи. Однако оригинальное дерево имеет ветвь {LACDA, ENTFA, LACLM}, которой нет у построенного.
 |
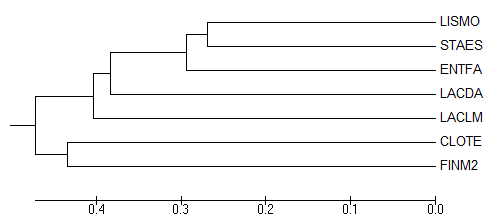 |
| Original tree | Boostrap consensus tree |
Ветвь {FINM2, CLOTE, LACLM} vs {LACDA, LISMO, STAES, ENTFA} имеет относительно высокую поддержку (по сравнению со значениями для других ветвей), хотя верной не является.