Я выбрала E2F/DP семейство ДНК-связывающих доменов (E2F_TDP, AC PF02319, (ссылка на страницу на сайте Pfam)). Это семейство содержит транскрипционный фактор E2F и его димеризационных партнеров TDP1 и TDP2, которые стимулируют E2F-зависимую транскрипцию. E2F связывается с ДНК в виде гомодимера или в виде гетеродимера в сочетании с TDP1/2, при этом гетеродимер увеличивает эффективность связывания. Узнавание центральной последовательности с/gGCGCg/с на ДНК-связывающего сайте симметрично, и аминокислоты, которые контактируют с этими основаниями, консервировативны среди всех известных E2F и DP белков.
Запустила JalView. Для выбранного семейства доменов скачала выравнивание из Pfam в JalView. Раскрасила по консервативности (ClustalX, By conservation порог 30). Добавила 3D структуру домена (1CF7) к последовательности E2F4_HUMAN. Сохранила выравнивание как проект 1.jar и в fasta-формате.
Для дальшейшей работы выбрала две доменные архитектуры: PF02319 с одним доменом E2F (рис. 1) и PF08781 - транскрипционный фактор DP (рис. 2).
| Рисунки 1 и 2. Выбранные домены Pfam. | |
Получила таблицу с информацией об архитектуре всех последовательностей, содержащих выбранный домен. Составила список последовательностей с указанием доменной архитектуры. Использовала сводную таблицу в Excel: строки – AC последовательностей, столбцы – домены Pfam. В список последовательностей добавила колонки с информацией о таксономической принадлежности.
Далее выбрала в качестве таксона Eukaryota, а в качестве подтаксонов - Alveolata, Metazoa и Viridiplantae. Выбрала по 25 последовательностей из каждой доменной архитектуры. Оставила только отобранные последовательности из всех выбранных архитектур с помощью скрипта filter_alignment.py .
Открыла "профильтрованное" выравнивание в JalView, удалила пустые колонки, создала группы по архитектурам, задала раскраску в каждой группе (ClustalX, By conservation порог 10),добавила разметку по вторичной структуре, исходя из последовательности с известной 3D структурой, удалила фрагменты и последовательности, которые выровнены явно неправильно, а также маленькие гэпы на месте спиралей и тяжей, составляющих ядро домена, удалила N- и C-концевые участки. Ниже привожу изображение полученного выравнивания (рис.3) и ссылку на него в формате .jar.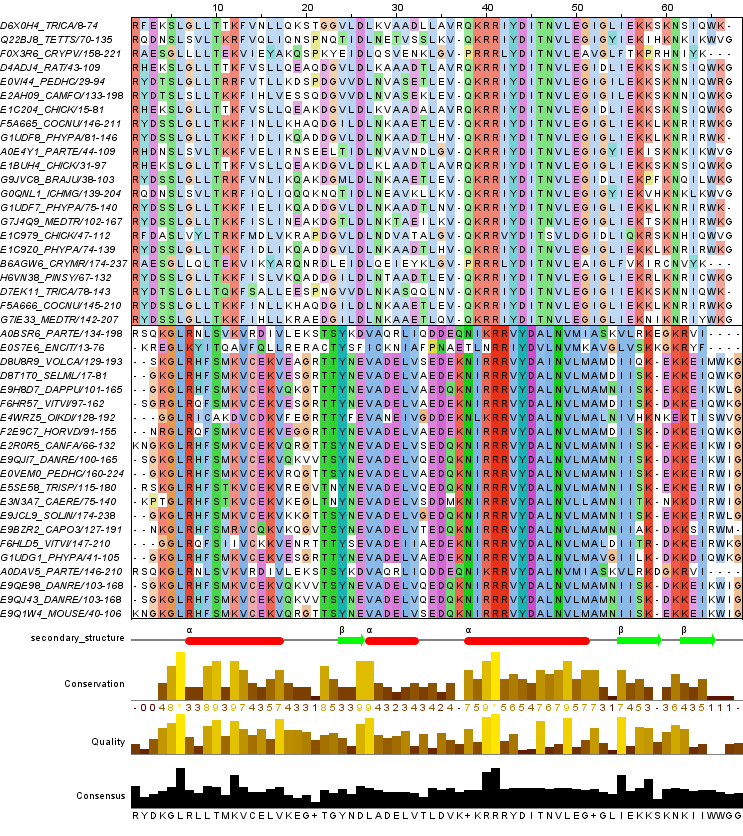 |
| Рисунок 3. Изображение полученного выравнивания. |
Из выравнивания видно, что позиции, соответствующие альфа-спиралям и бета-тяжам, сильно консервативны. Последовательности из второй группы консервативнее, чем из первой.