
Сделала имена последовательностей из практикума 9 говорящими: закодировала доменные архитектуры (1 для однодоменного белка E2F, 2 для двухдоменного E2F+DP) и сравниваемые таксоны (A - Alveolata, V - Viridiplantae, M - Metazoa). К именам всех последовательностей спереди добавила коды архитектуры и таксона.
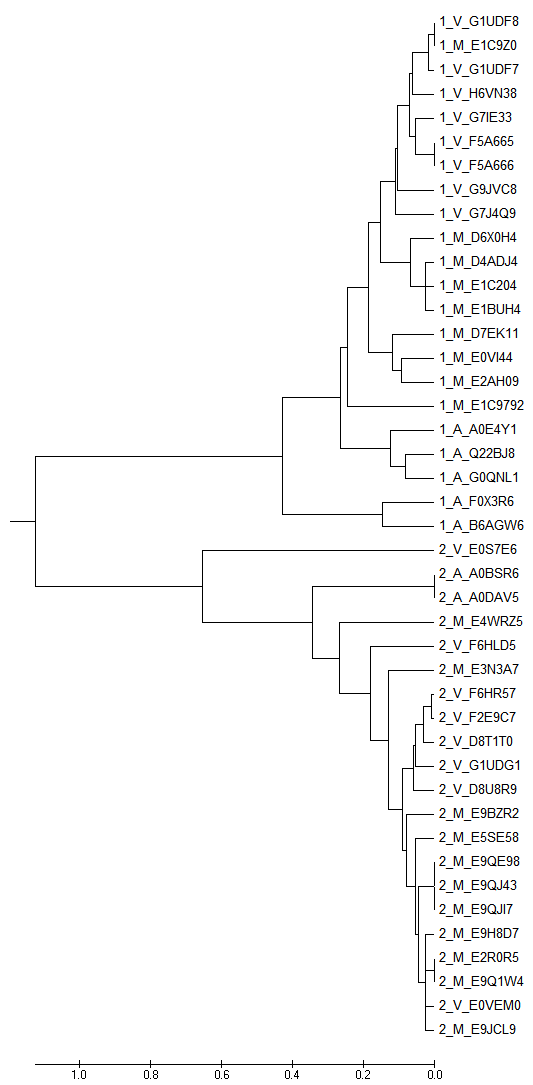
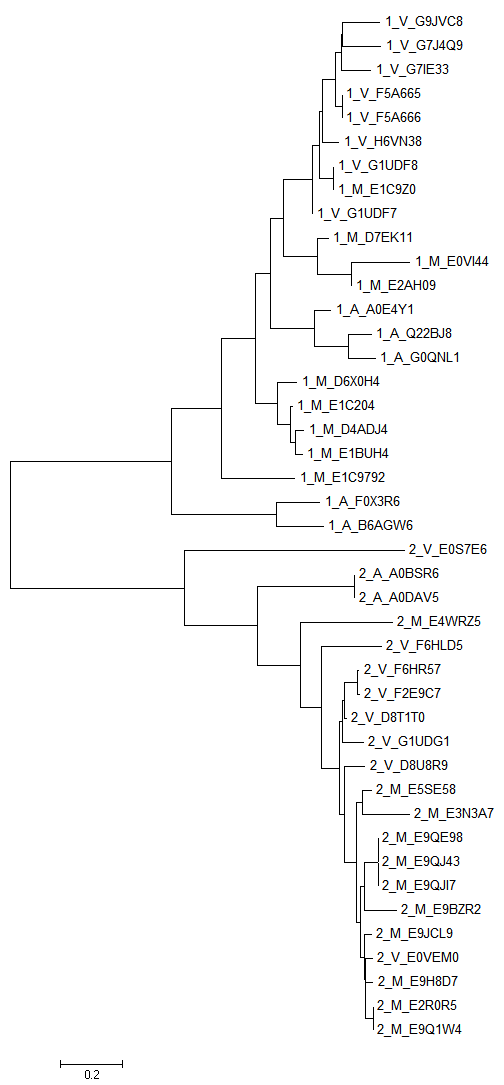
Построила филогенетическое дерево: использовала программу MEGA и методы UPGMA (Рис. 1 и 2) (предполагает молекулярные часы, строит укоренённое дерево) и Neighbor-Joining (Рис. 3 и 4)(не использует гипотезу о молекулярных часах и строит неукоренённое дерево). Были использованы параметры по умолчанию.
 |
 |
| Рисунки 1 и 2. Деревья, построенные методом UPGMA. Справа - топология дерева. | |
 |
 |
| Рисунки 3 и 4. Деревья, построенные методом Neighbor-Joining. Справа - топология дерева. | |
Создала рисунок дерева с использованием методов выделения клад для наглядной демонстрации результата с помощью программы ITOL (рис. 5). Здесь прослеживается эволюционно четкое разделение на две архитектуры.
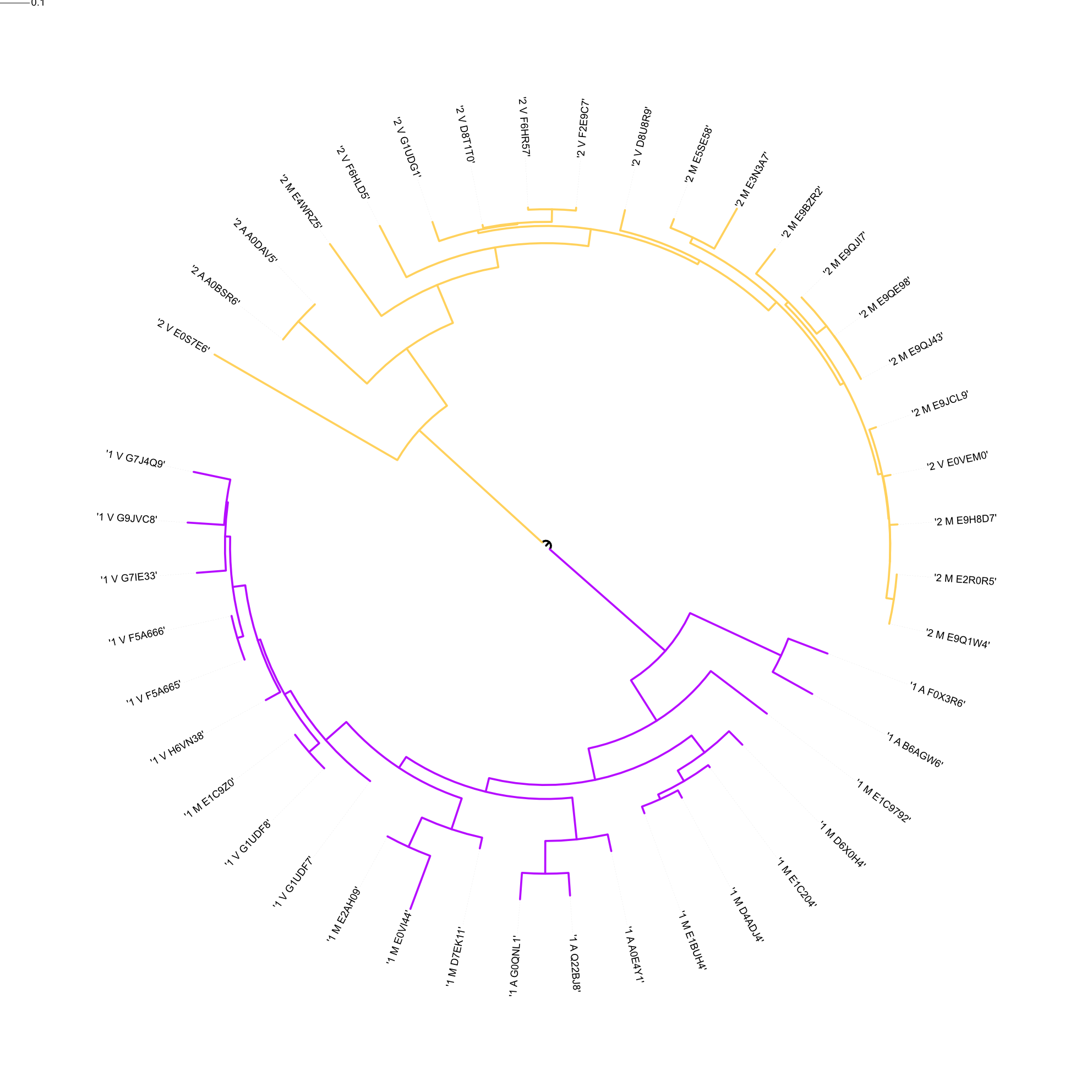 |
| Рисунок 5. Дерево, построенное с помощью программы ITOL. Желтым выделена ветвь двудоменного белка, фиолетовым - однодоменного. |
Скобочные формулы деревьев, построенных методом NJ: формат Newick с длиной ветвей и только топология.
Из полученных данных можно сделать предположение, что выделение двух рассматриваемых доменных архитектур произошло до разделения эукариот на царства. Дальнейшая эволюция доменов происходила сравнительно недавно и шла быстрее у двудоменных белков.