
Для определения вторичной структуры белка 1G6C были использованы программы DSSP и Stride. Границы спиралей и тяжей в выдаче программ (DSSP, Stride) оказалась близки к таковым, указанным в pdb-файле модели структуры 1G6C (рис. 1.1).
 |
| Рисунок 1.1. Mодель структуры 1G6C (показана только цепь А); α-спирали обозначены розовым цветом, β-листы – голубым цветом |
Программа DSSP определила границы α-спиралей в структуре следующим образом (для цепи A; для цепи B результаты аналогичны): 19-25, 41-51 (приведены номера аминокислотных остатков, обозначающих границы элемента). Результаты программы Stride отличаются лишь в границе одной спирали: 19-26, 41-51. Полученные границы можно сравнить с границами аннотации вторичной структуры в pdb-файле: 18-26, 40-52. Аналогичное сравнение можно проделать для определённых β-тяжей. Соответствующие границы приведены в таблице 1. Видно, что границы β-тяжей, определённые программами, в точности соответствуют таковым в pdb-файле, а предсказание границ α-спиралей меньше соответствует pdb-файлу. При этом алгоритмы DSSP и Stride показали сравнимое качество предсказания, а отличие каждой границы неверного предсказания от границы в pdb-файле составляет один аминокислотный остаток.
Таблица 1. Сравнение результатов определения элементов вторичной структуры программами DSSP и Stride для цепи A модели структуры 1G6C
| № элемента вторичной структуры |
Результат DSSP (номера остатков) |
Результат Stride (номера остатков) |
Аннотация в pdb-файле (номера остатков) |
α-спираль | |
| 1 | 19-25 | 19-26 | 18-26 | ||
| 2 | 41-51 | 41-51 | 40-52 | ||
| β-лист | |||||
| 1 | 28-32 | 28-32 | 28-32 | ||
| 2 | 55-58 | 55-58 | 55-58 | ||
С помощью SheeP была построена карта β-листа в цепи A структуры 1G6C (были использованы параметры по умолчанию) (рис. 1.2). По карте видно, что выбранный β-лист состоит из восьми тяжей. Изображение его в структуре приведено на рисунке 1.3. Один столбец карты соответствует одному «хребту» (crest) β-листа. Один из таких «хребтов», соответствующий аминокислотам в первом (продолжение в девятом) столбце карты (остаткам Ser27, Gly204, Val184, Gly149, Ala130, Gly109), показан на рисунке 1.4. Посмотрев на расположение активного центра, можно сделать вывод, что "желтой" стороной β-лист обращен к гидрофобному ядру.
 |
| Рисунок 1.2. Карта β-листа для структуры 1G6C |
Для участка β-листа (3 тяжа, 4-5 гребней) (рис. 1.5) на основании данных программы stride была получена схема водородных связей (рис. 1.6).


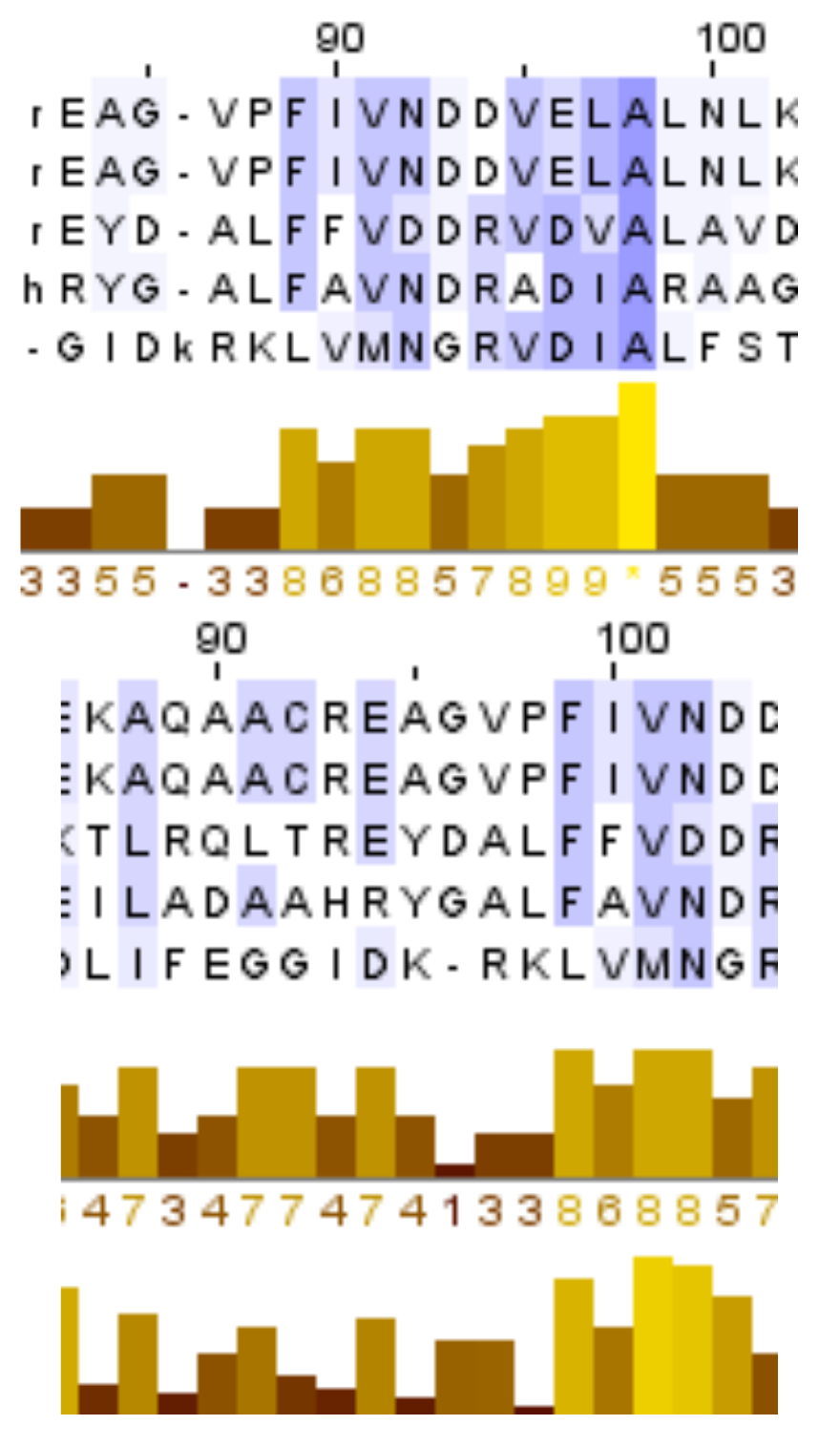
С помощью сервиса PDBeFold были выбраны 4 структурных гомолога 1G6C: 3O16, 1XI3, 3O63, 1YAD. Для гомологов 1G6C и самого белка были загружены выравнивание последовательностей по совмещению структур (попарное и множественное) и само совмещение структур (рис. 2.1).

Выравнивание по структуре (большие буквы в файле с выравниванием) можно сравнить с выравниванием последовательностей, выполненным, например, в программе JalView (с помощью Tcoffee со стандартными параметрами) (рис. 2.2-2.3).

При сравнении множественного выравнивания последовательностей с выравниванием последовательностей по структуре удаётся выявить несколько различий. Например, в выравнивании по последовательности Glu94 выравнивается с Gly94, в то время как в в выравнивании по структуре вместо Gly94 мы видим Asp94. Можно отметить, что все найденные несоответствия наблюдаются именно в неструктурированных участках (рис. 2.4).
С помощью сервиса PDBeFold был осуществлен поиск стрктурных гомологов для домена 1adj C:326-419, однако искомый домен найден не был. На рисунке 2.5 показана выдача с сайта. Результаты отсортированы по RMSD.

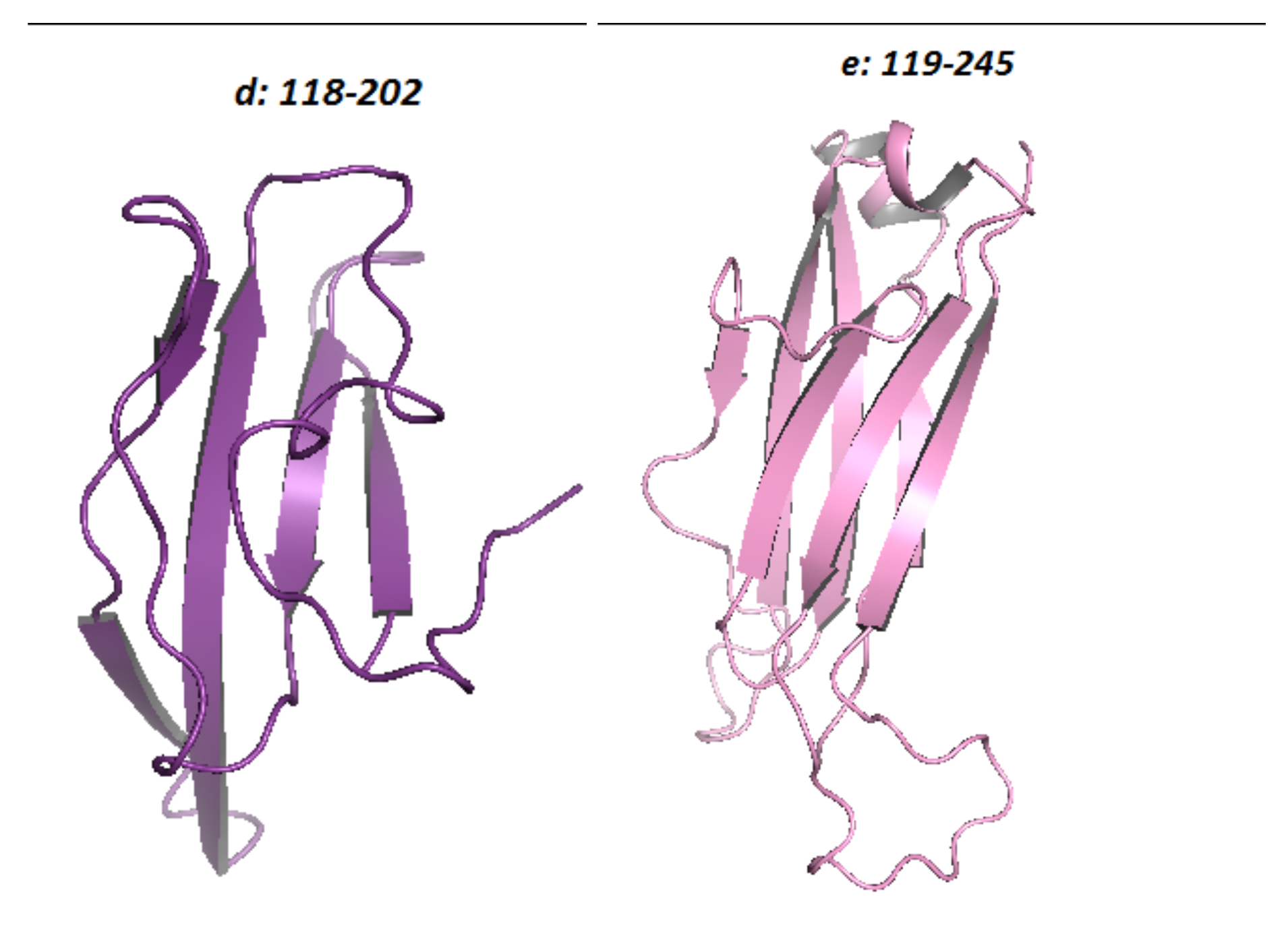
Для совмещения по заданному выравниванию были выбраны структуры константного домена человеческого Т-клеточного рецептора из цепи α (1oga,region d: 118-202) и из цепи β (1oga, region e: 119 - 245) (рис. 2.6).
С помощью сервиса SheeP были получены карты β-листов доменов (рис. 2.7-2.9). Карты приведенных ниже листов находятся в одной ориентации.

Рисунок 2.7. Карта бета-листа для домена из цепи α (map0).
Консервативные остатки цистеина задают выравнивание центрального тяжа. Остатки, спаренные с консервативным цистеином, задают выравнивание соседних тяжей. Таким образом было построено выравнивание. Используя информацию о выровненных остатках, в программе PyMol было построено совмещение этих доменов (рис. 2.9), в частности с помощью следующих команд:
select a, alpha and name CA and resi (122+133-135+155+175) select b, beta and name CA and resi (127+144-146+172+192) pair_fit a, b
Также было получено изображение участков совмещенных структур (рис. 2.10). Из рисунка видно, что ход полипептидной цепи совпадает.

Рисунок 2.10. Совмещение доменов T-клеточного рецептора. Цепь α - фиолетовым; β - серым.
С помощью сервиса Clud в структуре 1G6C был произведён поиск гидрофобных кластеров. Со значениями 5.4 и 3 для порога расстояния и размера кластера, соответственно, было найдено 6 кластеров, которые обозначены на рисунке 3.1.
 |
| Рисунок 3.1. Гидрофобные кластеры в структуре 1G6C |
Пожалуй, можно сказать, что найденные кластеры отвечают группам (с одним и более) элементов вторичной структуры белка, определяемым визуально.
Поиск гидрофобных кластеров в димере белка 3Q8T (цепях A и B структуры с мотивом coiled coil) позволяет различить гидрофобные кластеры в участках контакта двух спиралей (значения для порога расстояния и размера кластера – 4.5 и 3, соответственно) (рис. 3.2).
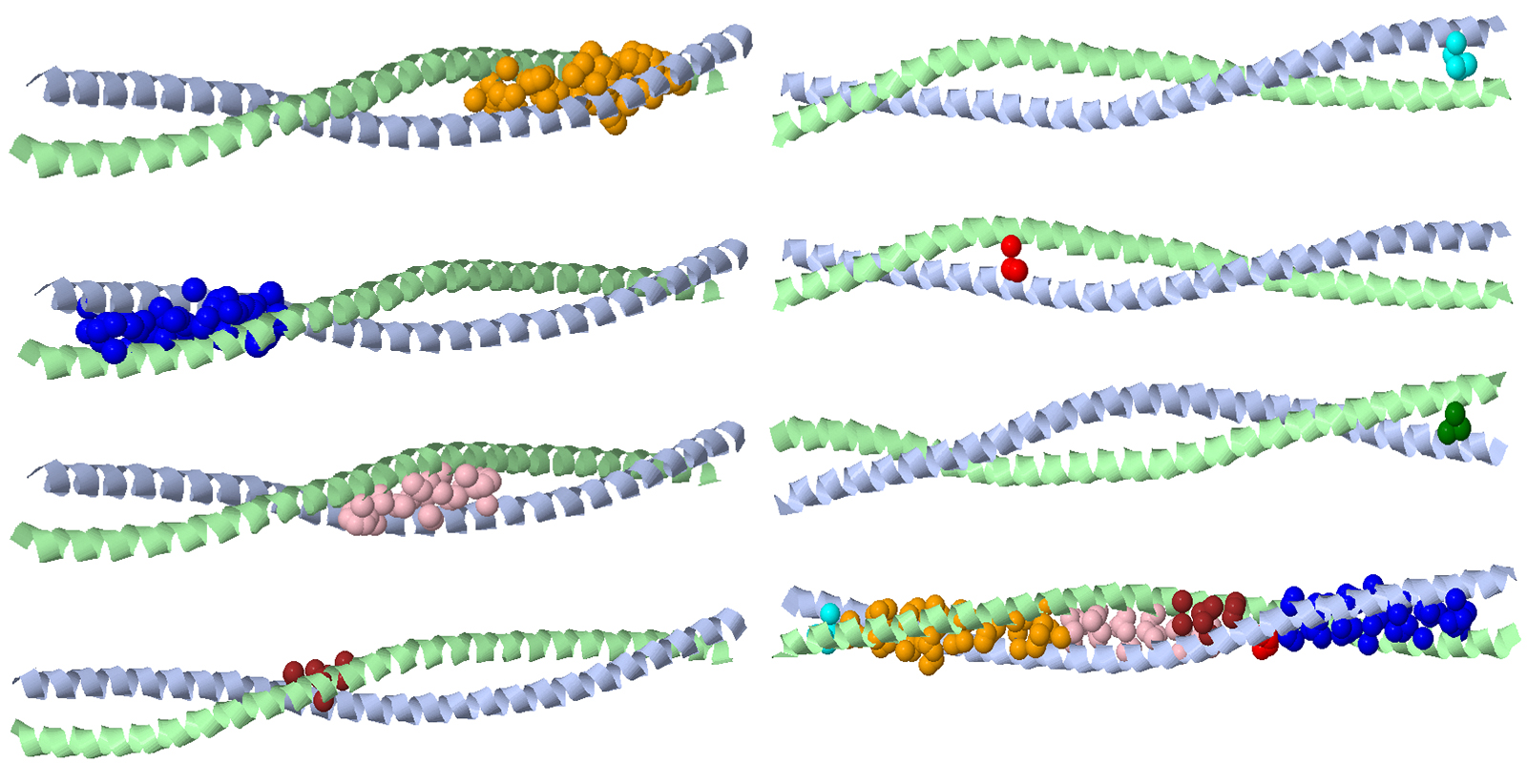 |
| Рисунок 3.2. Гидрофобные кластеры в участках контакта спиралей в структуре 3Q8T |
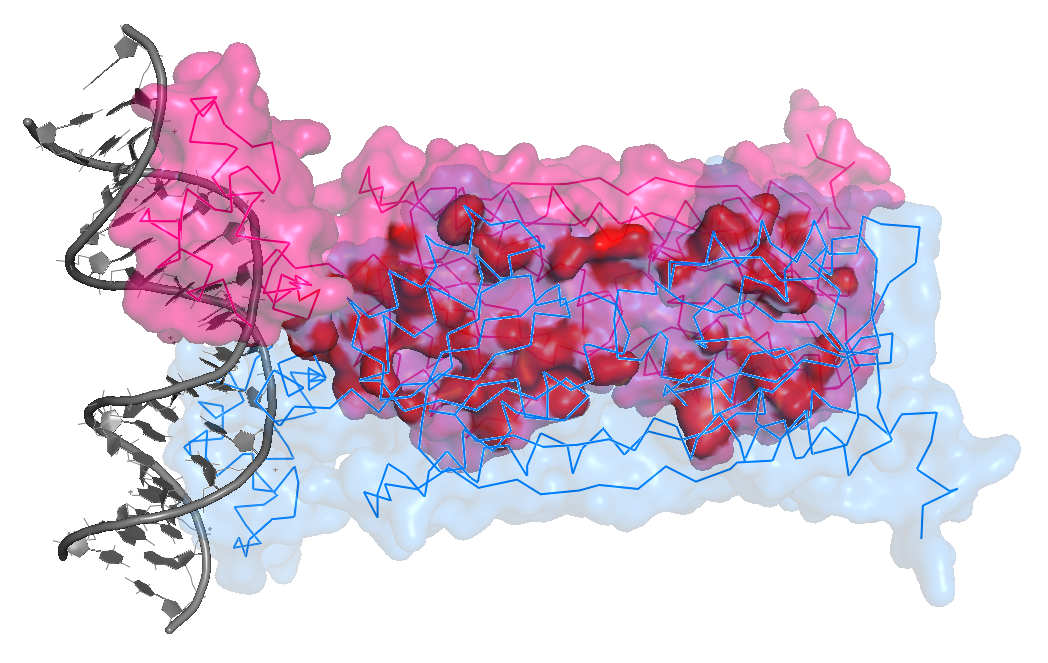
Для комплекса димера пуринового репрессора с ДНК (биологическая единица 1JFT) с использованием средств программы PyMol были созданы изображения, демонстрирующие контакт мономеров белка и между собой и контакт белка с ДНК. Контакты поверхности мономера белка с симметричным мономером и с двойной спиралью ДНК представлены на рисунках 4.1-4.3.

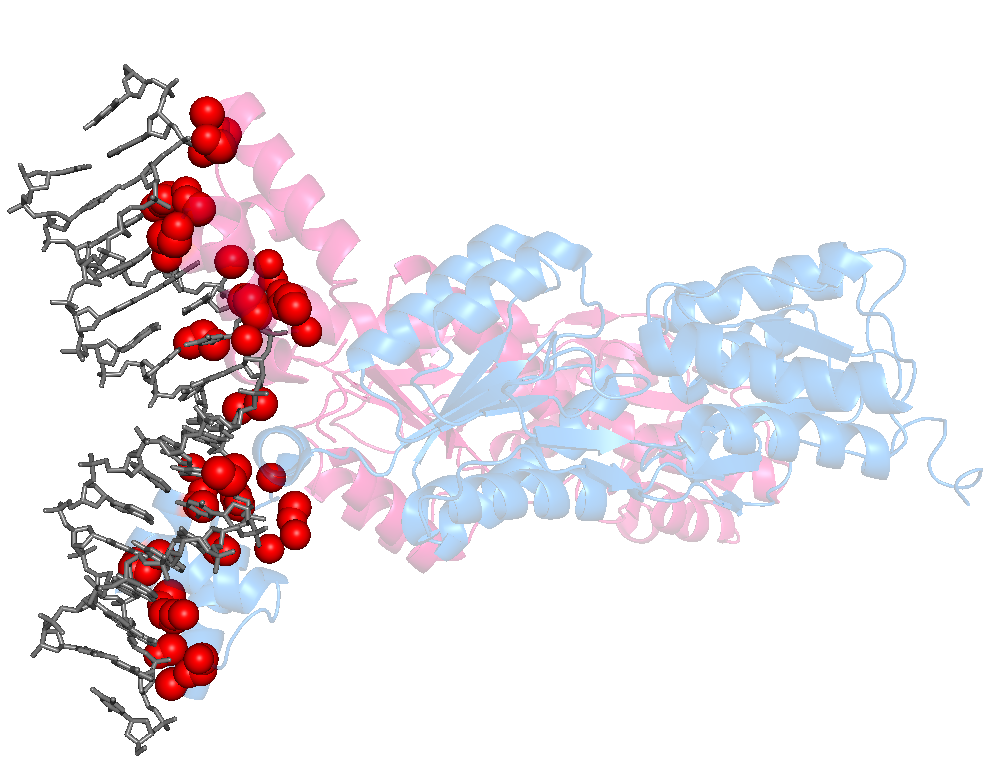
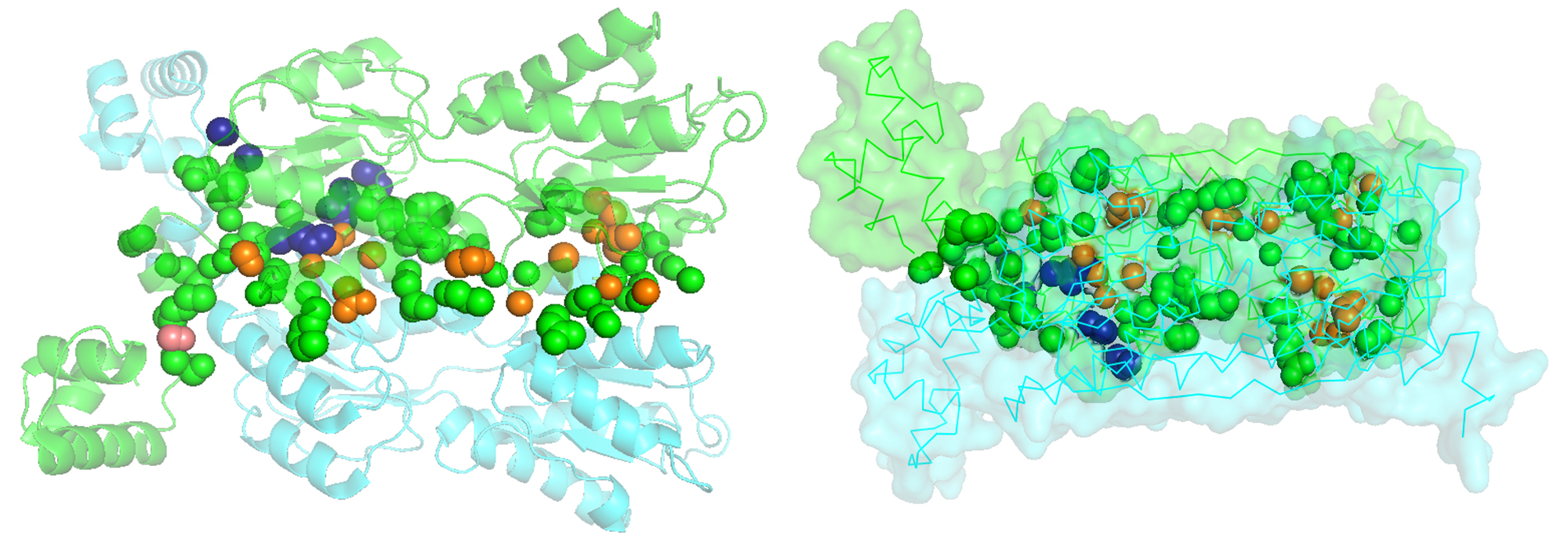
С помощью сервиса Clud в структуре 1JFT были определены гидрофобные кластеры объёмом не менее 10 атомов на интерфейсе мономеров белка (distance threshold – 5 Å) (рис. 4.4).
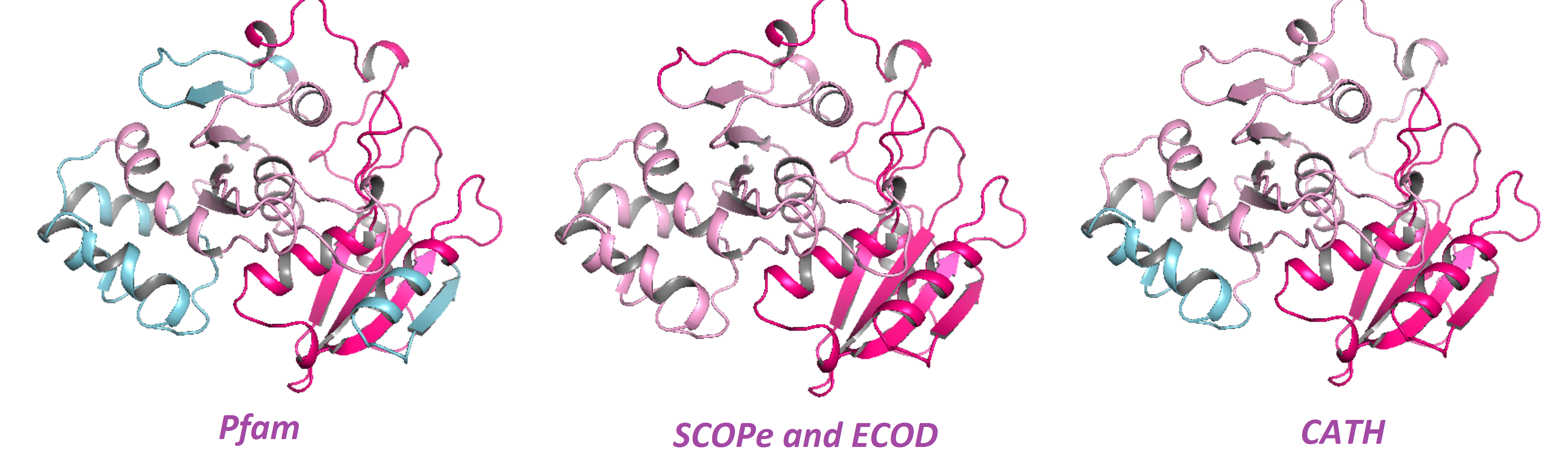
Для структуры 1CTT был произведен поиск доменов в Pfam, SCOPe, ECOD, CATH.
Структура 1CTT (TRANSITION-STATE SELECTIVITY FOR A SINGLE OH GROUP DURING CATALYSIS BY CYTIDINE DEAMINASE) соcтоит из субъединицы A. Указанные ниже домены, найденные для цепи A, довольно хорошо коррелируют друг с другом. Подробная информация о координатах доменов представлена в таблице 2.
Таблица 2. Координаты доменов SCOPe, ECOD, CATH и Pfam для цепи A модели структуры 1CTT
| Сервис | Координаты домена 1 | Координаты домена 2 |
| Pfam | 48-142 dCMP_cyt_deam_1 |
157-277 dCMP_cyt_deam_2 |
| SCOPe | 1-150 d1ctta1 |
151-294 d1e0ta3 |
| CATH | 29-179 1cttA01 |
180-294 1cttA02 |
| ECOD | 1-150 e1cttA1 |
151-294 e1cttA2 |
В результате поиска с использованием параметра Experimental Method: ELECTRON MICROSCOPY было найдено 917 структур. Скачанные последовательности белков представлены в FASTA файле.
При поиске структурных гомологов программой jFATCAT в PDB для структуры 1G6C, к сожалению, ни одного гомолога не было обнаружено. Поэтому была взята сходная структура того же белка - 2TPS. В PDBeFold было найдено 2258 записей, в то время как в jFATCAT с порогом на Р-value < 0.001 - 444. Всего лишь 62 записи из PDBeFold найдены в jFATCAT.
Список из PDBeFold: reslist.dat
Список из jFATCAT в PDB: jFATCAT.xlsx