EnsEMBL
1)
Я начал поиск в BLAT наиболее длинного экзона белка CLIC1. Для этого я прошел по ссылке BLAST/BLAT,
там я ввел нуклеотидную последовательность экзона и начал BLAT поиск в геноме человека.
В итоге я нашел несколько вариантов выравнивания с геномом, лучшим из которых было выравнивание с участком 6-й хромосомы,
который соответствует гену CLIC1 (тоже в 6-й хромосоме).
Тот факт, что было найдено 4 выравнивания, с различными участками хромосомы, означает, что либо наш экзон был недостаточной длины для точной
идентификации (скорее всего нет, т.к. длина экзона была примерно 180 нуклеотидов), либо такие же экзоны встречаются в генах, кодирующих гомологичные белки.
Под представленным рисунком, расположены окна, в которых, схематично указаны хорошо варавненные участки запроса.
Ниже расположена таблица со всеми обнаруженными фрагментами, в которой можно посмотреть:
Выравнивание, Начало и конец по последовательности хромосомы, расположение контигов, E-Value, Score, %ID, Длину и т.д.
Мое выравнивание:
Query location : exon 1 to 182 (-)
Database location : 6 31699994 to 31700175 (+)
Genomic location : 6 31699994 to 31700175 (+)
Alignment score : 912
E-value : 2.0e-153
Alignment length : 182
Percentage identity: 100.00
Query: 182 ctgtactatgtgtaactttggcaacaggttgcagtcagccagggtgagctcgttgccatc 123
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
Sbjct: 31699994 ctgtactatgtgtaactttggcaacaggttgcagtcagccagggtgagctcgttgccatc 31700053
Query: 122 caaaaacttcctctgagagacaccttcatcttcagcactggtttcatccacttcttctgg 63
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
Sbjct: 31700054 caaaaacttcctctgagagacaccttcatcttcagcactggtttcatccacttcttctgg 31700113
Query: 62 gaggggggatgttaagtaattgtctaaaaccttcagggctttcaggagtcccttctccag 3
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
Sbjct: 31700114 gaggggggatgttaagtaattgtctaaaaccttcagggctttcaggagtcccttctccag 31700173
Query: 2 at 1
||
Sbjct: 31700174 at 31700175
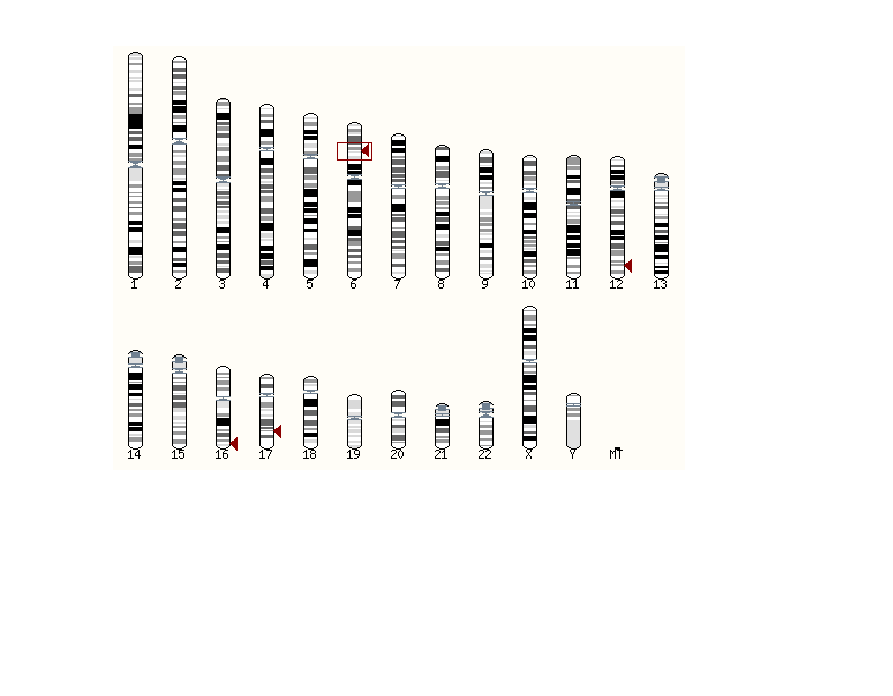
Пройдя по ссылке (Contig view) мы наблюдаем несколько схем:
В верху страницы есть схема, указывающая, как я понял, различные гаплотипы найденного участка в поле Ensemble exceptions.
Кроме того, на этой схеме двумя вертикальными красными полосами обозначены границы экзона (на картинке нет) в гене.
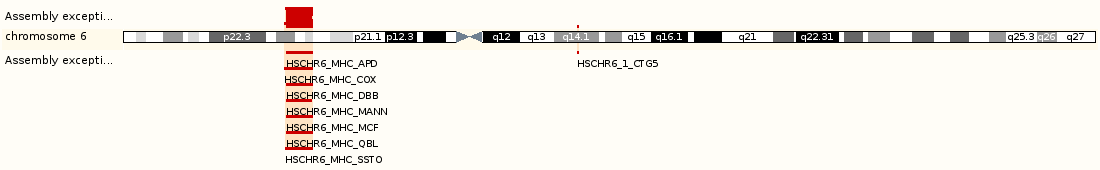
Далее в схеме Region in detail подробно описана структура фрагмента:
Синим цветом обозначены контиги, из которых складывается данный фрагмент. Contig, в который попадает мой ген- AL662899.5.
Надо заметить, что при переходе по ссылке (Contig view) мы перешли к рассмотрению более широкого фрагмента генома (+ 2000 нукл. с каждой стороны),
поэтому в данном фрагменте встречаются и др. гены и нкРНК.
На этой же схеме в разных участках фрагмента, прямоугольниками разных цветов обозначались:
1)Желтым цветом обозначены гены, описанные группой ученых HAVANA (специализирующихся на описании генов позвоночных)
и добавленные в EnsEMBL. Кстати, гены выделенные желтым, определены наиболее точно (почти все выделены, как Known protein coding)
Среди таких генов был и мой ген.
2)Серым - псевдогены
3)Красным - участки кодирующие белки
4)Синим - полученные транскрипты с неопределенными функциями
5)Бледно-фиолетовым - некодирующие РНК.
*Все это есть в поле Gene Legend
Как и следовало ожидать, найденный экзон попал на ген CLIC1.

В третьем окне представлен сильно увеличенный участок Contig-а, с выровненными экзонов с участком генома (экзоны выделены
прямоугольниками). По этому участку, мы видим координаты кодирующей части гена. Красным прямоугольником сверху, обозначен
наш экзон, с помощью которого осуществлялся поиск.
2)
UCSC показался более подробным, т.к. снизу приведен целый перечень того, что можно открыть или скрыть на схеме. Также в UCSC
легче выйти на выравнивание участка, просто кликнув на него (+ не смотря на само выравнивание видно по картинке, какие участки
хорошо варавненны, а какие нет (плохие выделены цветом на прямоугольнике)). Из недостатков, надо заметить, что UCSC менее понятен
чем EnsEMBL.
NCBI имеет довольно странное и малопонятное оформление
Vega очень похожа на EnsEMBL, только чуть более простая (как мне показалось, менее функциональная).
Главная страница (см. ниже)
©Abdullaev Eldar


