BLAST
Поиск гомологов белка
Был выполнен поиск гомологов субъединицы CarD комплекса кофеил-КоА-редуктазы и Etf из генома бактерии Acetobacterium woodii DSM 1030. Результаты оказались не столь впечатляющими, как представлялось изначально, но прежде чем перейти к их скудному обсуждению, лучше разобраться в параметрах Blast.
DATABASE: база данных, по которой ведется поиск
ORGANISM: организм, в котором ведется поиск; можно выбрать несколько
ALGORITHM: алгоритм поиска; существует быстрая версия, которая дает существенный выигрыш во времени (кажется, она плохо работает для малых identity, так что ее стоит использовать только для поиска очень близких белков); также есть delta-blast, который использует дополнительные ресурсы NCBI и ищет консервативные домены - поиск происходит быстрее; еще присутствуют psi- и phi-blast, которые строят позиционную матрицу, но немного разными способами
MAX TARGET SEQUENCES: максимально допустимое количество последовательностей в выдаче, после которого алгоритм останавливается
EXPECT THRESHOLD: максимально допустимое значение e-value, которое характеризует математическое ожидание появления выравнивания с таким же или большим весом в банке из такого же количества случайных последовательностей той же длины; e-value=m*n*2-B, где m - длина последовательности, n - размер базы данных, B - нормализованный вес в битах (фактически нужно построить 2B случайных последовательностей, чтобы получить одно с таким же весом или лучшим)
WORD SIZE: длина индексированных слов, на которые разбиваются все последовательности в базе данных; на основе этого составляется таблица сходства и выбираются последовательности для последующих итераций
MAX MATCHES IN A QUERY RANGE: ограничение на количество совпадений в заданной области последовательности; используется для избегания ситуации, когда более консервативный участок "затмевает" остальные, мешая алгоритму найти слабые совпадения
MATRIX: матрица сходства аминокислотных остатков, используемая для подсчета веса выравнивания
GAP COSTS: величина штрафа за появление гэпа и удлинение инделя
COMPOSITIONAL ADJUSTMENTS: улучшения матрицы, основанные на аминокислотном составе анализируемых последовательностей; например, веса могут масштабироваться с использованием определенных констант
FILTER: маскировка статистически значимых, но биологически неинтересных участков последовательности; также есть опция, маскирующая специфические повторы в человеческих последовательностях
MASK: for lookup only используется для маскировки исключительно в процессе составления оригинальной таблицы сходства, тогда как расширения производятся по полной последовательности; еще можно использовать нижний регистр, чтобы выделить маскированные области
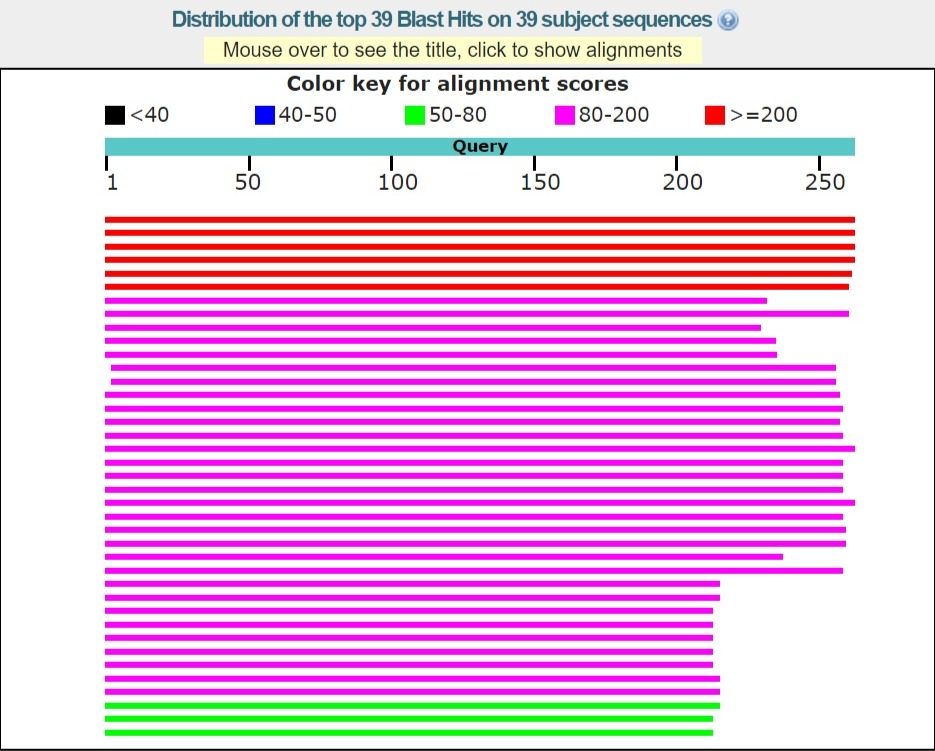
В результате работы алгоритма с единственным измененным параметром (количество результатов поиска - 20000) было найдено всего лишь 39 белков (рис. 1). При этом стоит заметить, что самый первый из них - та же запись, по которой производился поиск. Судя по всему, это довольно редкий белок, что неудивительно. Все результаты разбиваются на две группы: флавин-зависимые переносчики электронов и белки FixA с разнообразными окислительно-восстановительными функциями. Таблица находок была сохранена в директории block3/credits.
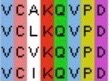
Для выравнивания были выбраны изначальный белок, еще три белка-переносчика электронов, один предположительный переносчик электронов и два белка FixA. Дальнейшая работа в Jalview привела к некоторым неожиданным результатам. Предположительно гомологичными являются первые четыре выбранные последовательности с идентификаторами H6LGM7.1, P52040.2, G3KIM7.1, P53577.2. Всего лишь три из этих белков являются флавин-зависимыми переносчиками электронов, однако если убрать белок FixA, картина не сильно меняется в лучшую сторону, так что я решила его оставить. Участки, свидетельствующие о гомологии, находятся в начале последовательности и ее середине. Первый (рис. 2) из них состоин из восьми аминокислот с 5-ой по 12-ую и содержит семь консервативных позиций. Несмотря на то, что седьмой остаток отличается у всех организмов, четыре представленных аминокислоты являются алифатическими, к тому же три из них (валин, лейцин, изолейцин) получаются друг из друга точечной мутацией первого нуклеотида кодона. Второй участок (рис. 2) состоит из тринадцати остатков с одиннадцатью консервативными позициями. Кроме того, аргинин и лизин оба являются положительно заряженными аминокислотами (лизин мог являться результатом точечной мутации). Ко всему этому можно добавить еще и тот факт, что у изначального белка в этом месте находится сайт связывания водорода - то есть у этих белков скорее всего (для остальных протеинов данных нет) совпадают аминокислоты в активном центре.
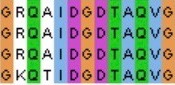
В общем у этих четырех белков 64 полностью консервативные позиции и 69 позиций, консервативных на 75%. Это довольно много, и первые три белка с уверенностью можно назвать гомологичными - у них похожие названия и функции. Хотелось бы присовокупить к ним же и белок FixA - из-за гомологичного участка, включающего сайт связывания водорода. При этом все четыре фермента имеют разные названия.
Карта локального сходства
Для построения карты (рис. 3) были выбраны два протеина: белок синтеза фолиевой кислоты (Q54YD9) и 7,8-дегидронеоптерин альдолазы (E6SLJ6). Blast строит шесть отдельных выравниваний для этих двух последовательностей (от 149 до 14 аминокислот).
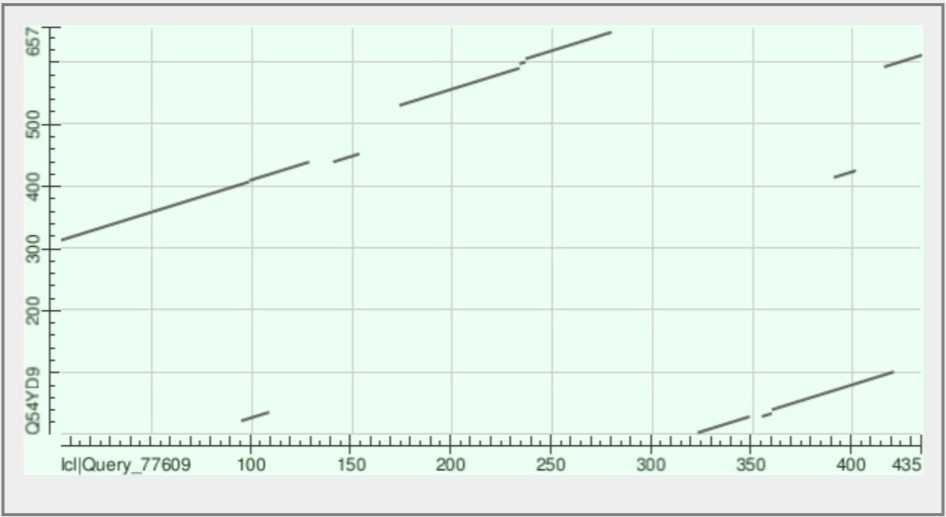
У последовательностей есть три крупных гомологичных участка, и они почти полностью покрывают альдолазу. Также можно заметить два коротких участка дупликации в белке синтеза фолиевой кислоты и небольшую делецию в середине альдолазы (там, где прерывается второй крупный участок сходства). Похожих мест у белков много, даже хочется предположить гомологию, но тут ее точно нет - а жаль.
Игры с Blast
Попытка проверить работу алгоритма на некодирующих последовательностях провалилась. Несмотря на то, что на фразу "QUOTHTHERAVENNEVERMORE" выдается 20000 результатов (рис. 4), ни один из них не является достаточно привлекательным для того, чтобы назвать последовательности гомологичными. Если же запустить blast на стихотворении Уолта Уитмена "To a Stranger", то он и вовсе не выдает результатов.

Для дальнейшего изучения параметров был выбран белок связывания иммуноглобулинов (P11021): поиск со стандартными параметрами (кроме количества результатов) выдает 1104 последовательности. При изменении wordsize количество результатов меняется незначительно (1135 для 2 и 1137 для 3). При увеличении штрафа за гэпы до максимально возможного тоже мало что изменяется. На самом деле были произведены попытки поменять все возможные параметры и они привели к... отсутствию какого-либо значительного отклика. Кажется, все дело в занимательной плашке, которой раньше не наблюдалось (рис. 5). Возможно ли, что этот белок настолько консервативен, что изменение параметров не сказывается на результатах работы алгоритма?
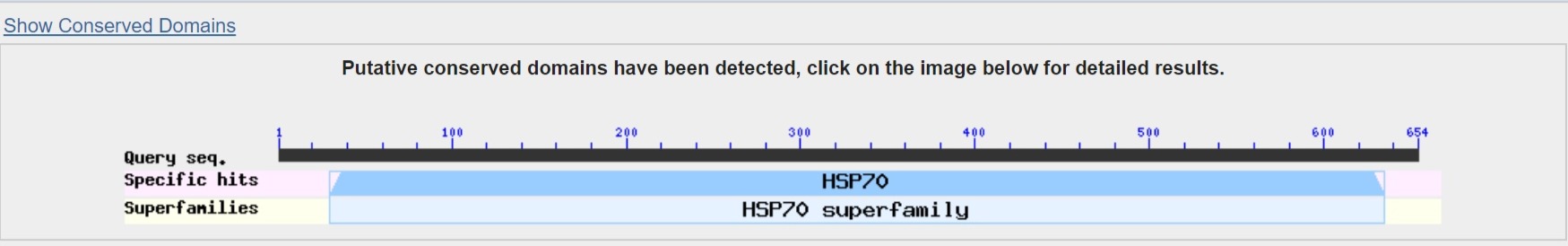
Вообще, поиски белка, над которым можно было бы ставить эксперименты, заняли некоторое время. Зато я увидела больше красивых плашек, сообщающих о консервативных участках (рис. 6).
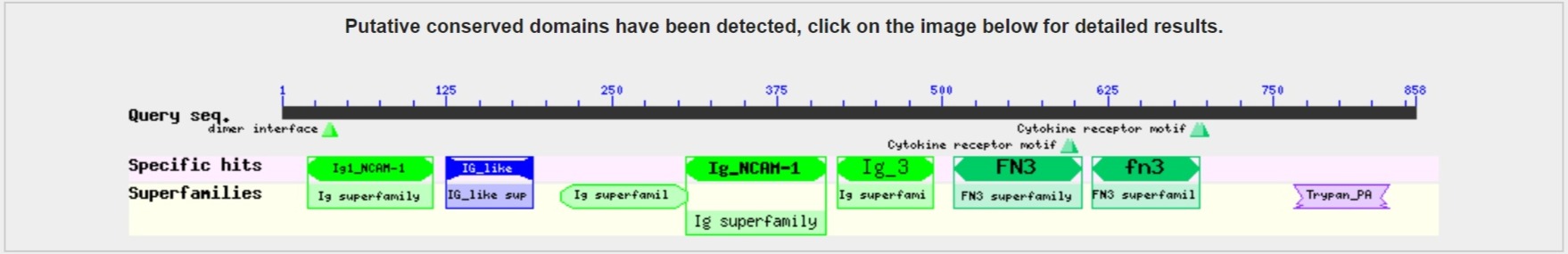
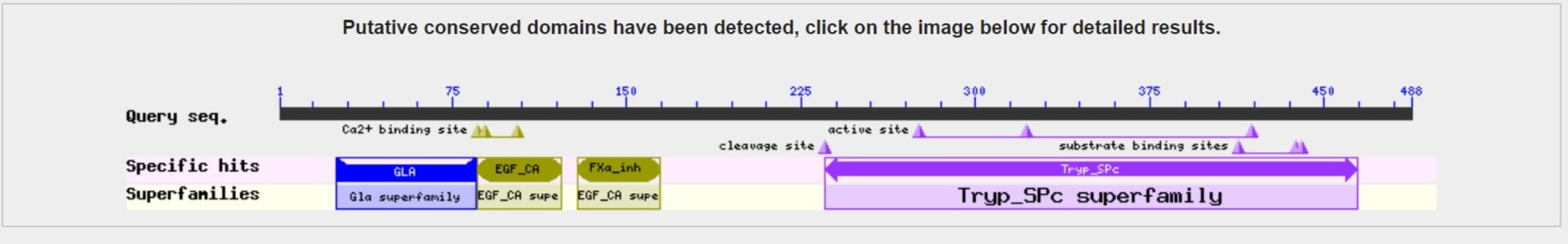
Все дальнейшие эксперименты были проведены с субъединицей ε АТФ-синтазы E. coli (P0A6E6). Изначальный поиск выдает 452 результата с наименьшим e-value 3e-98. При изменении параметра wordsize количество результатов увеличивается примерно на четверть (567 для 3 и 563 для 2). Если поменять матрицу на BLOSUM90, то минимальное e-value понизится на два порядка до 4e-100, а при использовании PAM30 увеличится до 1е-90. Уменьшение штрафов за гэпы до минимального не влияет на количество последовательностей, но закономерным образом понижает e-value аж до 3e-79, между тем максимальный штраф за гэпы дает 5e-107. Тем временем изменение алгоритма на delta дает, пожалуй, самые неожиданные результаты: 727 последовательностей с наименьшим e-value 6e-65.