Анализ транскриптомов
Практикум был выполнен для восьмой хромосомы.
| копирование файла | cp ../Human/rnaseq_reads/chr8.1.fastq . |
|---|---|
| анализ качества чтений | fastqc chr8.1.fastq |
| очистка чтений | java -jar /nfs/srv/databases/ngs/suvorova/trimmomatic/trimmomatic-0.30.jar SE -phred33 chr8.1.fastq chr8.1_trimmed.fastq TRAILING:20 MINLEN:50 |
| анализ качества чтений [2] | fastqc chr8.1_trimmed.fastq* |
| картирование чтений | hisat2 -x index -U chr8.1_trimmed.fastq -S chr8.1.sam --no-softclip |
| перевод в .bam | samtools view -b -o chr8.1.bam chr8.1.sam |
| сортировка чтений | samtools sort chr8.1.bam chr8.1_sorted |
| индексирование файла | samtools index chr8.1_sorted.bam |
| подсчет чтений | htseq-count -f bam chr8.1_sorted.bam -s no ../Human/rnaseq_reads/gencode.v19.chr_patch_hapl_scaff.annotation.gtf > results.txt |
| подсчет генов с ненулевым наложением | grep -vw 0 results.txt |
*--no-spliced-alignment используется только для днк-чтений, так как части мРНК в геноме могут быть разделены интронами
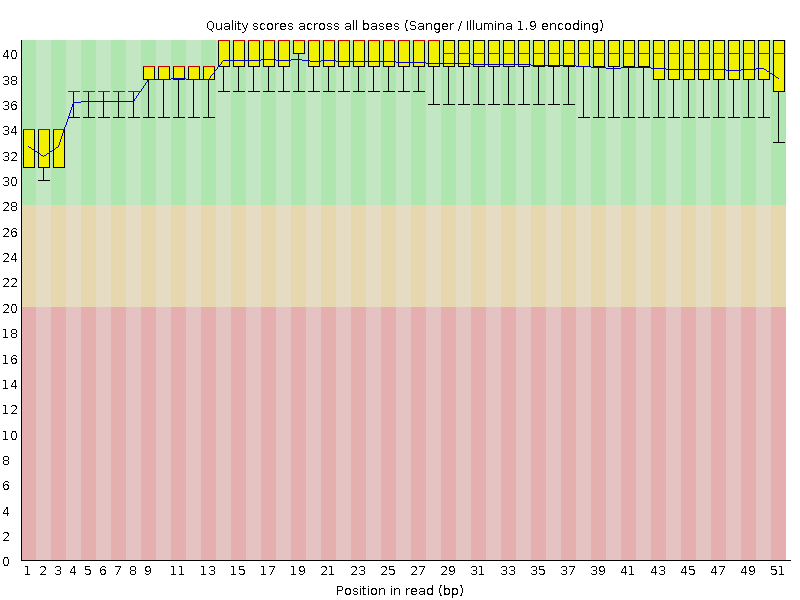
Результат работы команды Trimmomatic:
Input Reads: 17763 Surviving: 17612 (99,15%) Dropped: 151 (0,85%)
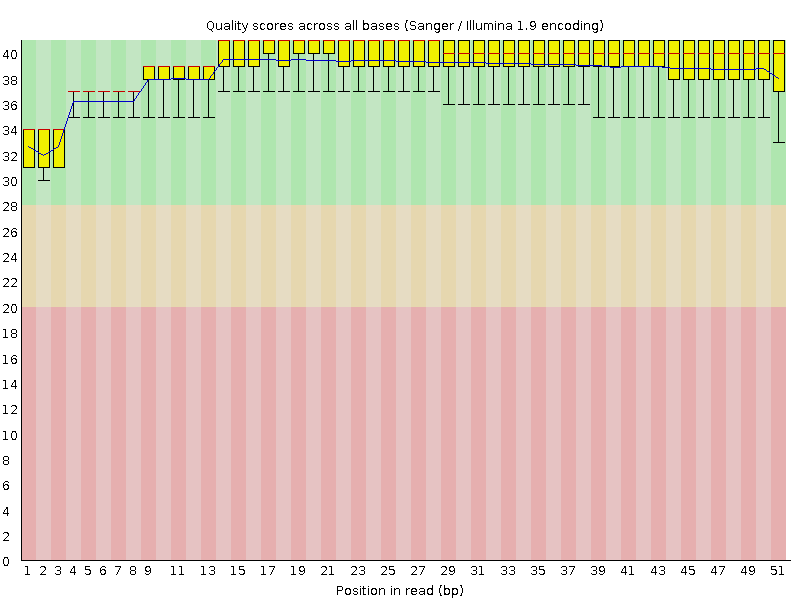
Скорее всего можно было обойтись и без триммирования. Убирается меньше процента чтений, и визуально разницы нет совершенно - изначально было довольно высокое качество.
Результат работы команды hisat2:
17612 reads; of these:
17612 (100.00%) were unpaired; of these:
301 (1.71%) aligned 0 times
17307 (98.27%) aligned exactly 1 time
4 (0.02%) aligned >1 times
98.29% overall alignment rate
На геном откартировалось 98,29% чтений. Качество хорошее.
Описание опций htseq-count:
- -f формат входных данных (sam по умолчанию)
- -s специфичность данных относительно какой-то цепи днк (yes - прямой - по умолчанию)
- -i атрибут GFF, используемый как feature ID (gene_id по умолчанию)
- -m правила, определяющие, когда именно чтение накладывается на ген (union по умолчанию)
Результаты наложения чтений:
ENSG00000104738.12 281
ENSG00000253729.3 15816
__no_feature 1209
__ambiguous 1
__not_aligned 301
__alignment_not_unique 8
Как видно из результатов выше, большая часть чтений накладывается на один из двух генов. Еще девять случаев неоднозначные или неуникальные, и чуть больше 1500 чтений не накладываются ни на что. Они могут быть остатками праймеров или поли-а-концами созревших мРНК.
Подробнее о двух белках. MCM4 - один из компонентов МСМ-комплекса, который является реплицирующей геликазой, необходимой для инициации и элонгации репликации ДНК в эукариотических клетках. PRKDC - каталитическая субъединица ДНК-зависимой протеинкиназы. Это серин/треонин-протеинкиназа, которая является сенсором повреждений ДНК. Она участвует в процессе негомологичного соединения концов, необходимого при двуцепочечных разрывах ДНК и рекомбинации.