Секвенирование
Консенсусная последовательность
Практикум выполнялся с помощью программы CodonCode Aligner. Консенсусная последовательность и выравнивание доступны для скачивания, так же как и исходные прочтения.
Немного информации про прочтения:
- явно нечитаемые участки в прямом прочтении составляют 1-21 и 711-717 нуклеотиды, но программа на всякий случай отрезает больше: 40 нуклеотидов в начале и 44 в конце
- в обратном это 1-34 и 708-718 соответственно, а программа обрезает 34 нуклеотида в начале и 42 в конце
- хроматограмма в целом довольно хорошего качества
Всего было четыре проблемных места, которые представлены на картинке ниже.
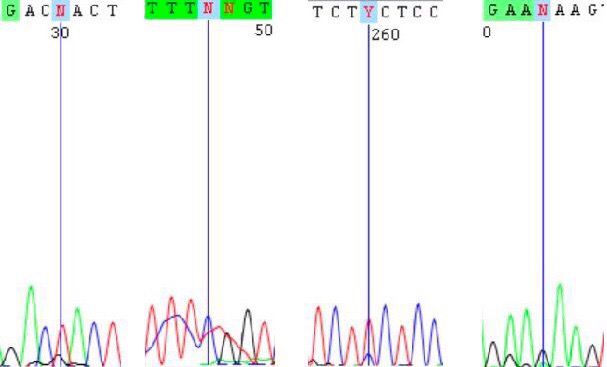
В первом и третьем случае очевидно, что изначально там должен был быть тимин. Во втором случае мог произойти небольшой затек, но предполагается, что это цитозин. В четвертом пик совсем маленький, но ровный и все равно значительно больше шума, так что можно поставить гуанин. Все исправления согласуются со вторым прочтением.
Также для оценки сигнала и потому что я люблю ggplot были построены графики, отражающие разницу в качестве сигнала у двух прочтений и консенсусной последовательности. В качестве данных были использованы файлы с расширением .qual, которые CodonCode автоматически создает вместе с экспортом последовательностей.
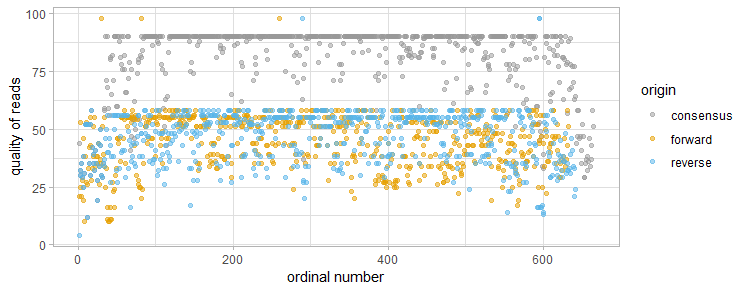
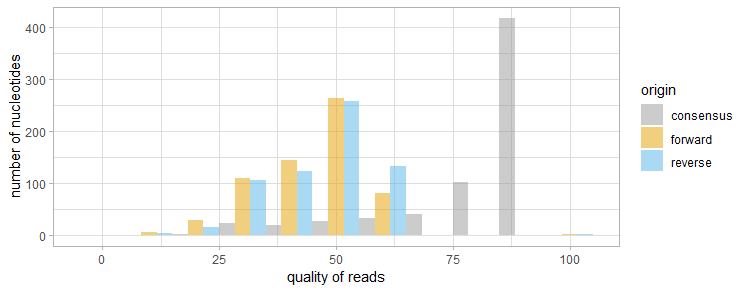
Плохая хроматограмма
В представленном ниже участке хроматограммы очень высок уровень шума, что затрудняет определение нуклеотидов. С 634 позиции, судя по всему, произошел затек краски. Около 620 и 610 позиций шум почти совпадает с сигналом, а на 615, 622 и 626 вообще сложно отличить, где шум, а где сигнал.