Визуализация
Выдача blast выглядит примерно так:
CLPX_CLOTE Q891J8 ATP-dependent Clp protease ATP-binding subuni... 538 0.0
CLPX_MOOTA Q2RL30 ATP-dependent Clp protease ATP-binding subuni... 533 0.0
CLPX_GEOKA Q5KWJ9 ATP-dependent Clp protease ATP-binding subuni... 520 0.0
CLPX_BACAN Q81LB9 ATP-dependent Clp protease ATP-binding subuni... 513 2e-180
CLPX_LISMO Q8Y7K9 ATP-dependent Clp protease ATP-binding subuni... 511 1e-179
CLPX_STAA8 Q2FXQ7 ATP-dependent Clp protease ATP-binding subuni... 498 1e-174
CLPX_ENTFA Q833M7 ATP-dependent Clp protease ATP-binding subuni... 498 2e-174
HSLU_GEOKA Q5L0N1 ATP-dependent protease ATPase subunit HslU OS... 105 6e-25
HSLU_ENTFA Q834K4 ATP-dependent protease ATPase subunit HslU OS... 103 4e-24
Q2RJP5_MOOTA Q2RJP5 ATP-dependent protease ATPase subunit HslU ... 100 4e-23
HSLU_LISMO Q8Y7J8 ATP-dependent protease ATPase subunit HslU OS... 99.8 6e-23
HSLU_BACAN Q81WK6 ATP-dependent protease ATPase subunit HslU OS... 98.2 2e-22
HSLU_STAA8 Q2FZ28 ATP-dependent protease ATPase subunit HslU OS... 98.2 2e-22
Q8Y8B1_LISMO Q8Y8B1 UVR domain-containing protein OS=Listeria m... 60.5 5e-10
CLPL_STAA8 Q2FV74 ATP-dependent Clp protease ATP-binding subuni... 57.8 4e-09
Q2RLR4_MOOTA Q2RLR4 AAA domain-containing protein OS=Moorella t... 50.1 6e-07
Дерево было построено методом UPGMA с бутстрепом.
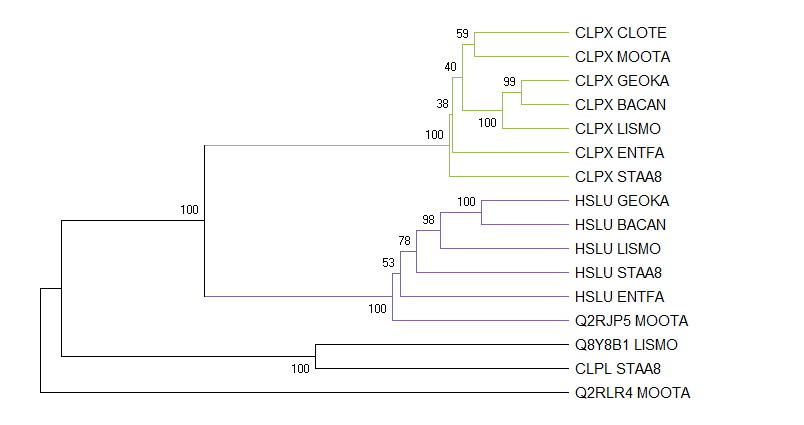
Ортологи:
- CLPX CLOTE & CLPX MOOTA
- HSLU BACAN & HSLU GEOKA
- Q8Y8B1 LISMO & CLPL STAA8
Паралоги:
- CLPX ENTFA & HSLU ENTFA
- HSLU STAA8 & CLPL STAA8
- Q8Y8B1 LISMO & CLPX LISMO
В группе CLPX по белку из каждой бактерии, у HSLU - все, кроме CLOTE. При этом филогения второй группы полностью совпадает с исходной, тогда как первая содержит только две нужные ветви (CLOTE & MOOTA, BACAN & GEOTA).
P.S. Доподлинно неизвестно, что происходит с надписями на картинке выше, но привести в порядок их не удалось.