Пуриновые и примидиновые азотистые основания.
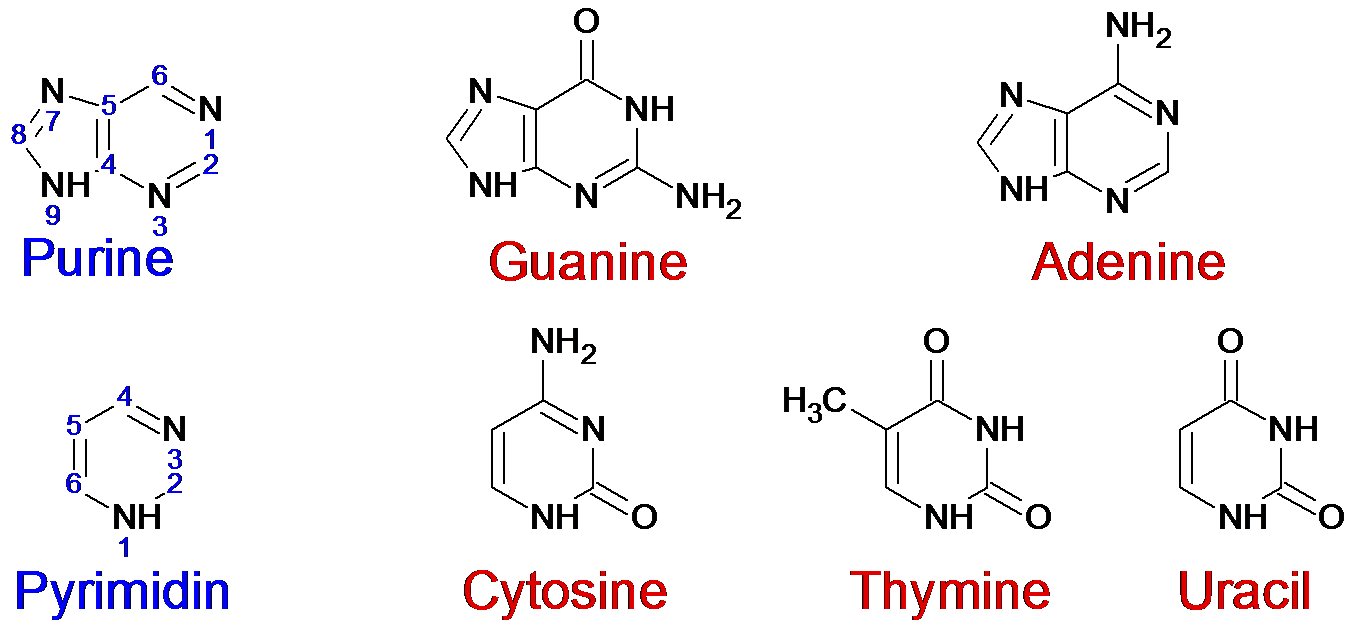
Канонические пары.
Изображения нуклеотидов и фрагмента ДНК,полученные с помощью программы ChemSketch.
A-форма ДНК.
В-форма ДНК.
Z-форма ДНК.
Упр.2.
Исходные файлы PDB 1mhd.pdb и 1h4s.pdb
Упр.3.
Заданные структуры ДНК и РНК не содержат разрывов.
Соответствующие координаты атомов ДНК и РНК в отдельных файлах.
Упр.1. Нахождение малых и больших бороздок.
Результаты упражнения с пояснениями в файле forms.sk2.
Упр.2. Сравнить основные спиральные параметры разных форм ДНК.
Откройте в RasMol файлы, полученные при выполнении задания 4.Скопируйте в отчет следующую таблицу.
Изучите структуры, полученные данные внесите в таблицу.
| A-форма | B-форма | *Z-форма | |
| Тип спирали (правая или левая) | левая | левая | правая |
| Шаг спирали (Å) | 28,03 | 33,75 | 43.50 |
| Число оснований на виток | 11 | 10 | 13 |
| Ширина большой бороздки (Å) | 18,49 (T19A-A26В) | 20,58 (G25В-T11А) | 23,51(C12A-C24B) |
| Ширина малой бороздки (Å) | 9,63(A26В-С8А) | 13,20 (Т11А-Т35В) | 20,07(C36B-C12A) |
См. подсказки.
Упр.3. Сравнить торсионные углы в структурах А- и В-форм.
С помощью команды Settings->Torsion RasMol измерьте торсионные углы выбранного в упр. 1 нуклеотида. Сравните значения углов в А- и В-форме,
сравните со значениями, приведенными в презентации.
Форма контроля — итоговая контрольная и отчет.
Задание 7.Определение параметров структур нуклеиновых кислот с помощью программ пакета 3DNA.
Упражнение 1.Торсионные углы нуклеотидов.
| Торсионные углы | A-форма | B-форма | Z-форма(для C) | Z-форма(для G) | RNA | DNA |
| alpha(˚) | -51.7 | -29.9 | -139.5 | 52.0 | -56.1 | -52.7 |
| beta(˚) | 174.8 | 136.4 | -136.7 | 179.0 | 166.1 | 147.3-158.4 |
| gamma | 41.7 | 31.1-31.2 | 50.9 | -173.8 | 59.5 | 41.5-47.9 |
| delta(˚) | 79.0-79.1 | 143.3-143.4 | 137.6 | 94.9 | 83.9 | 141.6 |
| epsilon(˚) | -147.8 | -140.8 | -96.5 | -103.6 | -154.9 | -154.3 или 157.9 |
| zeta(˚) | -75.0- -75.1 | -160.5 | 81.9 | -64.8 | -73.2 | -118.4 | �
| chi(˚) | -157.2 | -98.0 | -154.3 | 58.7 | -163.5 | -91.6 или -106.0 |
Вывод:
- в виду того,что Z-ДНК-полиформа,сравним торсионные углы А и В-структур:практически в 2 раза отличаются углы delta, zeta, чуть меньше углы alpha и chi.
-торсионные углы в структуре тРНК больше всего схожи с торсионными углами А-ДНК ;
-в структуре ДНК найболее "деформированным" нуклеотидом является T9 второй цепи(3 отклониния от среднего значения), а также Т12 цепи1 и A12 цепи2( в таблице nuclacid.xls отличия показаны красным цветом);
-средние значения торсионных углов для данной тРНК и ДНК проведены в файле nuclacids.xls.;
Упражнение 2.Структура водородных связей.
1.Номера нуклеотидов, образующих стебли(stems) во вторичной структуре заданной тРНК:
G4 -C69, G5 -C68, A6 -U67, G7 -C66, G49-U65, C50-G64, U51-A63, G52-C62, G53-C61, u54-G58, P55-G18, A38-U32, C39-G31, G40-C30, A41-U29, G42-C28, G43-C27, G44-A26, G10-C25, C11-G24, G12-C23, C13-G22, A14-U8 , G15-C48, G19-C56.
Примечание:U[5MU]- 5-METHYLURIDINE 5'-MONOPHOSPHATE и P[PSU]- PSEUDOURIDINE-5'-MONOPHOSPHATE.
2.В данной структуре есть 4(6) не Уотсон-Криковские пары нуклеотидов:
| Пара нуклеотидов | Число водородных связей | Атомы,длина связи (Å) | Атомы,длина связи (Å) | Примечание |
| G49-U65 | 2 | O6 - N3 ( 2.92) | N1 - O2 (2.94) | |
| u54-G58 | 2 | N3 - N7 (2.51) | O2 * O6 (2.55) | U[5MU]- 5-METHYLURIDINE 5'-MONOPHOSPHATE |
| P55-G18 | 2 | O4 * N1 (2.69) | O2'* O6 (3.43) | P[PSU]- PSEUDOURIDINE-5'-MONOPHOSPHATE |
| G44-A26 | 2 | O6 - N6 (3.05) | N1 - N1 (2.98) | |
| A14-U8 | 2 | N7 - N3 (2.65) | N6 - О2 (2.49) | В классической паре,например в A6 -U67, водородные связи возникают между N6 - O4 и N1 - N3. |
| G15-C48 | 2 | N1 - O2 (2.73) | N2 - N3 (2.77) | В классической паре,например в G5 -C68,3 водородные связи возникают между O6 - N4, N1 - N3 и N2 - O2. |
3.Дополнительные водородные связи в тРНК, стабилизирующие ее третичную структуру :
Упражнение 3.Возможные стекинг-взаимодействия.
В файле rna_old.out указаны возможные стекинг-взаимодействия между динуклеотидными парами данной тРНК.При этом в 5 колонках указываются площади перекрывания оснований относительно друг друга ( колонки i1-i2 и j1-j2) , площади перекрывания нуклеотида одной цепи относительно нуклеотида другой цепи (соответственно колонки i1-j2 и j1-i2), а также сумированная площадь перекрывания пар( в скобках дана площадь перекрывания только основных атомов пиримидинового или пуринового колец, без скобок-площадь,учитывающая нециклические атомы).
За пары с наибольшей площадью перекрывания приняты соответственно пары с сумированной площадью перекрывания,учитывающей нециклические атомы.
Это пары 9 Gu/GC (13.24), 10 uP/GG (13.24) и 5 GC/GU (13.11).
Также для сравнения взяты пары с минимальной площадью перекрывания:11 PA/UG (0.0), 18 GG/CA (0.80).
Упражнение 1.
Исходные скрипты:
-Множество атомов кислорода рибозы,
-Множество атомов кислорода в остатке фосфорной кислоты ,
-Множество атомов азота в азотистых основаниях ,
-Общий скрипт.
Упражнение 2.ДНК-белковые контакты в заданной структуре.
Таблица. Контакты разного типа в комплексе 1MHD.pdb
| Контакты атомов белка с | Полярные | Неполярные | Всего |
| остатками 2'-дезоксирибозы | 0 | 9 | 9 |
| остатками фосфорной кислоты | 9 | 2 | 11 |
| остатками азотистых оснований со стороны большой бороздки | 4 | 5 | 10 |
| остатками азотистых оснований со стороны малой бороздки | 4 | 3 | 7 |
Больше всего контактов возникает между атомами белка и остатками фосфорной кислоты,а также остатками азотистых оснований со стороны большой бороздки.Это объясняется тем,что данные атомы ДНК находятся "ближе" всего к белку,то есть на их взаимодействие с атомами белка не влияют другие связи в молекуле ДНК.
Упражнение 3. Популярная схему ДНК-белковых контактов,полученная с помощью программы nucplot
Упражнение 4.
а) аминокислотный остаток с наибольшим числом указанных на схеме контактов с ДНК: LEU71 (Цепи А или В,так как они идентичны) образует две связи с атомом фосфора длиной меньше 3.35 ангстрем.
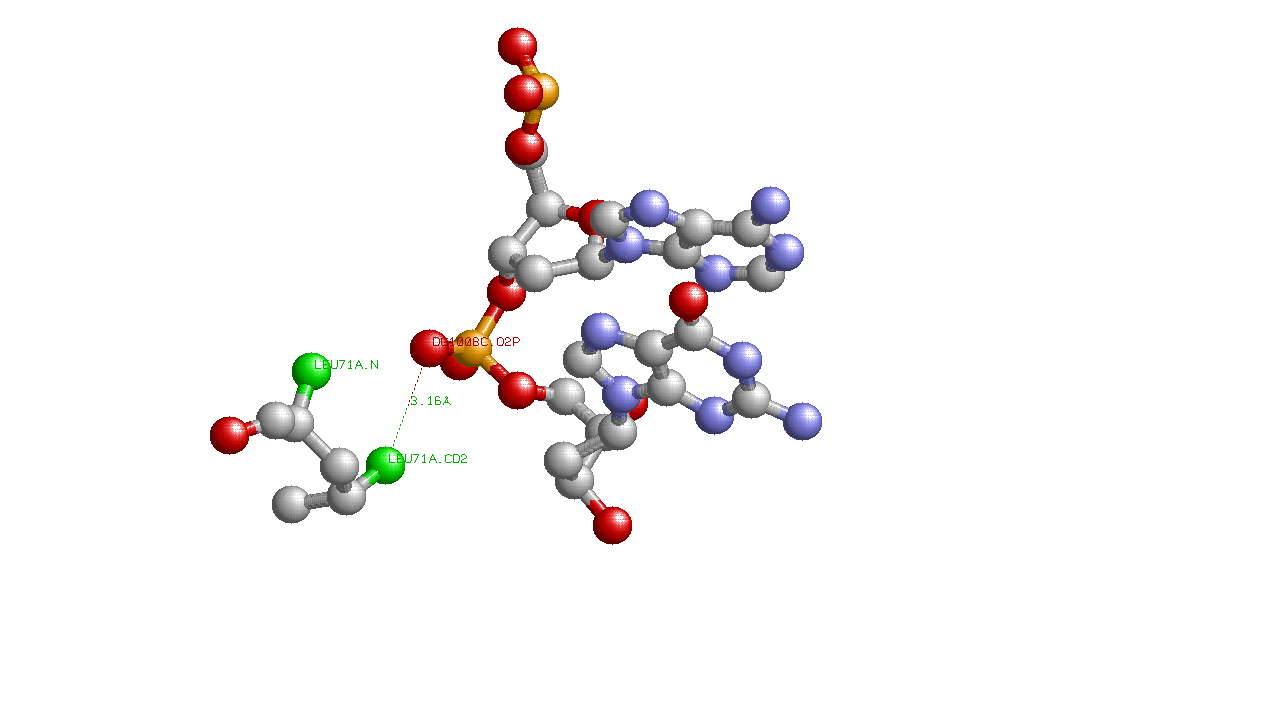
Примечание: на картинке зеленым показаны атомы Leu71:A,образующие связи с атомом кислорода DG1008C.O2P, длины этих связей 3.16 и 3.17 ангстрем(на картинке показана одна связь). б)по-моему мнению, наиболее важный аминокислотный остаток для распознавания последовательности ДНК -Arg74(B), так как он образует водородную связь непосредственно с G1003, или GLN76(B),образующий водородную связь с А2008.
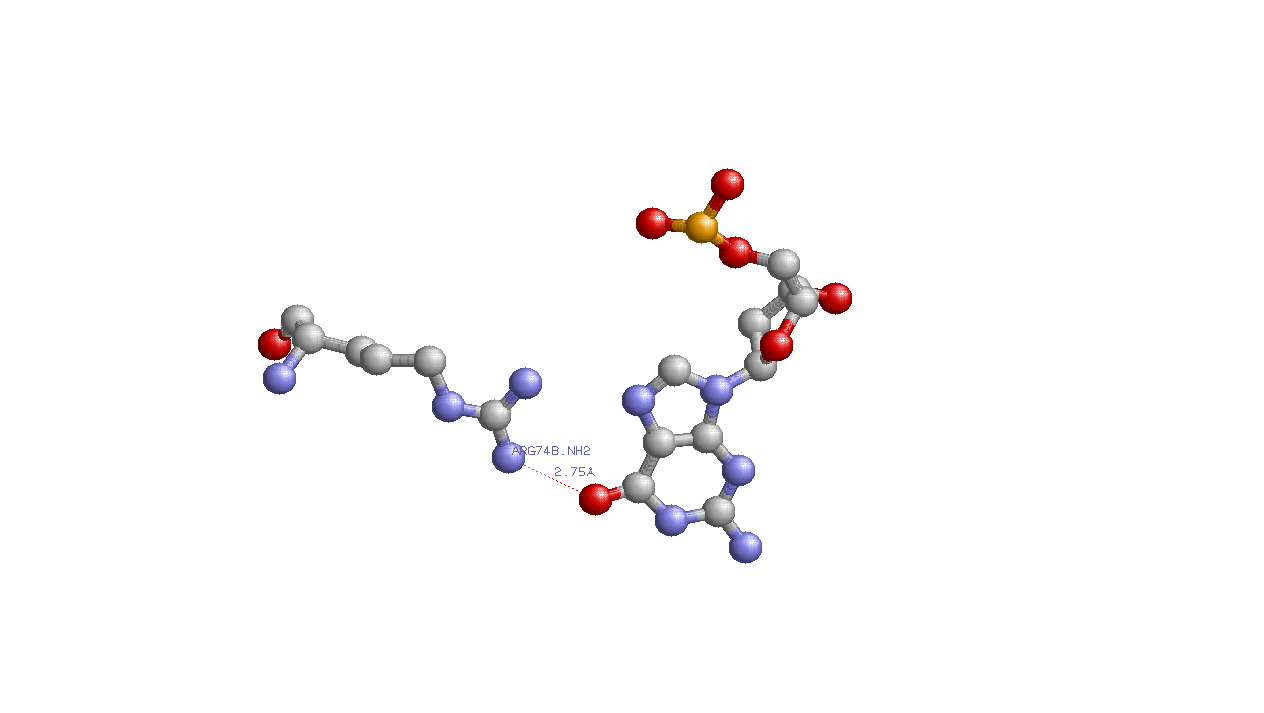
Контакт Arg74(B) и G1003.Водородная связь предположительно образована атомами NH2 аминокислоты и О6 гуанина.
Контакт Gln76(B) и A2008.Водородная связь предположительно образована атомами OE1 аминокислоты и N6 аденина.
Задание 9 (обязательное, результаты выполнения нужно привести в отчете)
Предсказание вторичной структуры заданной тРНКУпр.1. *Предсказание вторичной структуры тРНК путем поиска инвертированных повторов
Постарайтесь подобрать параметры для получения предсказания, наиболее близкого к реальной структуре.
Упр.2. *Предсказание вторичной структуры тРНК по алгоритму Зукера.
Программа mfold из пакета EMBOSS реализует алгоритм Зукера.
Постарайтесь подобрать параметры для получения предсказания, наиболее близкого к реальной структуре.
Результаты внесите в таблицу, приведенную ниже.
Сохраните и внесите в отчет картинку с лучшим предсказанием, а также укажите, каким по счету
оно было.
Реальная и предсказанная вторичная структура тРНК из файла XXXX.pdb
| Участок структуры (расшифровку названий см. на рис. 2 в статье О.О.Фаворовой) |
Позиции в структуре (по результатам find_pair) |
Результаты предсказания с помощью einverted |
Результаты предсказания по алгоритму Зукера |
| Акцепторный стебель | 5' 901-907 3' 5' 966-972 3' Всего 7 пар |
предсказано 2 пары из 7 реальных | |
| D-стебель | |||
| T-стебель | |||
| Антикодоновый стебель | |||
| Общее число канонических пар нуклеотидов |