| Команда | Функция |
|---|---|
| java -jar /nfs/srv/databases/ngs/suvorova/trimmomatic/trimmomatic-0.30.jar SE -phred33 SRR4240356.fastq SRR4240356noadapters.fastq ILLUMINACLIP:adapters.fasta:2:7:7 | Удаляет возможные адаптеры с ридов из файла SRR4240356.fastq, выход с ридами без адаптеров направляется в SRR4240356noadapters.fastq; последовательности адаптеров лежат в файле adapters.fasta. |
| java -jar /nfs/srv/databases/ngs/suvorova/trimmomatic/trimmomatic-0.30.jar SE -phred33 SRR4240356noadapters.fastq SRR4240356edited.fastq TRAILING:20 MINLEN:30 | Отрезает с конца каждого рида в файле SRR4240356noadapters.fastq нуклеотиды с качеством ниже 20 и оставляет риды длиной не менее 30 нуклеотидов; выходные данные записывает в SRR4240356edited.fastq. |
В итоге из 7358438 входных ридов было оставлено 7075381 ридов (96,15%) и выкинуто 283057 (3,85%); размер файла SRR4240356noadapters.fastq (до очистки) - 741 М, SRR4240356edited.fastq (после очистки) - 712 М.
| Команда | Функция |
|---|---|
| velveth ./ 29 -fastq -short SRR4240356edited.fastq | Создает в текущей директории k-меры длины 29 из коротких непарных ридов из файла SRR4240356edited.fastq. |
| velvetg ./ | На вход принимает файлы, которые создала velveth; в текущей директории собирает контиги из k-меров. |
N50 для получившихся контигов - 46003.
| ID контига | Длина контига | Покрытие |
|---|---|---|
| 7 | 115468 | 52.223586 |
| 20 | 106076 | 45.974914 |
| 8 | 75082 | 54.512946 |
| ID контига | Длина контига | Покрытие |
|---|---|---|
| 2 | 1 | 621.000000 |
| 26 | 173 | 569.387283 |
| 109 | 3 | 1.666667 |
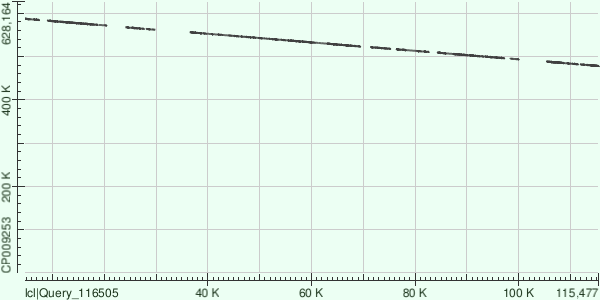
Координаты хромосомы, соответствующие контигу: 478095-587055. Число выравниваний: 17. По карте видно, что выравнивания легли на обратную цепь. Характеристика всех выравниваний, полученная с помощью команды infoalign: info7.txt.
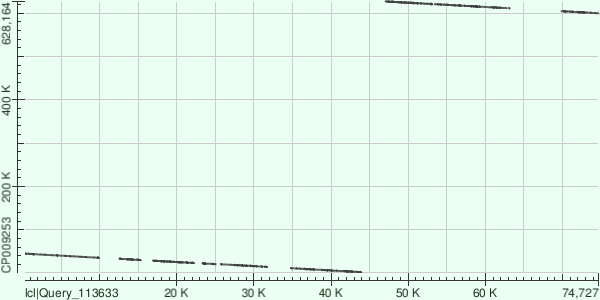
Buchnera aphidicola - бактерия, с кольцевой ДНК, поэтому координаты хромосомы, соответствующие контигу выглядят так: 2004-44693 и 599832-627104. Число выравниваний: 13. Как и в предыдущем контиге, выравнивания легли на обратную цепь. Файл с характеристикой всех выравниваний: info8.txt.
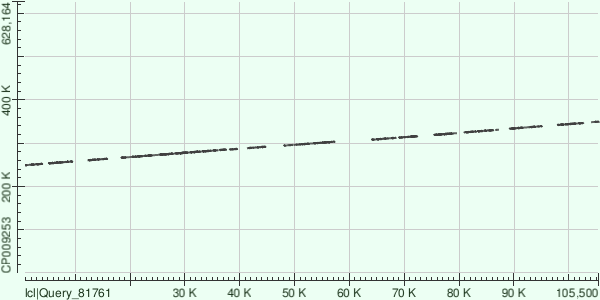
Координаты хромосомы, соответствующие контигу: 248967-349674. Число выравниваний: 20. Аналогично, выравнивания легли на обратную цепь. Файл с характеристикой всех выравниваний: info20.txt.
Резюмируя, можно сказать, что три самых крупных контига покрывают достаточно большой процент хромосомы, но до половины генома не дотягивают (оно и понятно, так как L50=4, четвертый контиг, с которым было бы покрыто больше половины генома - N50). Если говорить о расположении друг относительно друга, то стоит сказать, что контиг 7 достаточно близко прилежит к контигу 8: концевая координата крайнего выравнивания контига 7 - 587055, начальная координата крайнего выравнивания контига 8 - 599832; в то же время даже по картам видно, что контиг 20 расположен далеко от контигов 7 и 8.
| длина k-мера = 29 | длина k-мера = 25 | ||||
|---|---|---|---|---|---|
| ID контига | Длина контига | Покрытие | ID контига | Длина контига | Покрытие |
| 7 | 115468 | 52.223586 | 49 | 14084 | 80.661247 |
| 20 | 106076 | 45.974914 | 13 | 13372 | 81.418636 |
| 8 | 75082 | 54.512946 | 57 | 13359 | 77.077176 |
При уменьшении длины k-меров увеличивается вероятность того, что како-нибудь k-мер попадет в повторяющийся участок. При такой ситуации программе сложно соотнести, какое продолжение будет у контига после k-мера, который попал на повтор, тогда она просто останаваливает сборку контига и "режет" его на этом месте. То есть при уменьшении длины k-мера ожидается увеличение числа контигов и уменьшение их длин. Действительно, при уменьшении длины k-мера с 29 до 25 количество выходных последвательностей из файла stats.txt (выдача velvetg) увеличилось с 720 до 3572, а длина контигов, как видно из таблицы, значительно уменьшилась; при этом среднее покрытие увеличилось. Соответственно, значение N50 значительно уменьшилось: было 46003, стало 5332.
© Агаева Зара, 2018