
В рассматриваемой мною в шестом практикуме последовательности оказался фрагмент митохондриального гена I субъединицы цитохром с-оксидазы (COI):
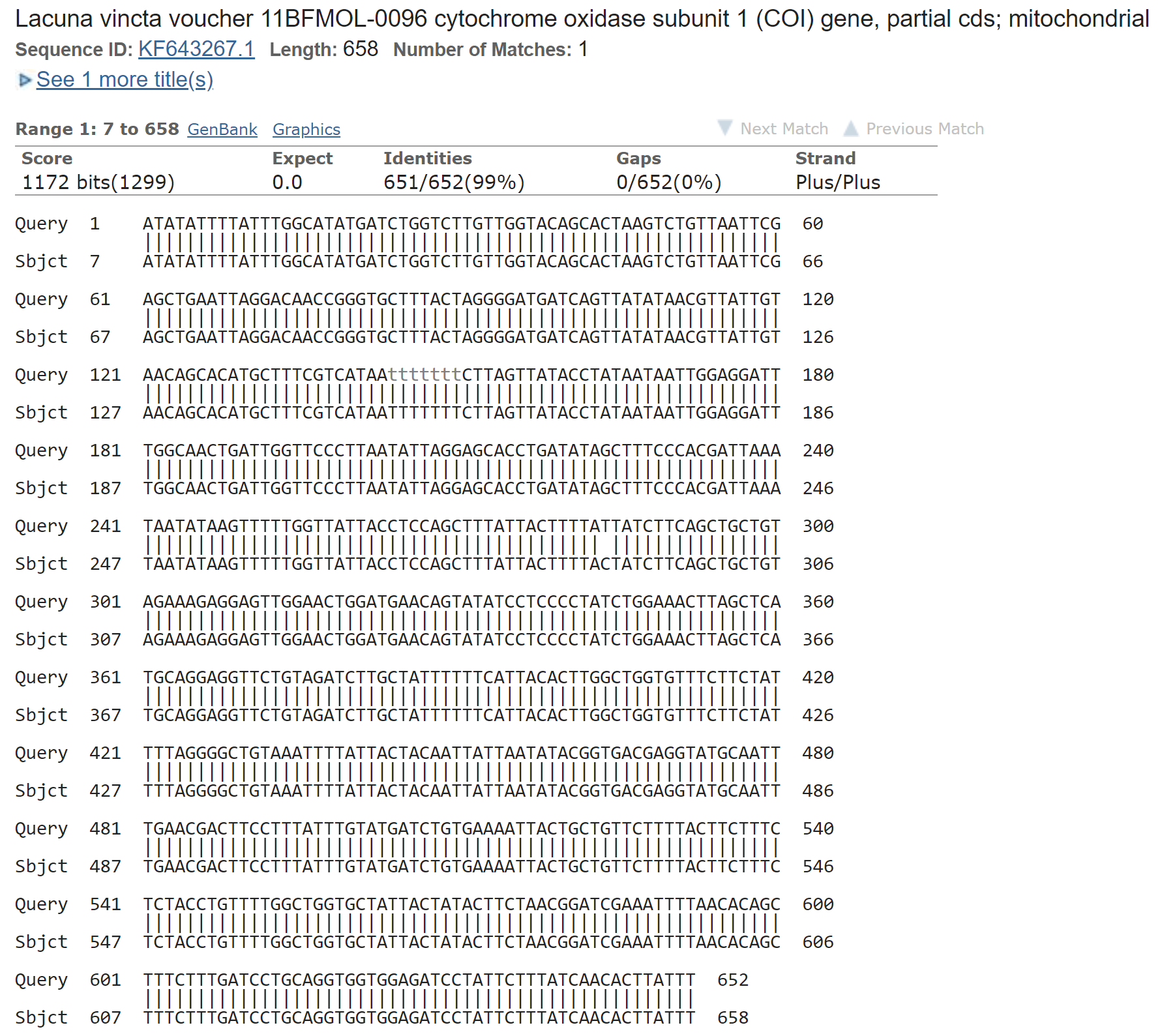
Этот ген встречается среди представителей одного класса, Гастроподов. Ниже приведено фрагмент множественного выравнивания находок, относящихся к разным видам. Видно, что некоторые последовательности выравнены друг относительно друга очень хорошо (напрмер, Echinolittorina trochoides и Lacuna vincta) и можно предположить что они относятся к одному порядку - действительно, эти 2 вида относятся к порядку Littorinimorpha (класс Gastropoda). Обратим внимание на последовательности, которые выравнялись плохо - Peasiella patula и Belomitra gymnobela. Первый организм - представитель порядка Littorinimorphа, второй - Neogastropoda; класс общий, Gastropoda. Таким образом, данный ген общий для представителей одного класса, Гастроподов.

Поиск гомологов митохондриального гена I субъединицы цитохром с-оксидазы. Я ограничила поиск родом Hastula (taxid:57628).
| BLAST | Измененные параметры (не по умолчанию) | Кол-во находок |
|---|---|---|
| megablast | 35 | все по умолчанию |
| blastn с параметрами по умолчанию | 48 | все по умолчанию |
| чувствительный blastn | 51 | Word size - 7; Match/Mismatch Scores - 1;-1 |
Видно, что blastn выдает больше находок, чем megablast. Выравнивание с одним из найденнных blastn генов, которых не оказалось в списке выдачи megablast, приведено ниже:
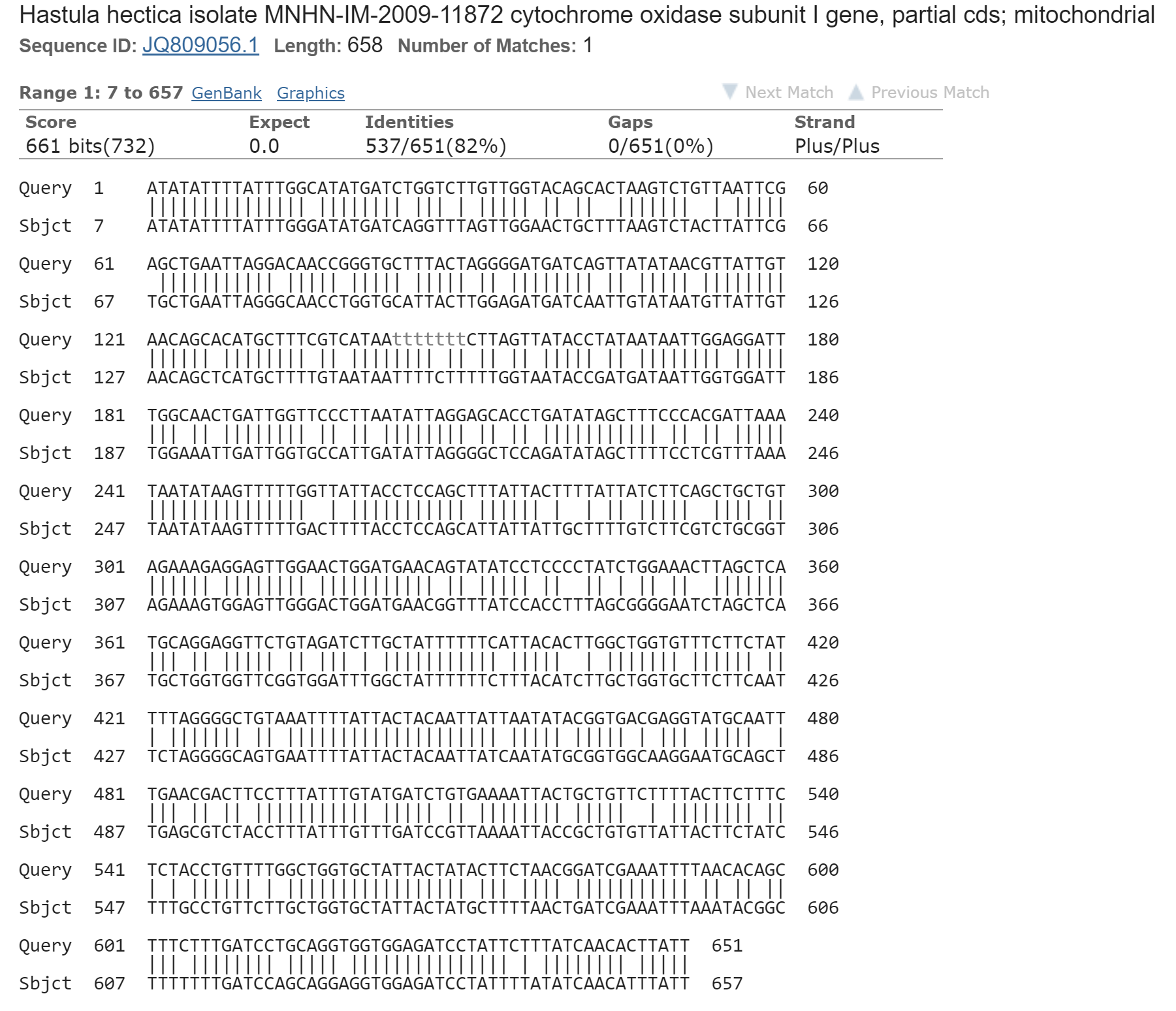
Длина слова для megablast равна по умолчанию 28. B данном выравнивании нет последовательности из, как минимум, 28 совпадающих нуклеотидов (подряд идущих), зато из 11 есть (длина слова для blastn с параметрами по умолчанию), поэтому этот ген в списке находок blastn есть, а megablasat - нет. Таким образом, megablast из-за высокого порога длины слова может пропустить достаточно много гомологичных последовательностей.
Кроме того, megablast не использует аффинные штрафы за гэпы (т е и за открытие гэпа, и за его продолжение вычитается штраф один и тот же), а в blastn по умолчанию открытие гэпа - минус 5, а за его продолжение - минус 2. Это так же подтверждает то, что megablast менее избирательный. В итоге сравнение megablast и blastn говорит о том, что megablast позволяет быстро найти в банке последовательности, максимально похожие на исходный ген, а blastn чувствителенее и нацелен на поиск гомологичных последлвательностей (это касается как blastn с параметрами по умолчанию так и без). Стоит еще упомянуть, что в список находок blastn, в отличие от megablast, вошли несколько коротких фрагментов целых генов (пример на скриншоте ниже), то есть megablast не учитывает небольшие фрагменты последовательностей, которые могут быть гомологичными участку исходного гена.
Сравнение blastn с разными параметрами показало,что выдача blastn с чувствительными парметрами включает в себя выдачу blastn с параметрами по умолчанию и помимо этого включает в себя больше находок с короткими выравниваниями.
В этом задании требуется найти гомологи белков по нуклеотидной базе данных, поэтому используется TBLASTN.
В геноме Amoeboaphelidium protococcarum есть 2 гена, кодирующих гомологичный искомому белок; параметры выравнивания для одного из них приведены ниже.
| Scaffold | Score (bits) | E-value | Identities | Positives | Gaps |
|---|---|---|---|---|---|
| 26 | 693 | 0.0 | 87% | 94% | 0 |
Query 43 QLERMNVYFNEASGNKYVPRAVLVDLEPGTMDAVRAGPFGQLFRPDNFVFGQSGAGNNWA 102
QL + VY+NEASG KYVPRAVLVDLEPGTMD+VRAGP+G LFRPDNF+FGQSGAGNNWA
Sbjct 109811 QLYLIVVYYNEASGGKYVPRAVLVDLEPGTMDSVRAGPYGNLFRPDNFIFGQSGAGNNWA 109632
Query 103 KGHYTEGAELVDQVLDVVRREAEGCDCLQGFQITHSlgggtgagmgtllISKIREEFPDR 162
KGHYTEGAELVD VLDVVR+EAEGCDCLQGFQITHSLGGGTGAGMGTLLISKIREE+PDR
Sbjct 109631 KGHYTEGAELVDSVLDVVRKEAEGCDCLQGFQITHSLGGGTGAGMGTLLISKIREEYPDR 109452
Query 163 MMATFSVVPSPKVSDTVVEPYNATLSVHQLVENSDETFCIDNEALYDICMRTLKLSNPSY 222
MM TFSVVPSPKVSDTVVEPYNATLSVHQLVENSDETFCIDNEALYDIC RTLKL+ P+Y
Sbjct 109451 MMCTFSVVPSPKVSDTVVEPYNATLSVHQLVENSDETFCIDNEALYDICFRTLKLTTPTY 109272
Query 223 GDLNHLVSAVMSGVTVSLRFPGQLNSDLRKLAVNMVPFPRLHFFMVGFAPLTSRGAHHFR 282
GDLNHLVSAVMSGVT S+RFPGQLN+DLRKLAVNMVPFPRLHFFMVGFAPLTSRG+ +R
Sbjct 109271 GDLNHLVSAVMSGVTTSIRFPGQLNADLRKLAVNMVPFPRLHFFMVGFAPLTSRGSQQYR 109092
Query 283 AVSVPELTQQMFDPKNMMAASDFRNGRYLTCSAIFRGKVSMKEVEDQMRNVQNKNSSYFV 342
A+SV ELT QMFD KNMMAASD R+GRYL +AIFRGK+SMKEV++QM +VQ KNSSYFV
Sbjct 109091 ALSVAELTTQMFDAKNMMAASDPRHGRYLAVAAIFRGKMSMKEVDEQMLSVQTKNSSYFV 108912
Query 343 EWIPNNVQTALCSIPPRGLKMSSTFVGNSTAIQELFKRIGEQFTAMFRRKAFLHWYTGEG 402
EWIPNNV+TA+C IPP+GLKMS+TF+GNSTAIQELFKRI +QF+ MF+RKAFLHWYTGEG
Sbjct 108911 EWIPNNVKTAVCDIPPKGLKMSATFIGNSTAIQELFKRISDQFSVMFKRKAFLHWYTGEG 108732
Query 403 MDEMEFTEAESNMNDLVSEYQQYQDA 428
MDEMEFTEAESNMNDLVSEYQQYQDA
Sbjct 108731 MDEMEFTEAESNMNDLVSEYQQYQDA 108654
Из TBB_NEUCR, он же тубулин, формируются микротрубочки - компоненты цитоскелета, характерного для эукариотов; последовательность тубулина достаточно консервативна. Тот факт, что в в геноме A. protococcarum нашелся ген, кодирующий гомологичный белок, вполне понятен, так как A. protococcarum - эукариот.
Поиск по геному A. protococcarum выдал 1 ген, гомологичный гену HSP71_YEAST, а также 6 генов (характеристики выравнивания одного из них приведены ниже), кодирующих некоторые, видимо, консервативные участки данного белка (белок однодоменный).
| Scaffold | Score (bits) | E-value | Identities | Positives | Gaps |
|---|---|---|---|---|---|
| 199 | 920 | 0.0 | 79% | 90% | 3 |
Query 2 SKAVGIDLGTTYSCVAHFANDRVDIIANDQGNRTTPSFVAFTDTERLIGDAAKNQAAMNP 61
SKA+GIDLGTTYSCV + N++V+IIAND GNRTTPS+VAFTD+ERL+GDAAKNQ +NP
Sbjct 1109256 SKAIGIDLGTTYSCVGVW*NEKVEIIAND*GNRTTPSYVAFTDSERLLGDAAKNQVGLNP 1109077
Query 62 SNTVFDAKRLIGRNFNDPEVQADMKHFPFKLIDVDGKPQIQVEFKGETKNFTPEQISSMV 121
NTVFDAKRLIGR F D EVQ+DMKH+PFK+ID GKP I VE+ GETK FTPE++S+MV
Sbjct 1109076 YNTVFDAKRLIGRKFADAEVQSDMKHWPFKVIDKAGKPFI*VEYLGETKTFTPEEVSAMV 1108897
Query 122 LGKMKETAESYLGAKVNDAVVTVPAYFNDSQRQATKDAGTIAGLNVLRIINEPTAAAIAY 181
L KMKETAE++LGAKV +AVVTVPAYFNDSQRQATKDAG+IAGLNV+RIINEPTAAAIAY
Sbjct 1108896 LTKMKETAEAFLGAKVTNAVVTVPAYFNDSQRQATKDAGSIAGLNVMRIINEPTAAAIAY 1108717
Query 182 GLDKKGK-EEHVLIFDLGGGTFDVSLLSIEDGIFEVKATAGDTHLGGEDFDNRLVNHFIQ 240
GLDKK K E++VLIFDLGGGTFDVSLL+IE+GIFEVKATAGDTHLGGEDFDNRLV HF Q
Sbjct 1108716 GLDKKTKGEKNVLIFDLGGGTFDVSLLTIEEGIFEVKATAGDTHLGGEDFDNRLVTHFAQ 1108537
Query 241 EFKRKNKKDLSTNQRALRRLRTACERAKRTLSSSAQTSVEIDSLFEGIDFYTSITRARFE 300
EFKRK+KKDLS N R+LRRLRTACERAKRTLSS+ Q S+EIDSLFEG+DFYTSITRARFE
Sbjct 1108536 EFKRKHKKDLSGNARSLRRLRTACERAKRTLSSATQASIEIDSLFEGVDFYTSITRARFE 1108357
Query 301 ELCADLFRSTLDPVEKVLRDAKLDKSQVDEIVLVGGSTRIPKVQKLVTDYFNGKEPNRSI 360
ELC DLFR TLDPVEKVLRD+K+DKSQVDEIVLVGGSTRIPKVQKLV+D+FNGKEPN++I
Sbjct 1108356 ELCGDLFRGTLDPVEKVLRDSKIDKSQVDEIVLVGGSTRIPKVQKLVSDFFNGKEPNKTI 1108177
Query 361 NPDEavaygaavqaaILTGDESSKTQDlllldvaplslGIETAGGVMTKLIPRNSTIPTK 420
NPDEAVAYGAAVQA+IL+G+ S KT DLLLLDVAPLSLGIETAGGV T LI RN+TIPTK
Sbjct 1108176 NPDEAVAYGAAVQASILSGETSEKT*DLLLLDVAPLSLGIETAGGVFTALIKRNTTIPTK 1107997
Query 421 KSEIFSTYADNQPGVLIQVFEGERAKTKDNNLLGKFELSGIPPAPRGVPQIEVTFDVDSN 480
KSEIFSTYADNQPGVLIQVFEGERA+T DN+ LGKFEL+GIPPAPRGVPQIEVTFD+D+N
Sbjct 1107996 KSEIFSTYADNQPGVLIQVFEGERARTADNHQLGKFELTGIPPAPRGVPQIEVTFDIDAN 1107817
Query 481 GILNVSAVEKGTGKSNKITITNDKGRLSKEDIEKMVaeaekfkeedekeSQRIASKNQLE 540
GILNVSA +K TG+SNKITITNDKGRLS+EDIE+MV+EAEK+K++DE+ + RI +KN LE
Sbjct 1107816 GILNVSASDKTTGRSNKITITNDKGRLSQEDIERMVSEAEKYKKQDEEATARIHAKNGLE 1107637
Query 541 SIAYSLKNTISE--AGDKLEQADKDTVTKKAEETISWLDSNTTASKEEFDDKLKELQDIA 598
S AY+L+NT+++ K+++ADK+T+ K ETISWLD N ASKEEF+ K KEL+ A
Sbjct 1107636 SYAYNLRNTLNDDNLKGKIDEADKETLEKAITETISWLD*NLEASKEEFESKQKELEGTA 1107457
Query 599 NPIMSKLYQ 607
NPIM+KLYQ
Sbjct 1107456 NPIMTKLYQ 1107430
| Contig | Score (bits) | E-value | Identities | Positives | Gaps |
|---|---|---|---|---|---|
| 999 | 540 | 8e-171 | 82% | 91% | 1 |
Query 2 SKAVGIDLGTTYSCVAHFANDRVDIIANDQGNRTTPSFVAFTDTERLIGDAAKNQAAMNP 61
SKA+GIDLGTTYSCV + N++V+IIANDQGNRTTPS+VAFTD+ERL+GDAAKNQ +NP
Sbjct 945 SKAIGIDLGTTYSCVGVW*NEKVEIIANDQGNRTTPSYVAFTDSERLLGDAAKNQVGLNP 766
Query 62 SNTVFDAKRLIGRNFNDPEVQADMKHFPFKLIDVDGKPQIQVEFKGETKNFTPEQISSMV 121
NTVFDAKRLIGR F D EV +DMKH+PFK+ID GKP IQVE+ GETK FTPE++S+MV
Sbjct 765 YNTVFDAKRLIGRKFADAEV*SDMKHWPFKVIDKAGKPFIQVEYLGETKTFTPEEVSAMV 586
Query 122 LGKMKETAESYLGAKVNDAVVTVPAYFNDSQRQATKDAGTIAGLNVLRIINEPTAAAIAY 181
L KMKETAE++LGAKV +AVVTVPAYFNDSQRQATKDAG+IAGLNV+RIINEPTAAAIAY
Sbjct 585 LTKMKETAEAFLGAKVTNAVVTVPAYFNDSQRQATKDAGSIAGLNVMRIINEPTAAAIAY 406
Query 182 GLDKKGK-EEHVLIFDLGGGTFDVSLLSIEDGIFEVKATAGDTHLGGEDFDNRLVNHFIQ 240
GLDKK K E++VLIFDLGGGTFDVSLL+IE+GIFEVKATAGDTHLGGEDFDNRLV HF Q
Sbjct 405 GLDKKTKGEKNVLIFDLGGGTFDVSLLTIEEGIFEVKATAGDTHLGGEDFDNRLVTHFAQ 226
Query 241 EFKRKNKKDLSTNQRALRRLRTACERAKRTLSSSAQTSVEIDSLFEGIDFYTSITRARFE 300
EFKRK+KKDLS N R+LRRLRTACERAKRTLSS+ Q S+EIDSLFEG+DFYTSITRARFE
Sbjct 225 EFKRKHKKDLSGNARSLRRLRTACERAKRTLSSATQASIEIDSLFEGVDFYTSITRARFE 46
Query 301 ELCADLFRSTLDPVE 315
ELC DLFR TLDPVE
Sbjct 45 ELCGDLFRGTLDPVE 1
HSP71_YEAST - белок теплового шока, встречается у всех живых организмов. Он облегчает транспорт белков через мембраны митохондрий и эндоплазматического ретикулума, участуют в фолдинге белков; массовая транскрипция этого белка активируется при наличии факторов стресса (повышение температуры, отравление организма токсинами, УФ облучение и т д). HSP71_YEAST экспрессируется в организме Saccharomyces cerevisiae, царство Fungi, что оправдывает наличие гомологичного гена в геноме A. protococcarum, родственному грибам протисту. Белок достаточно консервативен: он принадлежит семейству Hsp70 белков, информация о единственнном домене этого белка лежит в базе данных консервативных доменов Pfam.
PRPC_EMENI - митохондриальная 2-метилцитрат синтетаза, катализирующая реакцию конденсации ацетил-СоА и оксалоацетата с образованием цитрата в рамках цикла Кребса. A. protococcarum - эндопаразит водорослей, то есть анаэроб, и цикл Кребса не входит в его катаболитические процессы, соответственно, цитратсинтетаза не экспрессируется. Действительно, в геноме A. protococcarum не нашлось генов, гомологочных гену PRPC_EMENI: для всех четырех находок процент идентичности меньше 56.
Одним из белков, выданных поиском blastx по таксону Fungi оказалась глюкозо-6-фосфат дегидрогеназа, закодированная участком 1071-го контига приблизительно с 12800 по 14300 нуклеотиды. Выравнивания достаточно хорошие, 50-60% идентичности; ниже приведена одна из находок:
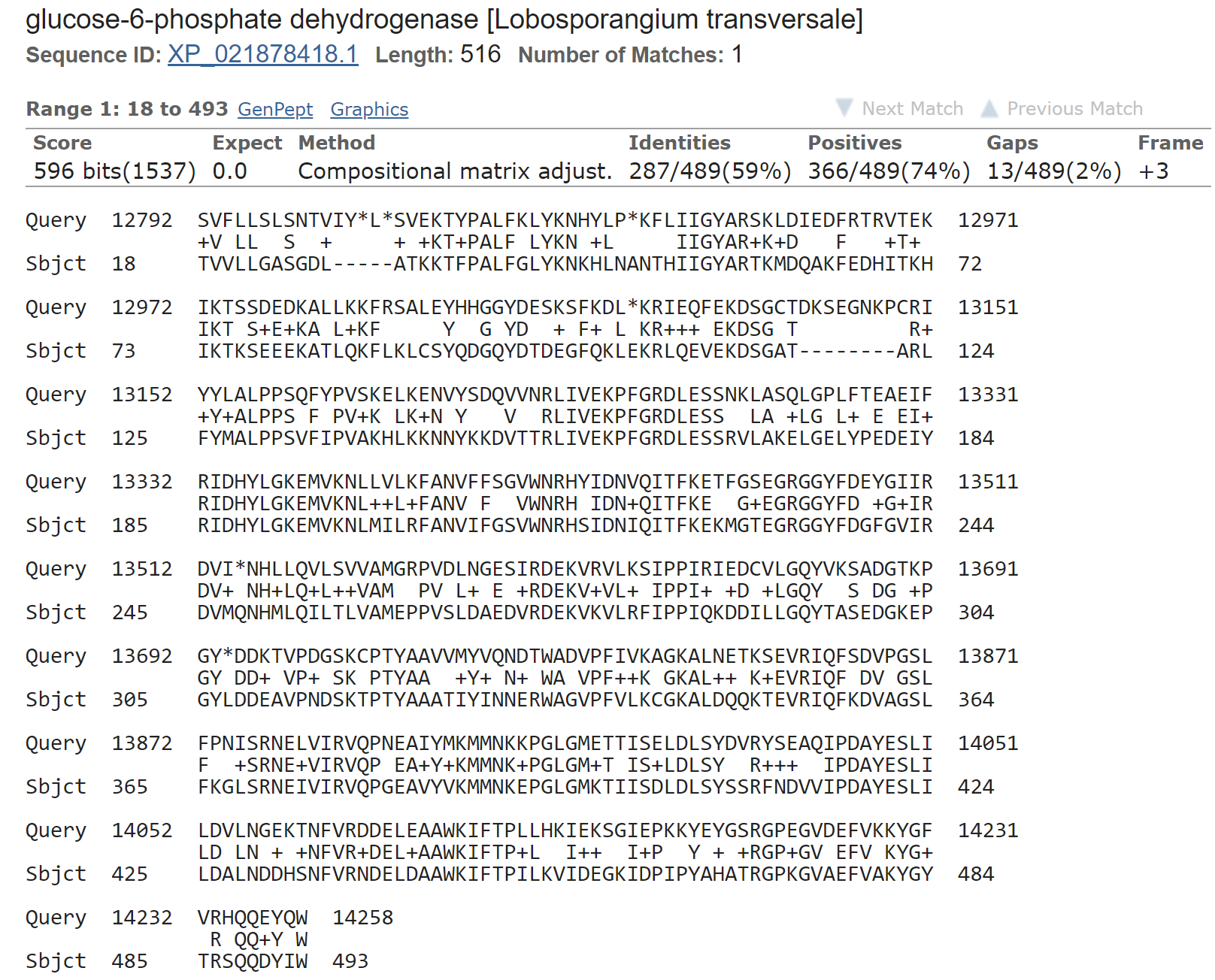
Глюкозо-6-фосфатдегидрогеназа участвует в первой реакции окислительного этапа пентозофосфатного гликолиза. Основная реакция фермента:
| D-глюкозо-6-фосфат + NADP+ → D-глюконо-1,5-лактон-6-фосфат + NADPH |
Лактон используется в следующих реакциях пентозофосфатного гликолиза, а восстановленный НАДФН в других метаболических процессах клетки.
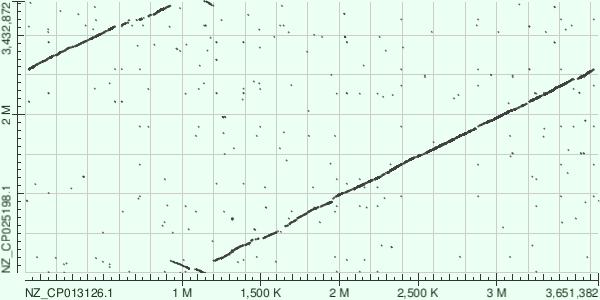
| Acidipropionibacterium acidipropionici strain CGMCC 1.2230 chromosome, complete genome NZ_CP013126.1 Acidipropionibacterium virtanenii strain JS278 chromosome, complete genome NZ_CP025198.1 |
Видно, что на участке 900 000 - 1 200 000 (границы очень приблизительны) произошла инверсия. Изображение на карте сдвинуто (участок 0 - 1 200 000), потому что прочтения последовательностей кольцевых ДНК NZ_CP013126.1 и NZ_CP025198.1 начинаются с разных мест. В целом, за исключением описанной инверсии, геномы схожи.
© Агаева Зара, 2018