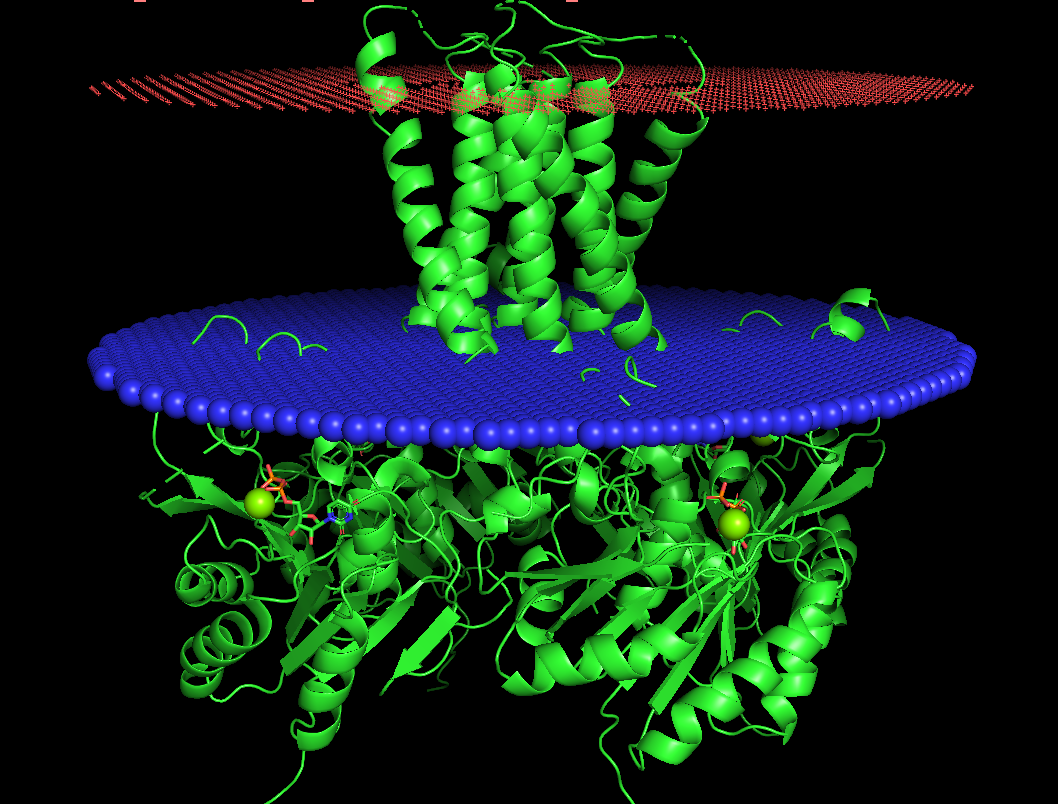
Таблица 1. Некоторые хараткеристики трансмембранной области тетрамерного белка Polyisoprenyl-phosphate glycosyltransferase GtrB [Synechocystis sp.] (PDB ID: 5EKE).
| Толщина гидрофобной части белка в мембране | Координаты трансмембранной области для α-спиралей | Среднее количество остатков в одной трансмембранной α-спирали | Расположение белка |
|---|---|---|---|
| 31.4 ± 1.2 Å | 1: 226-250, 2: 265-287 | 24 | внутренняя мембрана |

Рис. 1. Визуализация белка Polyisoprenyl-phosphate glycosyltransferase GtrB, полученная с помощью молекуялрного визуализатора PyMOL
Таблица 2. Некоторые хараткеристики трансмембранной области белка Phenol degradation pathway involved protein [Pseudomonas putida] (PDB ID: 4RL8).
| Толщина гидрофобной части белка в мембране | Координаты трансмембранной области для β-тяжей | Среднее количество остатков в одном трансмембранном β-тяже | Расположение белка |
|---|---|---|---|
| 23.4 ± 1.1 Å | 1: 16-24, 2: 47-54, 3: 64-73, 4: 90-100, 5: 109-118, 6: 136-146, 7: 154-162, 8: 182-191, 9: 198-204, 10: 225-232, 11: 240-245, 12: 258-266 | 9 | внешняя мембрана |
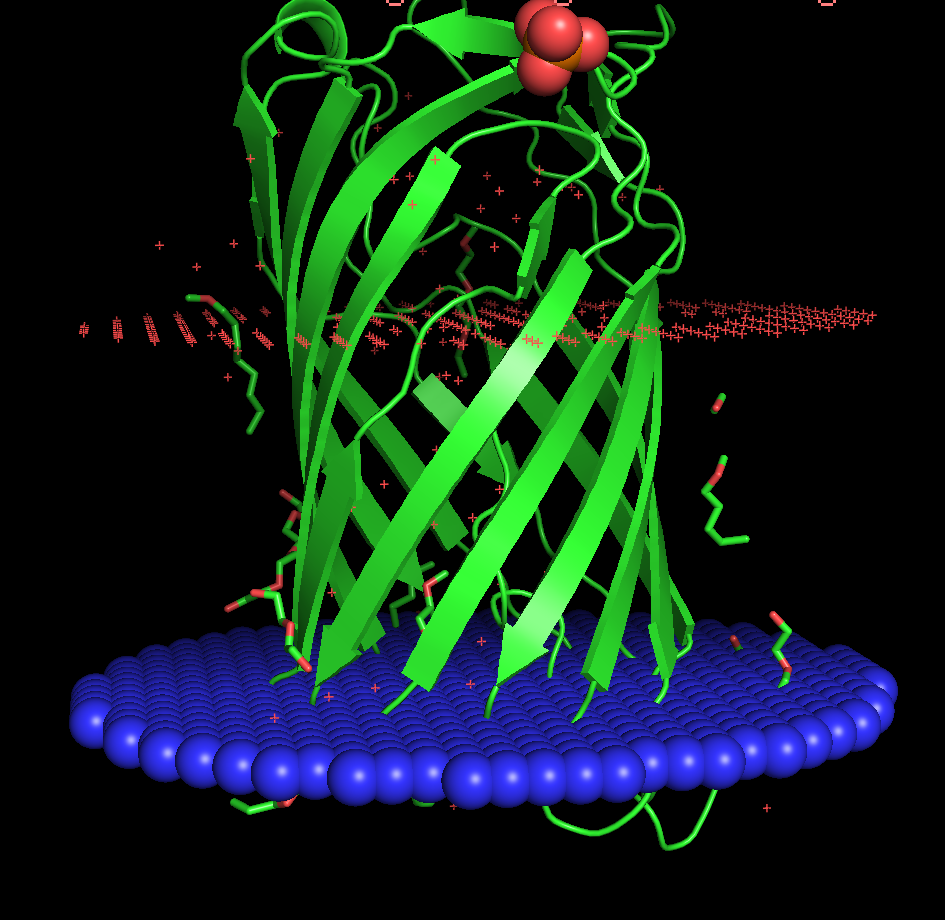
Рис. 1. Визуализация белка Phenol degradation pathway involved protein, полученная с помощью молекуялрного визуализатора PyMOL
# 5EKE:A|PDBID|CHAIN|SEQUENCE Length: 341 # 5EKE:A|PDBID|CHAIN|SEQUENCE Number of predicted TMHs: 2 # 5EKE:A|PDBID|CHAIN|SEQUENCE Exp number of AAs in TMHs: 44.85726 # 5EKE:A|PDBID|CHAIN|SEQUENCE Exp number, first 60 AAs: 0.04628 # 5EKE:A|PDBID|CHAIN|SEQUENCE Total prob of N-in: 0.17784 5EKE:A|PDBID|CHAIN|SEQUENCE TMHMM2.0 outside 1 252 5EKE:A|PDBID|CHAIN|SEQUENCE TMHMM2.0 TMhelix 253 272 5EKE:A|PDBID|CHAIN|SEQUENCE TMHMM2.0 inside 273 284 5EKE:A|PDBID|CHAIN|SEQUENCE TMHMM2.0 TMhelix 285 307 5EKE:A|PDBID|CHAIN|SEQUENCE TMHMM2.0 outside 308 341
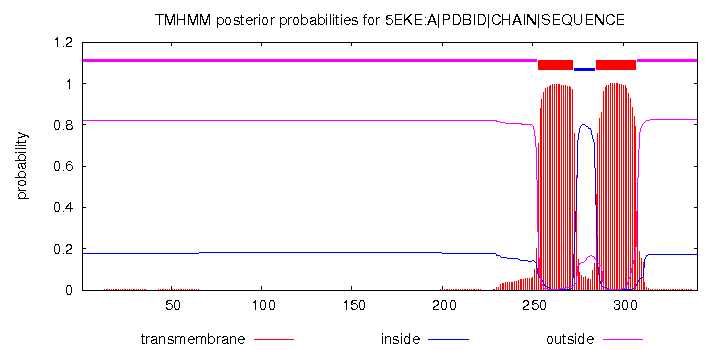
Рис. 3. Графический выход сервиса TMHMM: предсказания трансмембранных участков последовательности цепи А белка Polyisoprenyl-phosphate glycosyltransferase GtrB. По оси Ox отчисляются аминокислотные остатки (номера, отчитывающиеся от N-конца к C-концу), по Oy - вероятность этих остатков оказаться в каком-либо положении отностительно мембраны. Вероятность остатков оказаться в трансмембранной части белка отмечена сплошными красными областями на графике, розовый трек соответствует вероятности остатков оказаться с наружней стороны от мембраны, синий - с внуренней. На линейной схеме в верхней части рисунка приведено предсказанное программой расположение фрагментов цепи белка относительно мембраны: красные блоки соответствуют трансмембранным участкам, синие - находящимся в цитоплазме, розовые - находящимся снаружи от мембраны.
Prediction of 5EKE:A|PDBID|CHAIN|SEQUENCE ID 5EKE:A|PDBID|CHAIN|SEQUENCE FT TOPO_DOM 1 252 CYTOPLASMIC. FT TRANSMEM 253 274 FT TOPO_DOM 275 285 NON CYTOPLASMIC. FT TRANSMEM 286 311 FT TOPO_DOM 312 341 CYTOPLASMIC.
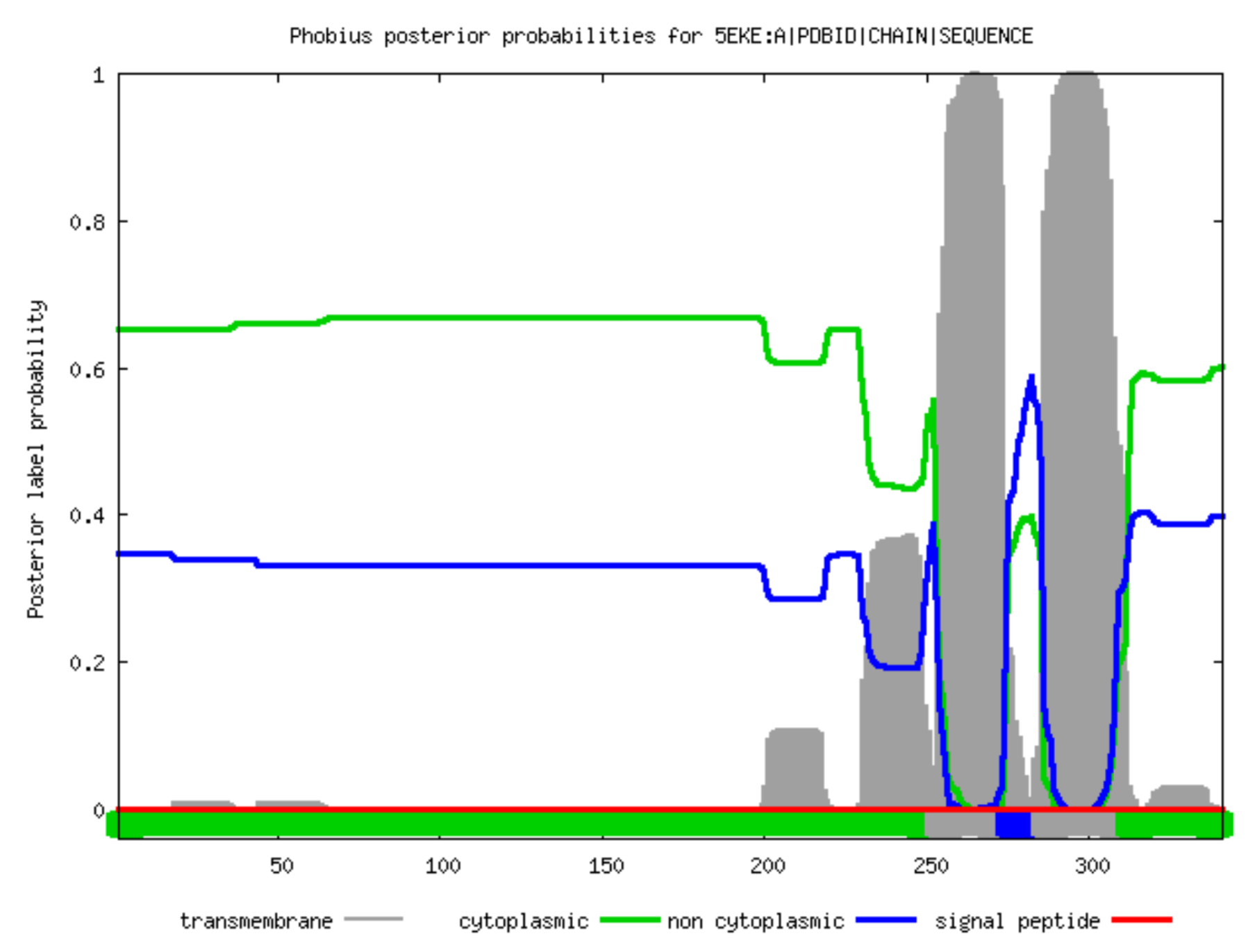
Рис. 4. Графический выход сервиса Phobius: предсказания трансмембранных участков последовательности цепи А белка Polyisoprenyl-phosphate glycosyltransferase GtrB. По оси Ox отчисляются аминокислотные остатки (номера, отчитывающиеся от N-конца к C-концу), по Oy - вероятность этих остатков оказаться в каком-либо положении отностительно мембраны. Вероятность остатков оказаться в трансмембранной части белка отмечена сплошными серыми областями на графике, зеленый трек соответствует вероятности остатков оказаться цитоплазматическими, синий - нецитоплазаматическими, красный - соответствует сигнальному пептиду. На линейной схеме в нижней части графика приведено предсказанное программой расположение фрагментов цепи белка относительно мембраны; блоки имеют те же цветовые обозначения, что и на графике.
Я привела выводы программ только для одной из цепей; для остальных картина аналогичная. Судя по текстовым выводам и линейным схемам программ TMHMM и Phobius, обе предсказали 2 трансмембранные спирали, как и описано в БД OPM, причем координаты каждой спирали, посчитанные с помощью TMHMM, близки к координатам, подсчитанные с помощью Phobius. Но если сравнивать выдачу этими сервисами координат спиралей с координатами из БД ОРМ, можно увидеть что они отличаются на 25-30 позиций. Также, по неизвестной мне причине, участки белка, которые в БД ОРМ отмечены как цитоплазматические, в выдаче TMHMM показаны как нецитоплазматические и наоборот, в то время как Phobius расположение участков цепи относительно мембраны предсказал правильно. Кроме того, Phobius присвоил 10% и 50% вероятности оказаться трансмембранными двум фрагментам цепи соответственно, находящимся перед первой трансмембранной спиралью, в то время как TMHMM присвоила им значительно гораздо меньший процент вероятности оказаться трансмембранными; то есть алгоритмы Phobius более чувствительны по сравнению с TMHMM.
Непосредственно сам Polyisoprenyl-phosphate glycosyltransferase GtrB в БД TCDB отсутствовал, поэтому я взяла белок Uncharacterized glycosyltransferase sll0501 [Synechocystis sp.], который был выдан мне в этой БД при поиске по PDB ID моего белка. TCID этого белка - 4.D.1.1.14. Аналогичная ситуация обстоит с Phenol degradation pathway involved protein, взяла Protein involved in meta-pathway of phenol degradation-like protein [Pseudomonas putida]. Его TCID - 9.B.153.4.3.
Таблица 3. Расшифровка TCID белков Uncharacterized glycosyltransferase sll0501 и Protein involved in meta-pathway of phenol degradation-like protein. В качестве шаблона TCID я использовала набор латинских букв - V.W.X.Y.Z.
| Позиция в TCID (из V.W.X.Y.Z) | 4.D.1.1.14 | 9.B.153.4.3 |
|---|---|---|
| V: класс транспортных белков | Белок транслокатор | Транспортная функция белка недостаточно хорошо описана |
| W: подкласс транспортных белков | Полисахарид-синтаза/экспортерный белок | Предположительно транспортный белок |
| X: семейство транспортных белков | Предположительно семейство гликозил-направленных полимераз | Предположительно семейство бета-бочковых поринов/альфа-амилаз или белков, участвующих в МетА-пути разложении фенола |
| Y: подсемейство транспортных белков | Подсемейство 1 (в БД TCDB точное название не указано) | Подсемейство 4 (в БД TCDB точное название не указано) |
| Z: субстрат или множество субстратов, транспортируемое данным белком | Конкретный переносимый субстрат в БД TCDB не был указан, известно, что данный белок обеспечивает гликозилирование полиизопренил фосфата. | Предположительно данный белок переносит через мембрану ионы и небольшие гидрофобные молекулы. |
© Агаева Зара, 2019